从宏基因组量化细菌生长动态
Introduciton
了解细菌在各种环境中的生长动态对于人类健康和环境监测等广泛领域至关重要。传统研究细菌生长的方法往往依赖于培养技术,这不仅耗时,而且对易培养的物种有偏向。然而,随着宏基因组测序技术的兴起,我们现在可以直接从环境样本中探讨整个微生物群落的生长动态。
宏基因组学通过测序样本中所有微生物的集体基因组,提供了微生物群落的全面快照。这种方法使研究人员能够绕过培养步骤,捕捉细菌在自然栖息地中的多样性和动态。通过分析宏基因组数据,我们可以推断细菌的生长速率、种群变化和群落相互作用。
本文主要介绍从宏基因组数据推断细菌生长速率的方法和简要应用。
Methods
PTR
Science 2015: https://www.science.org/doi/10.1126/science.aac4812
宏基因组测序增加了我们对微生物组在健康和疾病中的作用的理解,但它只提供了高度动态的生态系统的快照。在这里,作者表明不同微生物基因组的宏基因组测序读数覆盖模式包含单个波谷和单个波峰,后者与细菌复制起点一致。此外,峰和谷之间的测序覆盖率提供了物种生长速率的定量测量。

原理:是否可以通过检查跨细菌基因组的测序读数覆盖模式来从单个宏基因组样本中探测微生物群的生长动态。除了少数例子 之外,大多数细菌都具有单个圆形染色体,它从单个固定起点向单个终点双向复制。因此,在DNA复制过程中,已经被复制叉通过的区域将有两个拷贝,而尚未复制的区域将有一个拷贝。
总而言之,DNA 区域的拷贝数越接近复制起点,该区域的拷贝数就会越高;反之,该区域越接近复制终点,该区域的拷贝数就会越低。因此,复制起点附近的DNA拷贝数与复制终点附近的DNA拷贝数之间的比率,称之为峰谷比(Peak-to-Trough Ratio,PTR),应该反映细菌群体的生长速度。在较高的生长速率下,更大比例的细胞进行 DNA 复制,并且每个细胞中存在更活跃的复制叉。这导致近Origin DNA 和近 Terminus DNA 之间的比率高于 1:1,从而提供群落增长率的定量读数。
该文章在体外和体内、不同生长条件下以及复杂的细菌群落中证明了这一点。对于几种细菌物种,峰谷覆盖率(而非相对丰度)与炎症性肠病和 II 型糖尿病的表现相关。
文章提供的pipeline:http://genie.weizmann.ac.il/software/bac_growth.html ,该网页已经打不开了😂。
iRep
Nature Biotechnology 2016:https://www.nature.com/articles/nbt.3704
独立于培养物的微生物组研究增加了对微生物群落的复杂性和代谢潜力的理解。然而,为了了解单个微生物组成员对群落功能的贡献,确定哪些细菌正在积极复制非常重要。
作者开发了一种算法 iRep,它使用草稿质量的基因组序列和单时间点宏基因组测序来推断微生物群体的复制率。该算法根据从单个复制起点进行双向基因组复制产生的测序覆盖趋势来计算复制指数 (index of replication, iRep)。
作者应用这种方法来证明人类婴儿服用抗生素后微生物复制率增加。文章还表明,未培养的、与地下水相关的候选门辐射细菌很少在经历地球化学重大变化的地下群落中快速复制。该方法可以应用于任何基因组解析的微生物组研究,以跟踪生物体对不同条件的反应,识别活跃增长的种群并测量复制率以用于建模研究。

软件:https://github.com/christophertbrown/iRep,亲测可用
iRep 是一种通过单时间点宏基因组测序和草图质量基因组确定细菌复制率的方法。
-
安装:
pip install iRep注意:iRep 需要 Python 3。依赖项有lmfit,numpy,scipy,pandas,seaborn,matplotlib,cPickle。
-
脚本:
iRep: 使用草稿质量的基因组序列测量复制速率iRep_filter.py: 合并和/或过滤 iRep 输出bPTR: 使用完整的基因组序列测量复制速率(修改自 Korem 等人,Science 2015)gc_skew.py: 计算完整基因组序列的 GC 偏斜
-
示例用法:
-
iRep:
iRep -f sample_data/l_gasseri.fna -s sample_data/l_gasseri*sam -o test.iRep注意:为节省时间,可以同时分析多个基因组:
iRep -f genome1.fna genome2.fna -s sample_data/l_gasseri*sam -o test.iRep -
bPTR:
bPTR -f sample_data/l_gasseri.fna -s sample_data/l_gasseri*sam -o test.bPTR.tsv -plot test.bPTR.pdf -m coverage -
GC 偏斜:
gc_skew.py -f sample_data/l_gasseri.fna
-
-
使用说明:
运行 iRep 和 bPTR 需要每个目标微生物的基因组序列。每个程序都需要单独的 FASTA 文件作为输入。bPTR 需要完整(封闭)的基因组序列,而 iRep 需要高质量的草稿基因组(≥75% 完整性,≤175 个片段/Mbp 序列,≤2% 污染)。iRep 只使用长度≥5 Kbp 的 scaffold,因此基因组完整性估算需要基于通过此阈值的 scaffold。两种方法在基因组从研究样本中组装或已知系统中存在具有高度相似基因组序列的微生物时最为准确。第二组输入是 SAM 格式的映射文件,这些文件应通过使用 Bowtie2 将 DNA 测序读取映射到组装的基因组上生成。提供每个样本生成的 SAM 文件路径。
仅通过所有基因组和映射质量要求的 iRep 值应被视为有效。默认阈值:min cov. = 5,min wins. = 0.98,min r² = 0.9,max fragments/Mbp = 175,GC 校正 min r² = 0.0。
请注意,高水平的菌株变异会影响结果。
iRep 和 bPTR.py 输出包括结果表格(.tsv 文件)和 PDF,显示用于计算复制速率的基因组覆盖测量。
包含许多未映射读取的大型 SAM 文件解析速度较慢,可以使用 shrinksam 快速过滤 SAM 文件中的未映射读取。

如果有 “ImportError: No module named ‘_tkinter’” 错误。可以通过修改 bash MPLBACKEND 变量修复:
export MPLBACKEND="agg"
重要提示:
iRep 和 bPTR 都需要有序的 SAM 文件,这些文件可以使用 Bowtie2 的 --reorder 标志生成。有序的 SAM 文件中,表示一组配对读取的两个读取一个接一个排列,这对于这些脚本基于映射质量过滤读取(见 -mm 选项)是必需的。如果 SAM 文件未排序,请确保重新运行 Bowtie 并带上 --reorder 标志,或使用 --sort 选项排序 SAM 文件;否则,脚本在计算覆盖率时会错误选择要包含的映射读取。--sort 选项使用 Unix 排序重新排列 SAM 文件。此选项仅在使用映射质量截止时适用;然而,使用质量截止是推荐的,因为它有助于防止偏离目标的读取映射。
DEMIC
Nature Methods 2018: https://www.nature.com/articles/s41592-018-0182-0
对没有完整基因组序列的物种的微生物生长动态进行准确定量在生物学上很重要,但在宏基因组学中在计算上具有挑战性。在这里,作者提出了微生物群落的动态估计器(DEMIC;https://sourceforge.net/projects/demic/),这是一种基于重叠群和覆盖值的多样本算法,用于推断重叠群与复制起点的相对距离,并得出准确比较样品之间的细菌生长率。作者使用多个合成和真实数据集展示了 DEMIC 对于各种样本大小和组装质量的稳健性能。
软件:https://sourceforge.net/projects/demic/,perl报错未能解决
安装,下载压缩包解压即可:
wget -c https://master.dl.sourceforge.net/project/demic/DEMIC_v1.0.2.zip?viasf=1 -O DEMIC_v1.0.2.zip
tar -jxvf DEMIC_v1.0.2.zip
cd DEMIC_v1.0.2# 下载测试数据
wget -c https://sourceforge.net/projects/demic/files/test_data.zip?viasf=1 -O test_data.zip
unzip test_data.zip
DEMIC是一种计算工具,用于比较宏基因组样本中细菌的生长速率,基于推测的从复制起点到contig相对距离及其覆盖度。
如有任何关于DEMIC的问题,可以发邮件给工具作者:gy.james@163.com
- 命令和参数
如何运行DEMIC:
perl DEMIC.pl -S /input/sam_dir/ -F /input/fa_dir/ -O /output/dir/
DEMIC的参数如下:
-S, --sam_dir:输入SAM文件目录(由bowtie2生成)(必需)-F, --fasta_dir:输入FASTA文件目录,每个文件对应一个物种(必需)-O, --out_dir:输出文件目录(默认:./)-W, --window_size:计算覆盖度的窗口大小(默认:5000)-D, --window_step:计算覆盖度的窗口步长(默认:100)-M, --mapq_cutoff:计算覆盖度时的映射质量阈值(默认:5)-L, --mapl_cutoff:计算覆盖度时的映射长度阈值(默认:50)-R, --max_mismatch_ratio:每个读取作为命中的最大错配率(默认:0.03)-G, --log:输出日志文件名(可选)-H, --help:显示帮助信息-T, --thread_num:设置并行运行的线程数(默认:1)-Q, --quiet:运行时保持安静-A, --output_all:运行后保留临时文件(需要更多磁盘空间)
- 使用DEMIC的准备工作
(1) 共组装
不同样本中的宏基因组读取共同组装。请使用现有算法如MegaHit和Ray-Meta。例如,使用MegaHit从三个样本的宏基因组读取生成contigs:
cat sample1.fastq sample2.fastq sample3.fastq > samples.fastq
megahit -m 100000000000 -r samples.fastq -o MEGAHIT_assembly
(2) 比对
将宏基因组读取比对到contigs。请使用BowTie2。SAM文件需要用samtools排序。例如,使用以下命令比对sample1的读取并输出到目录SAM/:
bowtie2-build MEGAHIT_assembly/final.contigs.fa MEGAHIT_assembly/final.contigs.fa
bowtie2 -q -x MEGAHIT_assembly/final.contigs.fa -U sample1.fastq -S SAM/sample1.sam
排序比对并输出到SAM_sorted/sample1_sort.sam:
samtools view -bS SAM/sample1.sam | samtools sort - SAM/sample1_sort
samtools view -h SAM/sample1_sort.bam > SAM_sorted/sample1_sort.sam
请为所有样本的.sam文件执行此操作。
(3) 分类
将contigs聚类到组。请使用现有算法如MaxBin和MetaBAT。例如,使用MaxBin将contig聚类输出到maxbin/:
run_MaxBin.pl -contig MEGAHIT_assembly/final.contigs.fa -out maxbin -reads_list ./reads.list
(./reads.list包含上述三个.fastq文件的路径)
以上步骤是基于组装的宏基因组数据分析中常用的步骤。
DEMIC需要目录maxbin/和SAM_sorted/。
- 运行DEMIC示例
(1) 确保已安装并行Perl 5.10.1或更高版本,R 3.3.1或更高版本,并使用Linux或Mac OS X操作系统。
(2) 在R中安装“lme4”和“FactoMineR”包:
install.packages("lme4")
install.packages("FactoMineR")
(3) 在终端进入DEMIC目录,并将测试数据移动到该目录:
cd /path/to/DEMIC_v1.0.2/
mv /path/to/test_data/ ./
测试数据包括来自PTRC研究的三个样本的测序数据比对,这些数据格式化为目录SAM_sorted/中的已排序.sam。目录maxbin/中包括两个contig聚类,分别对应C. rodentium和E. faecalis。
(4) 运行DEMIC命令:
perl DEMIC.pl -S test_data/SAM_sorted/ -F test_data/maxbin/ -O DEMIC/
!!暂时会报错,且没有具体报错原因
(5) DEMIC将在大约1分钟内完成工作,并在目录DEMIC/中生成输出文件。
在该目录中,可以查看文件all_PTR.txt的详细信息:
$ cat DEMIC/all_PTR.txtSample1 Sample2 Sample3
ContigCluster1 1.5549 2.3313 1.8395
ContigCluster2 2.2430 1.7119 2.2856
输出文件易于理解。例如,C. rodentium(ContigCluster1)在Sample2中的生长速率最高。
- 常见问题
(1) 如何在DEMIC中使用多线程?
DEMIC支持多线程。使用多个CPU或内核时,请使用参数-T指定线程数。虽然优化了DEMIC的RAM使用,但使用多线程仍会略微增加RAM使用。建议选择不超过SAM文件数量和contig聚类数量的线程数。
(2) 如何设计实验和分析流程以更好地使用DEMIC?
虽然DEMIC适用于各种条件,但当实验和分析流程设计得更好时,它的性能可能更佳。
- 样本数量:DEMIC基于多个样本,对于包含六个或更多样本的物种,其准确性更高。建议以合理和可比的深度测序超过六个样本。不同样本可以来自对照组、重复实验、不同个体或条件。
- 组装器:组装方法对binning方法和DEMIC的性能很重要。能够为不同深度和物种生成相对较长contig且错误较少的组装器更为优选。在测试中使用了MEGAHIT,表现良好。
- 分类器:尽管DEMIC可以通过去除潜在污染物纯化contig聚类,但其仍受binning质量(如完整性)的影响。对于低或中等复杂度的微生物群落,建议使用MaxBin。
CoPTR
Genome Research 2022: https://genome.cshlp.org/content/32/3/558.full
细菌基因组的测序覆盖模式(通过峰谷比(PTR)总结)已被证明可以准确反映微生物的生长速率,揭示微生物动力学和宿主-微生物相互作用的新方面。在这里,作者介绍Compute PTR (CoPTR):一种用于从完整的参考基因组和组装中计算PTR的工具。
使用简单和复杂群落中的生长实验的模拟和数据,作者表明 CoPTR 比当前最先进的技术更准确,同时还提供了更多的总体 PTR 估计。作者进一步发展了一种理论,正式化了 PTR 的生物学解释。作者使用 2935 个物种的参考数据库,将 CoPTR 应用于来自 106 名炎症性肠病患者的 1304 个宏基因组样本的病例对照研究。作者表明,增长率是个性化的,仅与相对丰度松散相关,并且与疾病状态相关。最后,作者展示了如何将 PTR 与相对丰度和代谢组学相结合来研究它们对微生物组的影响。

软件:https://github.com/tyjo/coptr
CoPTR 是一种工具,用于根据完整参考基因组和草图组件的宏基因组测序数据计算峰谷比。 CoPTR 将来自多个宏基因组样本和参考数据库的 fastq 文件作为输入。它输出样本中每个物种的 log2 PTR。
快速使用:
# 项目下载
git clone https://github.com/tyjo/coptr
cd coptr# 测试数据
wget https://dl.dropboxusercontent.com/s/wrsxjimr6l96lcq/example-data.tar.gz
tar -xzvf example-data.tar.gz# 设置环境:
conda env create -f coptr.yml
conda activate coptr# 软件安装
pip install .# 索引参考数据库:
coptr index example-data/ref-db example-data/ref-db/example-index# 地图显示与参考相反:
coptr map example-data/ref-db/example-index example-data/fastq example-data/bam# 提取读取位置:
coptr extract example-data/bam example-data/coverage-maps#估计ptrs:
#请注意,最小读取标志是可选的。我们建议使用默认设置(5000次读取)。
coptr estimate example-data/coverage-maps out.csv --min-reads 2500# Output
cat out.csvlog2(PTR):genome_id/sample_id,ERR969281,ERR969282,ERR969283,ERR969285,ERR969286,ERR969428,ERR969429,ERR969430
l-gasseri-ref,,,,,,1.1840987863325785,1.1945539660363145,1.2879271469720541
e-coli-mag,1.2078623467253466,1.0375575947553943,0.9433005522894075,0.759132363901812,0.7846476652840171,,,
References
- Tal Korem et al., Growth dynamics of gut microbiota in health and disease inferred from single metagenomic samples.Science349,1101-1106 (2015). DOI:10.1126/science.aac4812
- Brown, C., Olm, M., Thomas, B. et al. Measurement of bacterial replication rates in microbial communities. Nat Biotechnol 34, 1256–1263 (2016). https://doi.org/10.1038/nbt.3704
- Gao, Y., Li, H. Quantifying and comparing bacterial growth dynamics in multiple metagenomic samples. Nat Methods 15, 1041–1044 (2018). https://doi.org/10.1038/s41592-018-0182-0
- Tyler A. Joseph et al., Accurate and robust inference of microbial growth dynamics from metagenomic sequencing reveals personalized growth rates. Genome Res. 2022. 32: 558-568 (2022). doi:
10.1101/gr.275533.121

关注公众号 ‘bio llbug’,获取最新推送。
相关文章:

从宏基因组量化细菌生长动态
Introduciton 了解细菌在各种环境中的生长动态对于人类健康和环境监测等广泛领域至关重要。传统研究细菌生长的方法往往依赖于培养技术,这不仅耗时,而且对易培养的物种有偏向。然而,随着宏基因组测序技术的兴起,我们现在可以直接…...

Linux---git工具
目录 初步了解 基本原理 基本用法 安装git 拉取远端仓库 提交三板斧 1、添加到缓存区 2、提交到本地仓库 3、提交到远端 其他指令补充 多人协作管理 windows用户提交文件 Linux用户提交文件 初步了解 在Linux中,git是一个指令,可以帮助我们做…...

【JavaScript】函数的动态传参
Javacript(简称“JS”)是一种具有函数优先的轻量级,解释型或即时编译型的编程语言。虽然它是作为开发Web页面的脚本语言而出名,但是它也被用到了很多非浏览器环境中,JavaScript基于原型编程、多范式的动态脚本语言&…...

从0到1,AI我来了- (4)AI图片识别的理论知识-II
上篇文章,我们理解了我们程序的神经网络设计,这篇我们继续,把训练迭代过程分析一下,完成这两篇文章,下面问题,应该能回答了。 一张图片,如何被计算机读懂?pytorch 封装的网络&#…...

2024 Java 高分面试宝典 一站式搞定技术面
前言 每年9月和10月,被业界称为“金九银十”,这是人才市场一年中最活跃的时期。此时,企业为了来年的业务扩展,纷纷加大招聘力度,空缺岗位众多,招聘需求集中。同时,初秋的招聘活动也避开酷暑&am…...

MongoDB - 聚合操作符 $eq、$gte、$in、$sum、$avg
文章目录 1. $eq2. $gte3. $in4. $sum5. $avg 1. $eq $eq比较两个值并返回:true (当值相等时)|false(当值不相等时) { $eq: [ <expression1>, <expression2> ] }构造测试数据: db.inventory…...

C语言 | Leetcode C语言题解之第279题完全平方数
题目: 题解: // 判断是否为完全平方数 bool isPerfectSquare(int x) {int y sqrt(x);return y * y x; }// 判断是否能表示为 4^k*(8m7) bool checkAnswer4(int x) {while (x % 4 0) {x / 4;}return x % 8 7; }int numSquares(int n) {if (isPerfect…...

在appium中,如何通过匹配图片来进行断言?
在Appium中进行图片匹配断言,可以使用OpenCV来实现。以下是使用Appium和OpenCV进行图片匹配断言的示例代码。 首先,需要确保安装了必要的库: pip install opencv-python-headless pip install opencv-python pip install numpy然后…...

昇思25天学习打卡营第21天|CV-Shufflenet图像分类
打卡 目录 打卡 ShuffleNet 网络介绍 ShuffleNet 模型架构 Pointwise Group Convolution Channel Shuffle ShuffleNet模块 ShuffleNet 模块代码 构建ShuffleNet网络 模块代码 模型训练和评估 模型训练 模型评估 模型预测 ShuffleNet 网络介绍 ShuffleNetV1是旷视科…...

python 图片转文字、语音转文字、文字转语音保存音频并朗读
一、python图片转文字 1、引言 pytesseract是基于Python的OCR工具, 底层使用的是Google的Tesseract-OCR 引擎,支持识别图片中的文字,支持jpeg, png, gif, bmp, tiff等图片格式 2、环境配置 python3.6PIL库安装Google Tesseract OCR 3、安…...

SSRF (服务端请求伪造)
🎼个人主页:金灰 😎作者简介:一名简单的大一学生;易编橙终身成长社群的嘉宾.✨ 专注网络空间安全服务,期待与您的交流分享~ 感谢您的点赞、关注、评论、收藏、是对我最大的认可和支持!❤️ 🍊易编橙终身成长社群&#…...

SQL中的LEFT JOIN、RIGHT JOIN和INNER JOIN
在SQL中,JOIN操作是连接两个或多个数据库表,并根据两个表之间的共同列(通常是主键和外键)返回数据的重要方法。其中,LEFT JOIN(左连接)、RIGHT JOIN(右连接)和INNER JOIN…...

[网鼎杯 2020 朱雀组]Nmap(详细解读版)
这道题考察nmap的一些用法,以及escapeshellarg和escapeshellcmd两个函数的绕过,可以看这里PHP escapeshellarg()escapeshellcmd() 之殇 (seebug.org) 两种解题方法: 第一种通过nmap的-iL参数读取扫描一个文件到指定文件中第二种是利用nmap的参数写入we…...

【React】详解“最新”和“最热”切换与排序
文章目录 一、基本概念和初始化二、切换与排序功能的实现1. 函数定义和参数2. 设置活动 Tab3. 定义新列表变量4. 根据排序类型处理列表4.1 按时间降序排序4.2 按点赞数降序排序 5. 更新评论列表 三、渲染导航 Tab 和评论列表1. map 方法2. key 属性3. className 动态赋值4. onC…...

BUUCTF [MRCTF2020]Ezpop
这道题对于刚接触到pop链的我直接把我整懵了,一边看着魔术方法一边分析 魔术方法可以看这里PHP 魔术方法 - 简介 - PHP 魔术方法 - 简单教程,简单编程 (twle.cn) 代码解析 经过以上的分析我们可以理一下解题思路:接收参数反序列化之前先触发…...

RV1126 Linux 系统,接外设,时好时坏(一)应该从哪些方面排查问题
在 Linux 系统中接外设时,遇到“时好时坏”的问题,可能是由多种因素引起的。以下是一些排查问题的建议。 1. 硬件方面的排查 1.1 连接检查 物理连接: 确保外设与主板之间的连接良好,检查插头、插座及线缆是否牢固。引脚配置: 确认设备树中引脚的配置是否正确,尤其是引脚…...

Vue实现简单小案例
一、创建文件夹 二、引用vue.js <script src"../js/vue.js"></script> 三、准备一个容器 <div id"app"><h1>Hello,{{name}}</h1> </div> 四、创建实例 <script>new Vue({el:"#app", //el用于指…...

【MATLAB APP】建立独立桌面APP
背景:已有MATLAB APP的.mlapp文件,但客户提出需要可以直接使用的exe文件。 要求:点开即用,无需下载MATLAB。使用者无法修改APP的代码。 一、环境配置 APP创建者:安装MATLAB R2023a,配置Application Compile…...

Spring的优缺点?
Spring的优缺点 直接回答相关的Spring的特点: IOC AOP 事务 简化开发: 容易集成JDBCTemplateRestTemplate(接口远程调用)邮件发送相关异步消息请求支持 更加深入就讲源码了 优点: 方便解耦,简化开发…...

第一百八十三节 Java IO教程 - Java目录事件、Java异步I/O
Java IO教程 - Java目录事件 当文件系统中的对象被修改时,我们可以监听watch服务以获取警报。 java.nio.file包中的以下类和接口提供watch服务。 Watchable接口WatchService接口WatchKey接口WatchEvent接口WatchEvent.Kind接口StandardWatchEventKinds类 可监视对…...

2021-03-15 iview一些问题
1.iview 在使用tree组件时,发现没有set类的方法,只有get,那么要改变tree值,只能遍历treeData,递归修改treeData的checked,发现无法更改,原因在于check模式下,子元素的勾选状态跟父节…...
指令的指南)
在Ubuntu中设置开机自动运行(sudo)指令的指南
在Ubuntu系统中,有时需要在系统启动时自动执行某些命令,特别是需要 sudo权限的指令。为了实现这一功能,可以使用多种方法,包括编写Systemd服务、配置 rc.local文件或使用 cron任务计划。本文将详细介绍这些方法,并提供…...
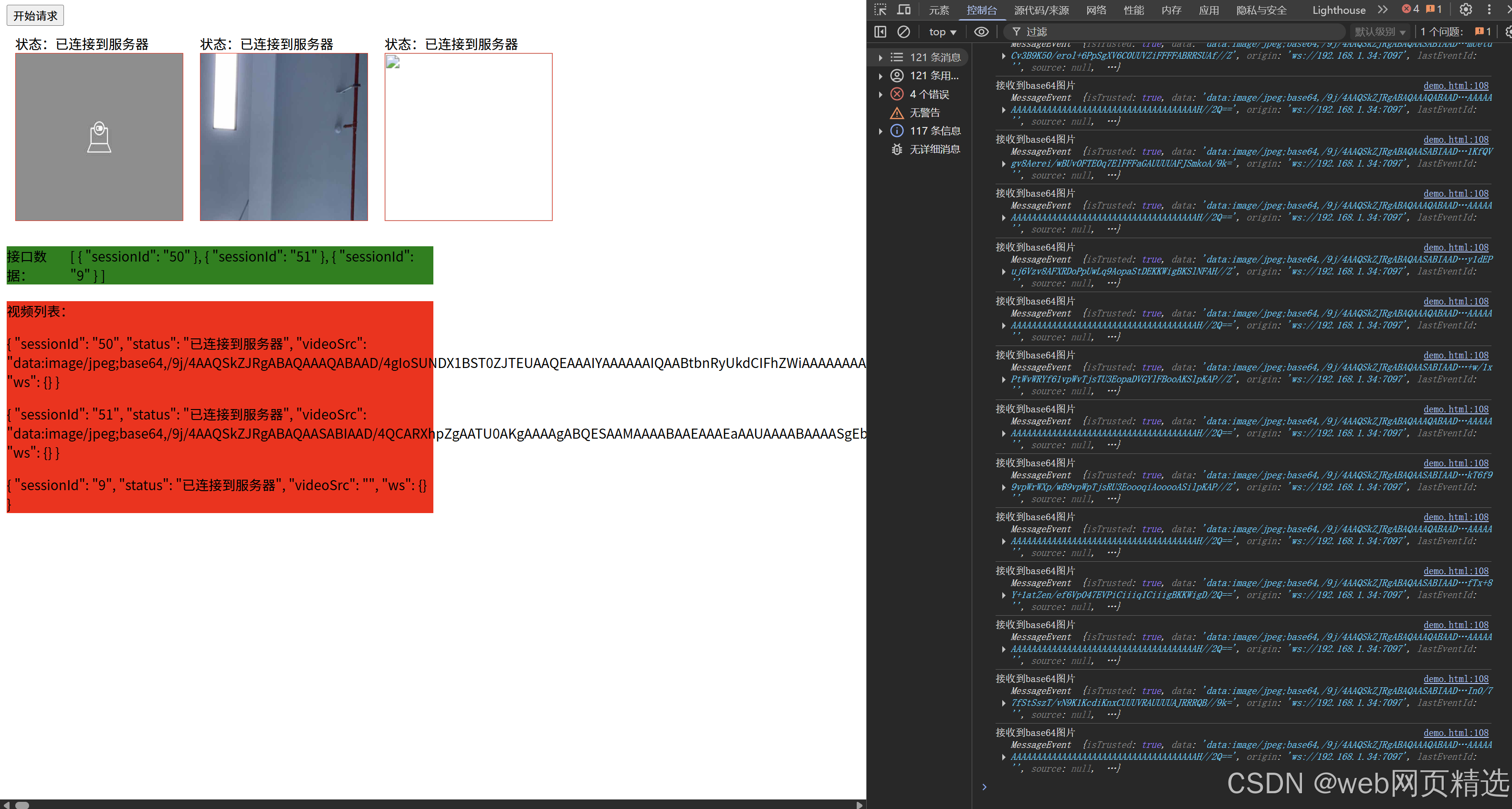
uniapp微信小程序视频实时流+pc端预览方案
方案类型技术实现是否免费优点缺点适用场景延迟范围开发复杂度WebSocket图片帧定时拍照Base64传输✅ 完全免费无需服务器 纯前端实现高延迟高流量 帧率极低个人demo测试 超低频监控500ms-2s⭐⭐RTMP推流TRTC/即构SDK推流❌ 付费方案 (部分有免费额度&#x…...

QT: `long long` 类型转换为 `QString` 2025.6.5
在 Qt 中,将 long long 类型转换为 QString 可以通过以下两种常用方法实现: 方法 1:使用 QString::number() 直接调用 QString 的静态方法 number(),将数值转换为字符串: long long value 1234567890123456789LL; …...
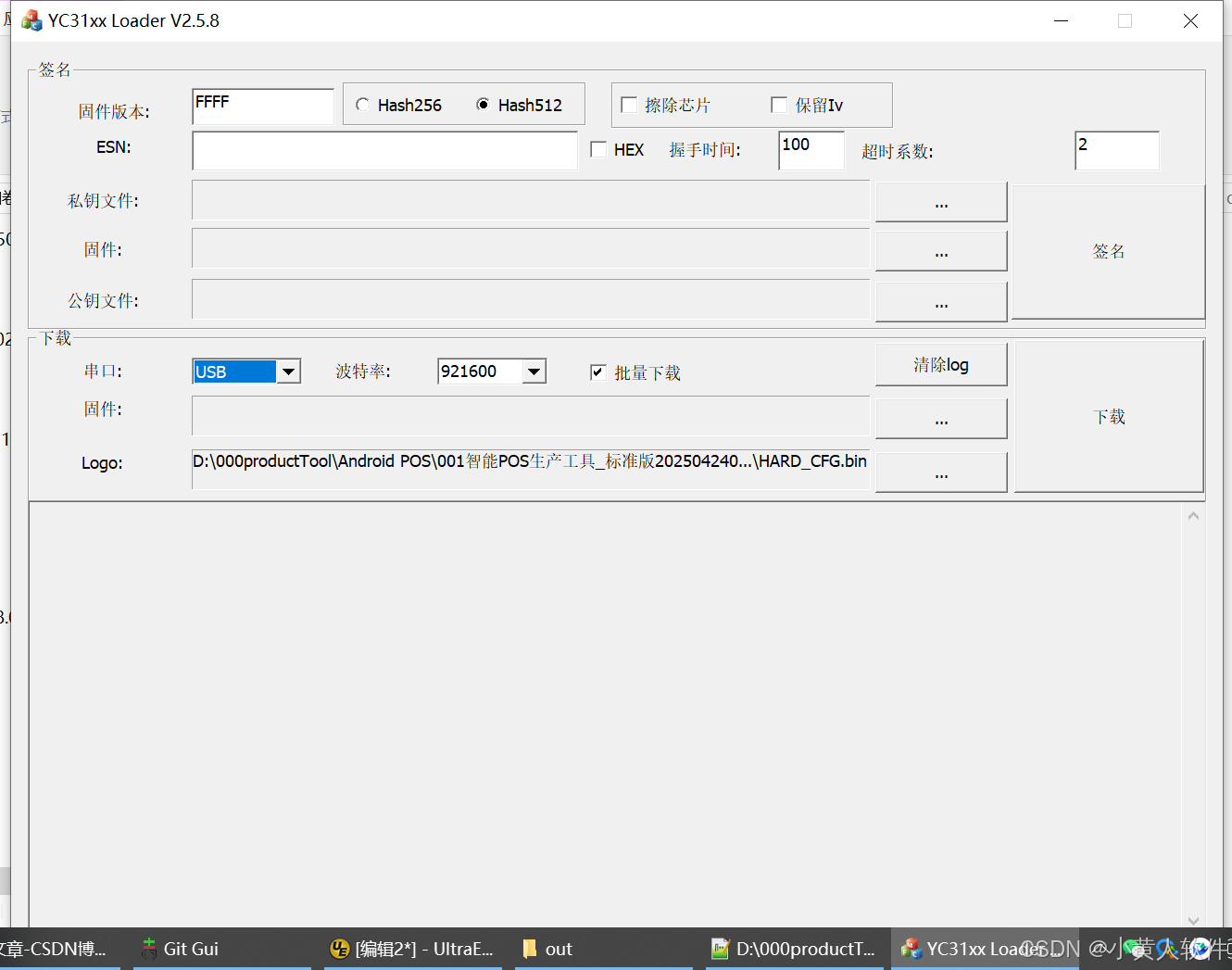
C++ Visual Studio 2017厂商给的源码没有.sln文件 易兆微芯片下载工具加开机动画下载。
1.先用Visual Studio 2017打开Yichip YC31xx loader.vcxproj,再用Visual Studio 2022打开。再保侟就有.sln文件了。 易兆微芯片下载工具加开机动画下载 ExtraDownloadFile1Info.\logo.bin|0|0|10D2000|0 MFC应用兼容CMD 在BOOL CYichipYC31xxloaderDlg::OnIni…...

使用Matplotlib创建炫酷的3D散点图:数据可视化的新维度
文章目录 基础实现代码代码解析进阶技巧1. 自定义点的大小和颜色2. 添加图例和样式美化3. 真实数据应用示例实用技巧与注意事项完整示例(带样式)应用场景在数据科学和可视化领域,三维图形能为我们提供更丰富的数据洞察。本文将手把手教你如何使用Python的Matplotlib库创建引…...
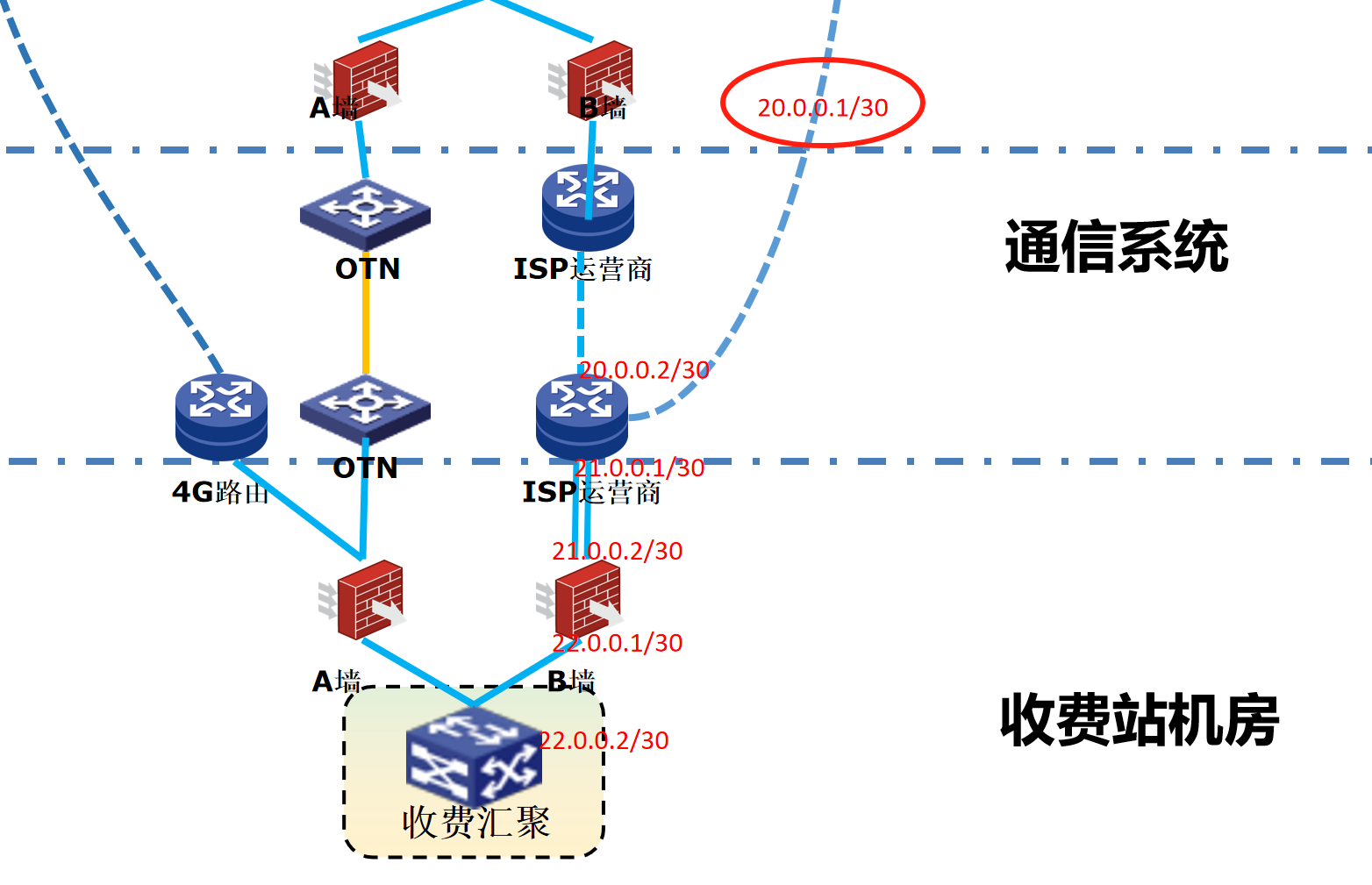
浪潮交换机配置track检测实现高速公路收费网络主备切换NQA
浪潮交换机track配置 项目背景高速网络拓扑网络情况分析通信线路收费网络路由 收费汇聚交换机相应配置收费汇聚track配置 项目背景 在实施省内一条高速公路时遇到的需求,本次涉及的主要是收费汇聚交换机的配置,浪潮网络设备在高速项目很少,通…...
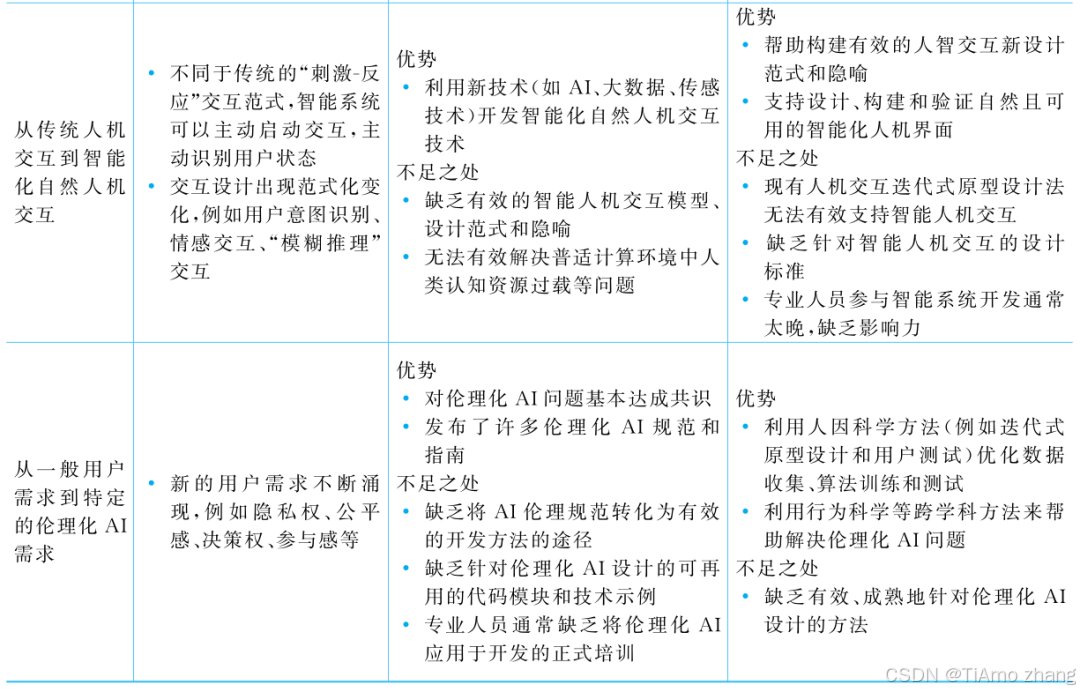
人机融合智能 | “人智交互”跨学科新领域
本文系统地提出基于“以人为中心AI(HCAI)”理念的人-人工智能交互(人智交互)这一跨学科新领域及框架,定义人智交互领域的理念、基本理论和关键问题、方法、开发流程和参与团队等,阐述提出人智交互新领域的意义。然后,提出人智交互研究的三种新范式取向以及它们的意义。最后,总结…...
混合(Blending))
C++.OpenGL (20/64)混合(Blending)
混合(Blending) 透明效果核心原理 #mermaid-svg-SWG0UzVfJms7Sm3e {font-family:"trebuchet ms",verdana,arial,sans-serif;font-size:16px;fill:#333;}#mermaid-svg-SWG0UzVfJms7Sm3e .error-icon{fill:#552222;}#mermaid-svg-SWG0UzVfJms7Sm3e .error-text{fill…...
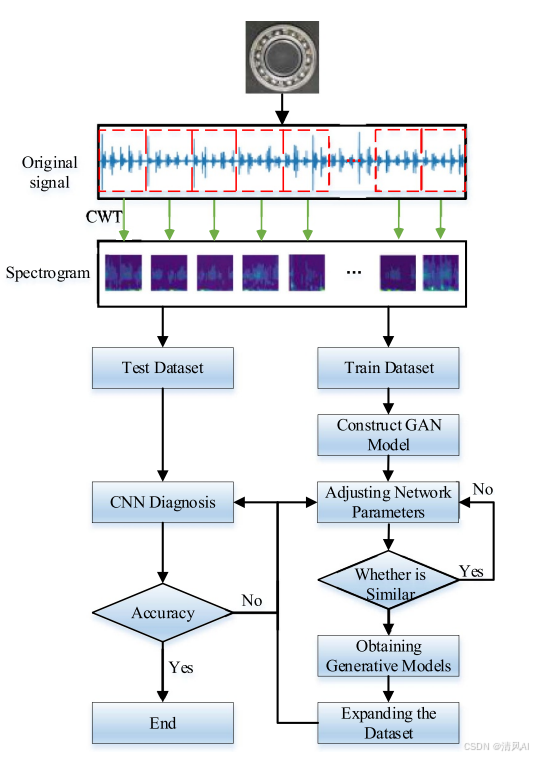
基于IDIG-GAN的小样本电机轴承故障诊断
目录 🔍 核心问题 一、IDIG-GAN模型原理 1. 整体架构 2. 核心创新点 (1) 梯度归一化(Gradient Normalization) (2) 判别器梯度间隙正则化(Discriminator Gradient Gap Regularization) (3) 自注意力机制(Self-Attention) 3. 完整损失函数 二…...
