DMNet复现(一)之数据准备篇:Density map guided object detection in aerial image
一、生成密度图
密度图标签生成
采用以下代码,生成训练集密度图gt:
import cv2
import glob
import h5py
import scipy
import pickle
import numpy as np
from PIL import Image
from itertools import islice
from tqdm import tqdm
from matplotlib import pyplot as plt
from sortedcontainers import SortedDict
from scipy.ndimage.filters import gaussian_filter
from scipy.spatial import KDTree
import argparse"""
Code for DMnet, density map ground truth generation
Author: Changlin Li
Code revised on : 7/15/2020Given dataset(train/val/test) generate ground truth for given dataset.
Default format for source data: The input images are in jpg format and raw annotations are in txt format
(Based on Visiondrone 2018/19/20 dataset)Sample code to run:python Generate_density_map_official.py . gaussian_kernels.pkl distances_dict.pkl --mode val
"""# point_class_pair = {}
# annotation_stats = {0: 17, 1: 14, 2: 20, 3: 32, 4: 35, 5: 45, 6: 29, 7: 30, 8: 46, 9: 18}
# min_sigma, max_sigma = min(annotation_stats.values()), max(annotation_stats.values())
# print(min_sigma, max_sigma)def get_img_paths(path_sets):"""Return all images from all pathes in 'path_sets'"""img_paths = []for path in path_sets:for img_path in glob.glob(os.path.join(path, '*.jpg')):img_paths.append(img_path)return img_pathsdef save_computed_density(density_map, out_path):"""Save density map to h5py format"""with h5py.File(out_path, 'w') as hf:hf['density'] = density_mapdef compute_sigma(gt_count, distance=None, min_sigma=1, method=1, fixed_sigma=15):"""Compute sigma for gaussian kernel with different methods :* method = 1 : sigma = (sum of distance to 3 nearest neighbors) / 10* method = 2 : sigma = distance to nearest neighbor* method = 3 : sigma = fixed value** if sigma lower than threshold 'min_sigma', then 'min_sigma' will be used** in case of one point on the image sigma = 'fixed_sigma'"""if gt_count > 1 and distance is not None:if method == 1:sigma = np.mean(distance[1:4]) * 0.1elif method == 2:sigma = distance[1]elif method == 3:sigma = fixed_sigmaelse:sigma = fixed_sigmaif sigma < min_sigma:sigma = min_sigmareturn sigmadef find_closest_key(sorted_dict, key):"""Find closest key in sorted_dict to 'key'"""keys = list(islice(sorted_dict.irange(minimum=key), 1))keys.extend(islice(sorted_dict.irange(maximum=key, reverse=True), 1))return min(keys, key=lambda k: abs(key - k))def gaussian_filter_density(non_zero_points, map_h, map_w, distances=None, kernels_dict=None, min_sigma=2, method=1,const_sigma=15):"""Fast gaussian filter implementation : using precomputed distances and kernels"""gt_count = non_zero_points.shape[0]density_map = np.zeros((map_h, map_w), dtype=np.float32)for i in range(gt_count):point_x, point_y, category = non_zero_points[i]sigma = compute_sigma(gt_count, distances[i], min_sigma=min_sigma, method=method, fixed_sigma=const_sigma)# closest_sigma = annotation_stats[category]closest_sigma = find_closest_key(kernels_dict, sigma)# print(i,closest_sigma)kernel = kernels_dict[closest_sigma]full_kernel_size = kernel.shape[0]kernel_size = full_kernel_size // 2min_img_x = max(0, point_x - kernel_size)min_img_y = max(0, point_y - kernel_size)max_img_x = min(point_x + kernel_size + 1, map_w - 1)max_img_y = min(point_y + kernel_size + 1, map_h - 1)assert max_img_x > min_img_xassert max_img_y > min_img_ykernel_x_min = kernel_size - point_x if point_x <= kernel_size else 0kernel_y_min = kernel_size - point_y if point_y <= kernel_size else 0kernel_x_max = kernel_x_min + max_img_x - min_img_xkernel_y_max = kernel_y_min + max_img_y - min_img_yassert kernel_x_max > kernel_x_minassert kernel_y_max > kernel_y_mindensity_map[min_img_y:max_img_y, min_img_x:max_img_x] += kernel[kernel_y_min:kernel_y_max,kernel_x_min:kernel_x_max]return density_mapdef get_gt_dots(ann_path, img_height, img_width, mode="train"):"""Load Matlab file with ground truth labels and save it to numpy array.** cliping is needed to prevent going out of the array"""txt_list = open(ann_path, 'r').readlines()gt = format_label(mode, txt_list)assert gt.shape[1] == 3gt[:, 0] = gt[:, 0].clip(0, img_width - 1)gt[:, 1] = gt[:, 1].clip(0, img_height - 1)return gtdef set_circles_on_img(image, bbox_list, circle_size=2):"""Set circles on images at centers of bboxes in bbox_list"""for bbox in bbox_list:cv2.circle(image, (bbox[0], bbox[1]), circle_size, (255, 0, 0), -1)return imagedef generate_gaussian_kernels(out_kernels_path='gaussian_kernels.pkl', round_decimals=3, sigma_threshold=4, sigma_min=0,sigma_max=20, num_sigmas=801):"""Computing gaussian filter kernel for sigmas in linspace(sigma_min, sigma_max, num_sigmas) and saving them to dict. """if os.path.exists(out_kernels_path):# If kernel has been pre-computed, then returnprint("Kernel already created!\nExiting...\n")returnkernels_dict = dict()sigma_space = np.linspace(sigma_min, sigma_max, num_sigmas)for sigma in tqdm(sigma_space):sigma = np.round(sigma, decimals=round_decimals)kernel_size = np.ceil(sigma * sigma_threshold).astype(np.int)img_shape = (kernel_size * 2 + 1, kernel_size * 2 + 1)img_center = (img_shape[0] // 2, img_shape[1] // 2)arr = np.zeros(img_shape)arr[img_center] = 1arr = scipy.ndimage.filters.gaussian_filter(arr, sigma, mode='constant')kernel = arr / arr.sum()kernels_dict[sigma] = kernelprint(f'Computed {len(sigma_space)} gaussian kernels. Saving them to {out_kernels_path}')with open(out_kernels_path, 'wb') as f:pickle.dump(kernels_dict, f)def compute_distances(out_dist_path='distances_dict.pkl', raw_label_dir='D:/BaiduNetdiskDownload/VisDrone', n_neighbors=4,leafsize=1024, data_limit=None, mode="train", img_affix=".jpg"):if os.path.exists(out_dist_path):# If distance has been computed, then directly load distance file.print("Distrance pre-computation already created!\nExiting...\n")returndistances_dict = dict()full_img_paths = glob.glob(f'{raw_label_dir}/VisDrone2019-DET-train/images/*' + img_affix) + \glob.glob(f'{raw_label_dir}/VisDrone2019-DET-val/images/*' + img_affix) + \glob.glob(f'{raw_label_dir}/VisDrone2019-DET-test-dev/images/*' + img_affix)if data_limit and data_limit < len(full_img_paths):full_img_paths = full_img_paths[:data_limit]for img_path in tqdm(full_img_paths):ann_path = img_path.replace(img_affix, '.txt')ann_path = ann_path.replace("images", "annotations")img = plt.imread(img_path)non_zero_points = get_gt_dots(ann_path, *img.shape[0:2], mode=mode)tree = KDTree(non_zero_points.copy(), leafsize=leafsize) # build kdtreedistances, _ = tree.query(non_zero_points, k=n_neighbors) # query kdtreedistances_dict[img_path] = distancesprint(f'Distances computed for {len(full_img_paths)}. Saving them to {out_dist_path}')with open(out_dist_path, 'wb') as f:pickle.dump(distances_dict, f)def format_label(mode, txt_list):format_data = []# required format: xmin, ymin, xmax, ymax, class_id, clockwise direction# Given format: <bbox_left>,<bbox_top>,<bbox_width>,<bbox_height>,class_idfor idx, i in enumerate(txt_list):coord_raw = [int(x) for x in i.replace("\n", "").split(',') if len(x) != 0]coord = coord_raw[:6]# print(coord)if len(coord) != 6:# 4 coord + 1 classprint("Failed to parse annotation!")exit()# if coord[-1] not in class_list and coord[-1]>len(class_list):# print('warning found a new label :', coord[-1])# exit()if coord[2] <= 0 or coord[3] <= 0:print("Error encountered!\nFind out 0 height(width)!")print("This bounding box has been discarded! ")continue# print("Pull out corrd matrix:\n")# print(coord)# exit(-1)if not 0 < coord[-1] < 11:# class 0 and 11 are not in our interestcontinueif mode == "VisDrone2019-DET-val" or "VisDrone2019-DET-test":# in this case, score is the last 2 element.# No consideration for score 0 in evalif int(coord[-2]) == 0:continueif int(coord_raw[-2]) == 2:continuebbox_left, bbox_top = coord[0], coord[1]bbox_right, bbox_bottom = coord[0] + coord[2], coord[1] + coord[3]# Scale class number back to range 0-9center_x, center_y = int((bbox_left + bbox_right) * 0.5), int((bbox_top + bbox_bottom) * 0.5)format_data.append([center_x, center_y, coord[-1] - 1])# if not filename:# continue# if filename not in point_class_pair:# point_class_pair[filename] = {}# coord_pair = str(center_x) + " " + str(center_y)# if coord_pair not in point_class_pair[filename]:# point_class_pair[filename][coord_pair] = coord[-1] - 1# else:# if point_class_pair[filename][coord_pair] != coord[-1] - 1:# assert True, \# "duplicate coordination shows in current file : " + str(filename)return np.array(format_data)def parse_args():parser = argparse.ArgumentParser(description='DMNet--Density map ground truth generation')parser.add_argument('root_dir', default=".",help='the path for source data')parser.add_argument('precomputed_kernels_path', default="gaussian_kernels.pkl",help='the path to save precomputed kernels')parser.add_argument('precomputed_distances_path', default="distances_dict.pkl",help='the path to save precomputed distance')parser.add_argument('--image_prefix', default=".jpg", help='the path to save precomputed distance')parser.add_argument('--mode', default="train", help='Indicate if you are working on train/val/test set')parser.add_argument('--showden', action='store_true', help='show results')args = parser.parse_args()return argsif __name__ == "__main__":# General setupargs = parse_args()data_limit = Noneprecomputed_kernels_path = args.precomputed_kernels_pathprecomputed_distances_path = args.precomputed_distances_pathimg_affix = args.image_prefixshowden = args.showdenmode = args.moderoot_dir = args.root_dirmin_sigma = 0max_sigma = 20# create dir to save train/val density mapif not os.path.exists(os.path.join('/root/autodl-tmp/VisDrone2019/VisDrone2019-DET-train', 'dens')):os.makedirs(os.path.join('/root/autodl-tmp/VisDrone2019/VisDrone2019-DET-train', 'dens'), exist_ok=True)if not os.path.exists(os.path.join('/root/autodl-tmp/VisDrone2019/VisDrone2019-DET-val', 'dens')):os.makedirs(os.path.join('/root/autodl-tmp/VisDrone2019/VisDrone2019-DET-val', 'dens'), exist_ok=True)if not os.path.exists(os.path.join('/root/autodl-tmp/VisDrone2019/VisDrone2019-DET-test-dev', 'dens')):os.makedirs(os.path.join('/root/autodl-tmp/VisDrone2019/VisDrone2019-DET-test-dev', 'dens'), exist_ok=True)# create pre-computed kernel to speed up density map generationgenerate_gaussian_kernels(precomputed_kernels_path, round_decimals=3, sigma_threshold=4,sigma_min=min_sigma, sigma_max=max_sigma, num_sigmas=801)with open(precomputed_kernels_path, 'rb') as f:kernels_dict = pickle.load(f)kernels_dict = SortedDict(kernels_dict)# uncomment to generate and save dict with distancescompute_distances(out_dist_path=precomputed_distances_path, raw_label_dir=root_dir, mode=mode)with open(precomputed_distances_path, 'rb') as f:distances_dict = pickle.load(f)# print(distances_dict)data_root = modeimg_paths = glob.glob(f'{root_dir}/{data_root}/images/*.jpg')method = 3const_sigma = 15with open(str(mode) + ".txt", "w") as fileloader:# Prepared for the training algorithms that requires a txt output file# with all input images listedfor img_path in tqdm(img_paths):fileloader.write(img_path)fileloader.write("\n")data_folder, img_sub_path = img_path.split('images')ann_path = img_path.replace(img_affix, '.txt')ann_path = ann_path.replace("images", 'annotations')# load img and gtimg = Image.open(img_path)# print(img_path)width, height = img.sizegt_points = get_gt_dots(ann_path, height, width, mode=mode)distances = distances_dict[img_path]density_map = gaussian_filter_density(gt_points, height, width, distances,kernels_dict, min_sigma=min_sigma, method=method,const_sigma=const_sigma)den_name = os.path.join(root_dir, data_root, "dens", img_path.split("/")[-1].replace("jpg", "npy"))print(den_name)if showden:plt.imshow(img)plt.imshow(density_map, alpha=0.75)plt.show()else:np.save(den_name, density_map)
使用以下命令进行创建:
# 生成训练集gt密度图
python image_cropping/Generate_density_map_official.py /root/autodl-tmp/VisDrone2019 gaussian_kernels.pkl distances_dict.pkl --mode VisDrone2019-DET-train
# 生成验证集gt密度图
python image_cropping/Generate_density_map_official.py /root/autodl-tmp/VisDrone2019 gaussian_kernels.pkl distances_dict.pkl --mode VisDrone2019-DET-val
# 生成测试集gt密度图
python image_cropping/Generate_density_map_official.py /root/autodl-tmp/VisDrone2019 gaussian_kernels.pkl distances_dict.pkl --mode VisDrone2019-DET-test-dev
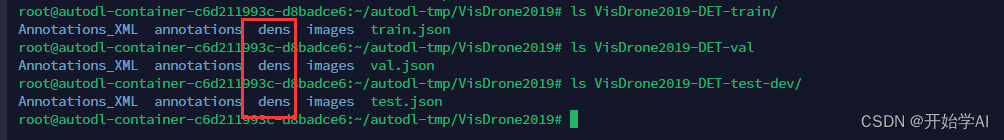
在以上文件夹 生成了如下的dens文件夹 保存了密度图的npy文件。
密度图预测
根据官方仓库说明 下载MCNN代码 进行训练预测即可
裁剪区域
# 生成训练集
python image_cropping/density_slide_window_official.py /root/autodl-tmp/VisDrone2019 70_70 0.08 --output_folder /root/autodl-tmp/VisDrone2019/Crop_70_0.08 --mode VisDrone2019-DET-train
# 生成验证集
python image_cropping/density_slide_window_official.py /root/autodl-tmp/VisDrone2019 70_70 0.08 --output_folder /root/autodl-tmp/VisDrone2019/Crop_70_0.08 --mode VisDrone2019-DET-val# 生成测试集
python image_cropping/density_slide_window_official.py /root/autodl-tmp/VisDrone2019 70_70 0.08 --output_folder /root/autodl-tmp/VisDrone2019/Crop_70_0.08 --mode VisDrone2019-DET-test-dev
部分切分后的效果图,可以看到部分目标还是存在截断现象,是一个优化点。





由于不清楚是否会将crop后的图像与标签,用于到训练过程中,这里我就不生成训练的crop图和标签了,下一步准备直接用训练好的模型进行融合测试。论文是采用验证集进行测试的,这儿我准备把验证集和测试集(test-dev)都测试一遍。

最后按以下结构需要组织一下文件

最后我的目录结构如下
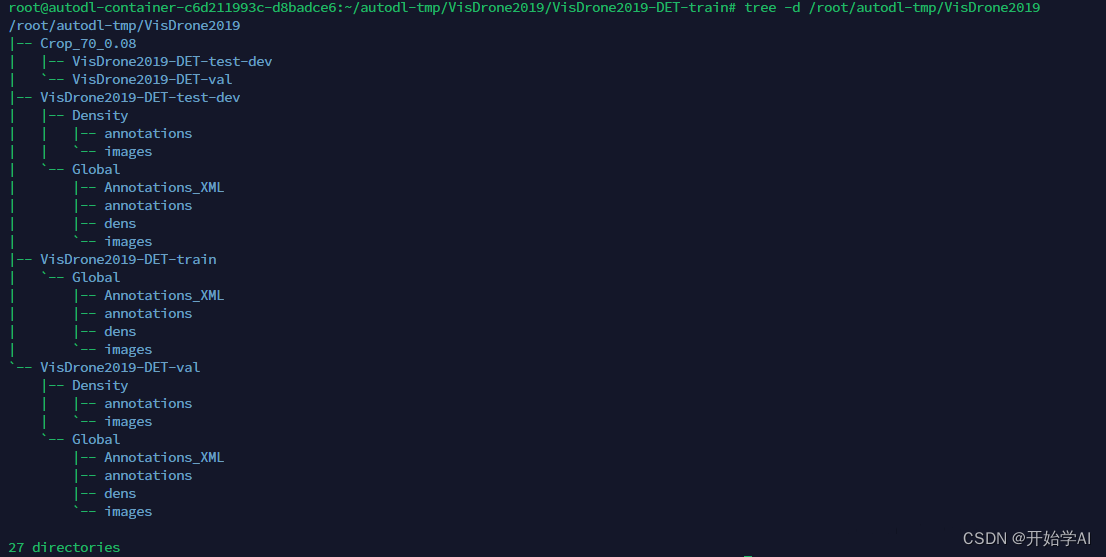
数据格式转换
需要先把txt转为voc,再把voc转为coco
# 生成测试集的voc格式
python fusion_detection/create_VOC_annotation_official.py /root/autodl-tmp/VisDrone2019 --output_folder VisDrone2019_finally --mode VisDrone2019-DET-test-dev
# 生成测试集的coco格式
python fusion_detection/VOC2coco_official.py /root/autodl-tmp/VisDrone2019/VisDrone2019_finally --mode VisDrone2019-DET-test-dev
这过程太耗时间了。最后把中间文件夹删除,整个格式如以下:
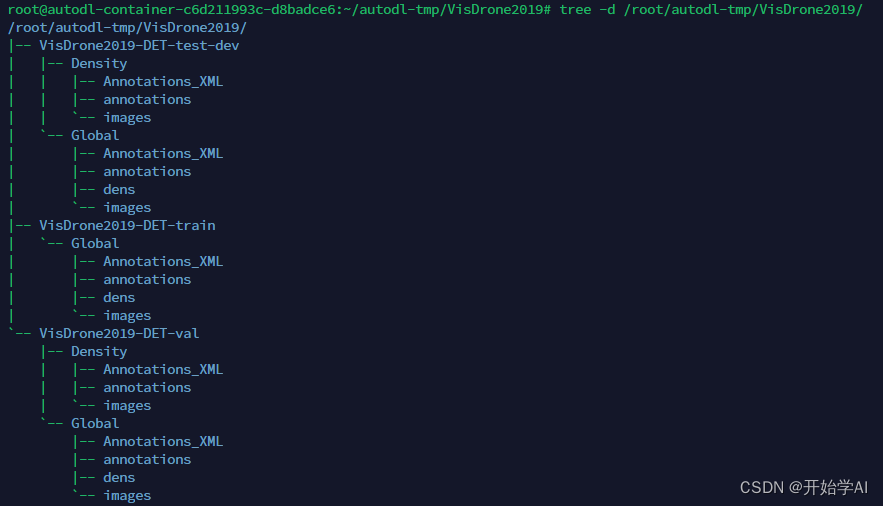
其中coco的json文件在以下位置
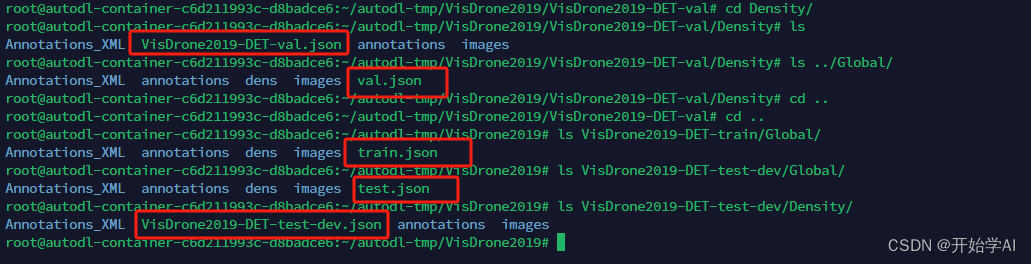
过程太繁琐了。
有需要生成后的数据,可以联系!
相关文章:

DMNet复现(一)之数据准备篇:Density map guided object detection in aerial image
一、生成密度图 密度图标签生成 采用以下代码,生成训练集密度图gt: import cv2 import glob import h5py import scipy import pickle import numpy as np from PIL import Image from itertools import islice from tqdm import tqdm from matplotli…...

k8s相关命令-命名空间
k8s相关命令目录 文章目录 前言一、创建命名空间二、删除命名空间三、查看命名空间列表四、查看命名空间列表五、查看特定命名空间下所有资源六、删除特定命名空间下所有资源 前言 记录k8s命名空间的相关操作命令 一、创建命名空间 kubectl create namespace <namespace&g…...

CG Magic分享同一场景里下,VR渲染器和CR渲染器哪个好?
渲染操作时,VR渲染器和CR渲染器的对比成为常见问题了。这个问题很多人都会问。 今天CG Magic小编通过一个真实的项目,就是同一场景下来比较一下VR渲染器和CR渲染器的区别。 以下图为例是用来测试的场景当年的最终图。采用了当年的一个伊丽莎白大街152号的…...
Hive工作原理
Hive 工作原理详解-阿里云开发者社区 Hive的服务端组件 1. Driver组件:该组件包括:Compiler、Optimizer、Executor,它可以将Hive的编译、解析、优化转化为MapReduce任务提交给Hadoop1中的JobTracker或者是Hadoop2中的SourceManager来进行实际的执行相应…...

vue 使用this.$set设置对象属性值时,不更新试图
vue 使用this.$set设置对象属性值时,不更新试图。 后来发现是因为若对象中存在该属性时,只更新值,不添加响应监测。 //vue/src/core/observer/index.js 源码片段/*** Set a property on an object. Adds the new property and* triggers ch…...
uniapp视频播放功能
UniApp提供了多种视频播放组件,包括视频播放器(video)、多媒体组件(media)、WebView(内置Video标签)等。其中,video和media组件是最常用的。 video组件 video组件是基于HTML5 vide…...

Java面向对象七大原则以及设计模式单例模式和工厂模式简单工厂模式
面向对象的七大原则(OOP) 1,开闭原则: 对扩展开发,对修改关闭 2.里氏替换原则: 继承必须确保超类所拥有的子类的性质在子类中仍然成立 3.依赖倒置原则: 面向接口编程,不要面向实现编程&am…...

Linux 遍历目录(cd 命令)
Linux 遍历目录(cd 命令) 文章目录 Linux 遍历目录(cd 命令)一、cd 命令二、绝对文件路径三、相对文件路径 一、cd 命令 在 Linux 文件系统上,可以使用 cd 命令将 shell 会话切换到另一个目录。cd 命令的格式也很简单…...
整合Nginx实现反向代理
针对后端启动多个服务,接口需要统一请求路径时,可以使用nginx进行请求地址反向代理。 1.下载: nginx 2.下载完成后解压,找到配置文件nginx.conf(在解压文件的conf目录中),在http中增加以下示例代码&#x…...

Linux:IP转INT详解
一、IP地址介绍 IP地址(Internet Protocol Address)是指互联网协议地址,是所有连接到网络设备的唯一标识符。IP地址由32位二进制数表示,通常以四段十进制数(每个数值范围为0-255)表示,例如192.1…...
43.MQ—RabbitMQ
目录 一、MQ—RabbitMQ。 (1)同步调用与异步调用。 (1.1)同步调用。 (1.2)异步调用。 (2)MQ之间的区别。 (3)RabbitMQ学习。 (3.1…...
)
Leetcode154. 寻找旋转排序数组中的最小值(存在重复元素)
力扣(LeetCode)官网 - 全球极客挚爱的技术成长平台 已知一个长度为 n 的数组,预先按照升序排列,经由 1 到 n 次 旋转 后,得到输入数组。例如,原数组 nums [0,1,4,4,5,6,7] 在变化后可能得到:…...

docker查看镜像的latest对应的具体版本
查询容器镜像时,TAG只显示latest,而不是显示具体的版本号 docker images # 显示内容 REPOSITORY TAG IMAGE ID CREATED SIZE nginx latest 605c77e624dd 20 months ago 141MB redis latest 7614ae945…...
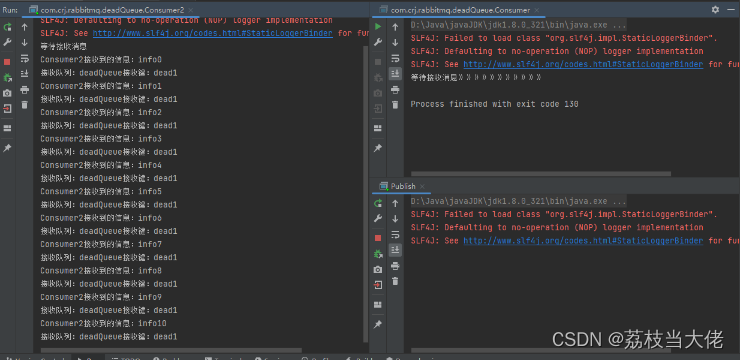
RabbitMQ深入 —— 死信队列
前言 前面荔枝梳理了RabbitMQ中的普通队列、交换机以及相关的知识,在这篇文章中荔枝将会梳理RabbitMQ的一个重要的队列 —— 死信队列,主要了解消息流转到死信队列的三种的方式以及相应的实现demo。希望能帮助到有需要的小伙伴~~~ 文章目录 前言 死信队…...

【React + Umi】自定义离开页面拦截弹框事件
在 react umi 中对离开页面的行为进行自定义弹窗拦截控制。以下为可选的方案分析。 wrapper 首先,因为项目框架是 umi,最先想到了 umi 路由的 wrapper 装饰器,但仔细一想又不太对, wrapper 争对于跳转到某个特定页面的前置行为…...
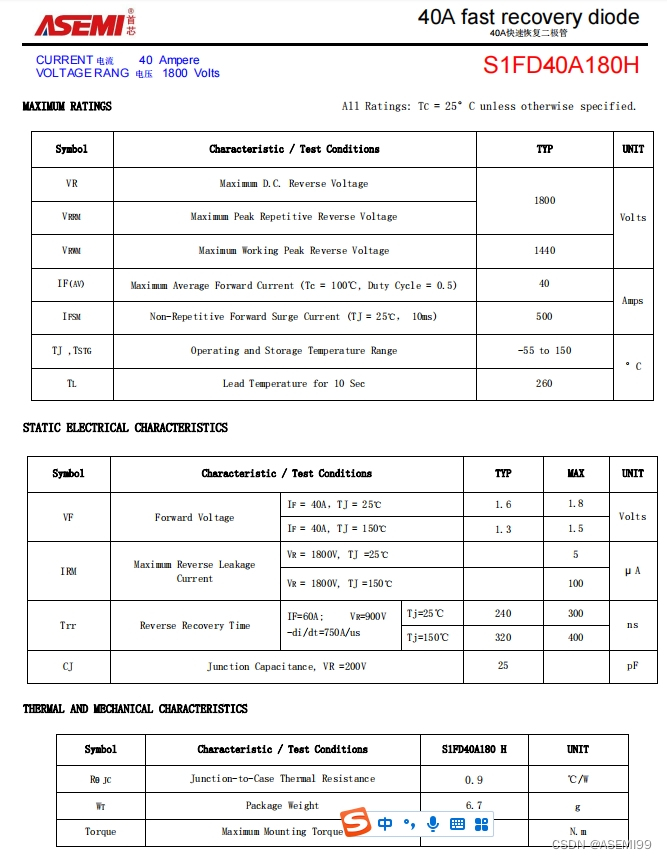
S1FD40A180H-ASEMI快恢复二极管S1FD40A180H
编辑:ll S1FD40A180H-ASEMI快恢复二极管S1FD40A180H 型号:S1FD40A180H 品牌:ASEMI 封装:TO-247 特性:大功率、快恢复二极管 正向电流:40A 反向耐压:1800V 恢复时间:<300n…...
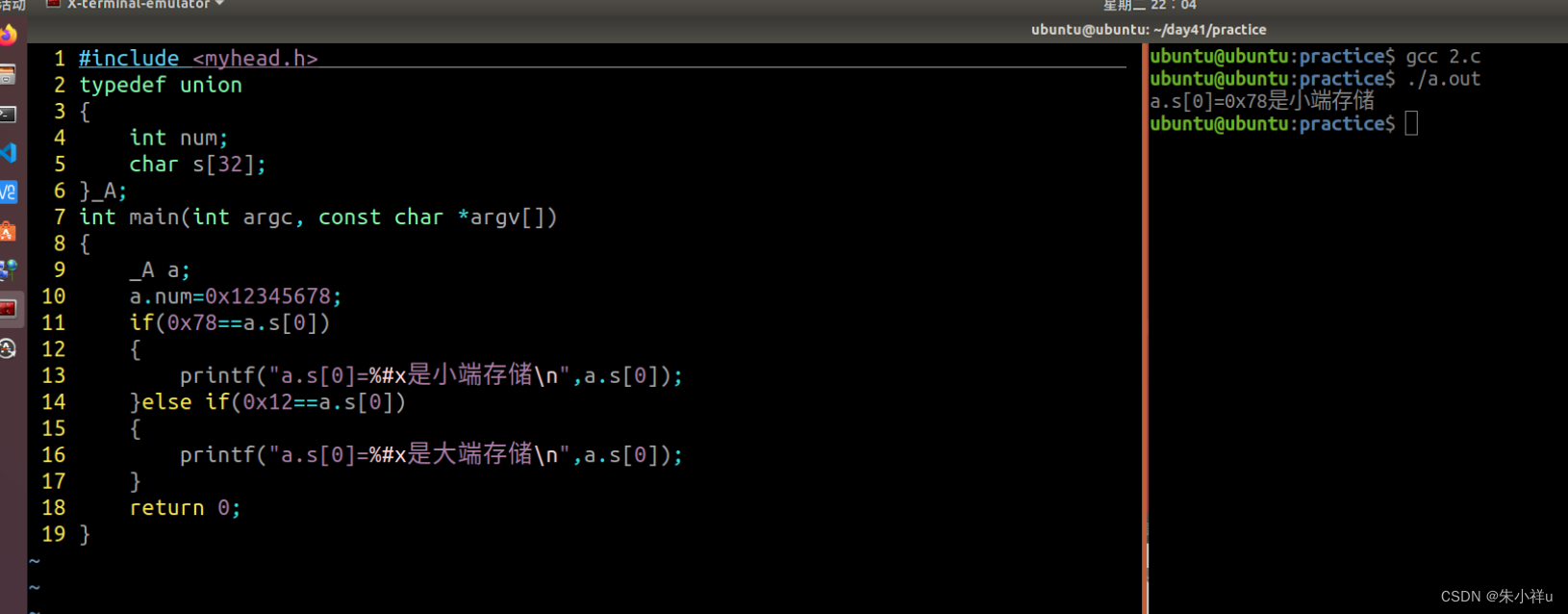
网络编程 day1
1->x.mind网络编程基础 2->简述字节序的概念,并用共用体(联合体)的方式计算本机的字节序 1.字节序是指不同类型的CPU主机,内存存储多字节整数序列的方式 2.小端字节序:低序字节存储在低地址上 3.大端字节序&a…...
《深入PostgreSQL的存储引擎:原理与性能》
🌷🍁 博主猫头虎(🐅🐾)带您 Go to New World✨🍁 🐅🐾猫头虎建议程序员必备技术栈一览表📖: 🛠️ 全栈技术 Full Stack: 📚…...

python开发之个微群聊机器人的开发
简要描述: 退出群聊 请求URL: http://域名地址/quitChatRoom 请求方式: POST 请求头Headers: Content-Type:application/jsonAuthorization:login接口返回 参数: 参数名必选类型说明wI…...
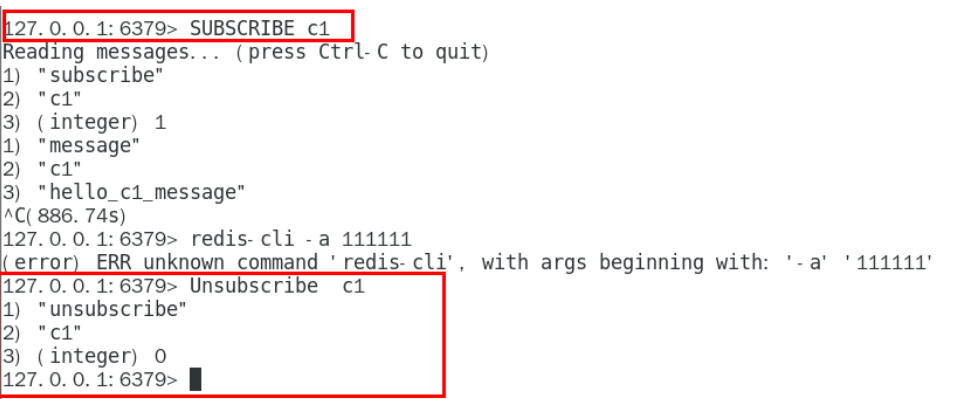
【Redis7】--4.事务、管道、发布和订阅
文章目录 事务1.Redis事务2.Redis事务特性3.Redis事务命令3.1MULTI3.2EXEC3.3DISCARD3.4WATCH3.5UNWATCH 4.不保证原子性4.1"全体连坐"4.2"冤头债主" 5.事务执行流程 管道1.pipeline的使用2.pipeline小总结 发布和订阅1.常用命令1.1SUBSCRIBE1.2PUBLISH1.3…...
)
uniapp 对接腾讯云IM群组成员管理(增删改查)
UniApp 实战:腾讯云IM群组成员管理(增删改查) 一、前言 在社交类App开发中,群组成员管理是核心功能之一。本文将基于UniApp框架,结合腾讯云IM SDK,详细讲解如何实现群组成员的增删改查全流程。 权限校验…...

【OSG学习笔记】Day 18: 碰撞检测与物理交互
物理引擎(Physics Engine) 物理引擎 是一种通过计算机模拟物理规律(如力学、碰撞、重力、流体动力学等)的软件工具或库。 它的核心目标是在虚拟环境中逼真地模拟物体的运动和交互,广泛应用于 游戏开发、动画制作、虚…...
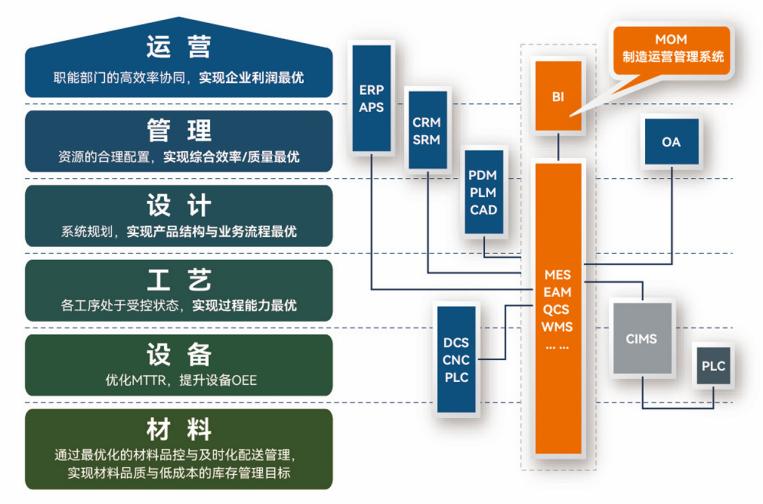
盘古信息PCB行业解决方案:以全域场景重构,激活智造新未来
一、破局:PCB行业的时代之问 在数字经济蓬勃发展的浪潮中,PCB(印制电路板)作为 “电子产品之母”,其重要性愈发凸显。随着 5G、人工智能等新兴技术的加速渗透,PCB行业面临着前所未有的挑战与机遇。产品迭代…...

Frozen-Flask :将 Flask 应用“冻结”为静态文件
Frozen-Flask 是一个用于将 Flask 应用“冻结”为静态文件的 Python 扩展。它的核心用途是:将一个 Flask Web 应用生成成纯静态 HTML 文件,从而可以部署到静态网站托管服务上,如 GitHub Pages、Netlify 或任何支持静态文件的网站服务器。 &am…...

镜像里切换为普通用户
如果你登录远程虚拟机默认就是 root 用户,但你不希望用 root 权限运行 ns-3(这是对的,ns3 工具会拒绝 root),你可以按以下方法创建一个 非 root 用户账号 并切换到它运行 ns-3。 一次性解决方案:创建非 roo…...

Linux云原生安全:零信任架构与机密计算
Linux云原生安全:零信任架构与机密计算 构建坚不可摧的云原生防御体系 引言:云原生安全的范式革命 随着云原生技术的普及,安全边界正在从传统的网络边界向工作负载内部转移。Gartner预测,到2025年,零信任架构将成为超…...
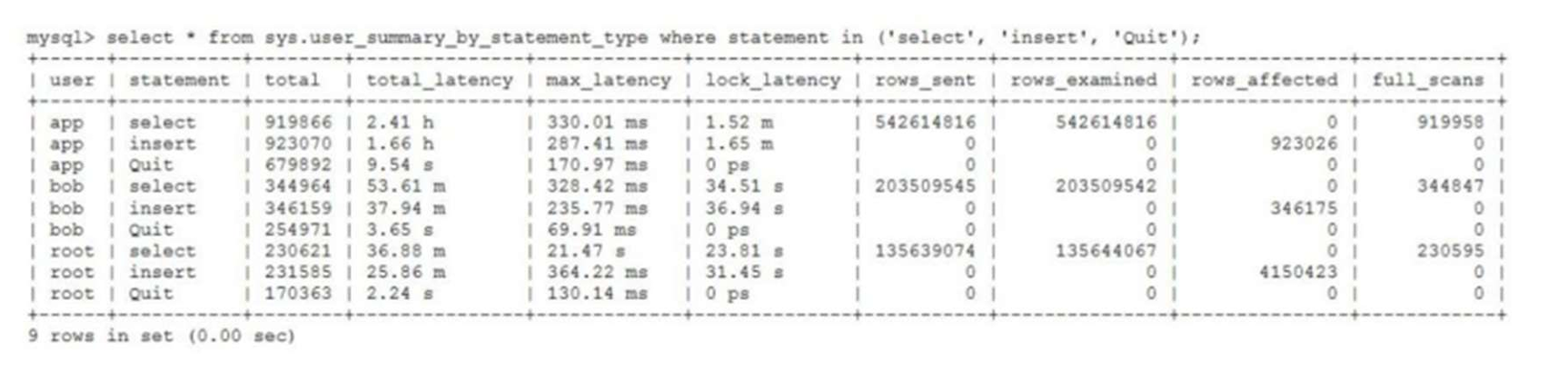
MySQL 8.0 OCP 英文题库解析(十三)
Oracle 为庆祝 MySQL 30 周年,截止到 2025.07.31 之前。所有人均可以免费考取原价245美元的MySQL OCP 认证。 从今天开始,将英文题库免费公布出来,并进行解析,帮助大家在一个月之内轻松通过OCP认证。 本期公布试题111~120 试题1…...
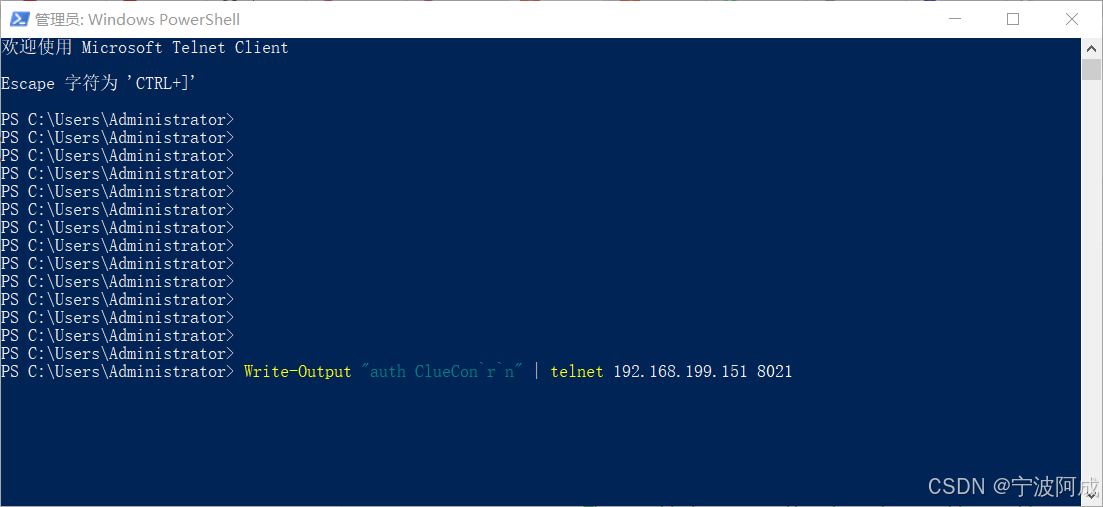
用docker来安装部署freeswitch记录
今天刚才测试一个callcenter的项目,所以尝试安装freeswitch 1、使用轩辕镜像 - 中国开发者首选的专业 Docker 镜像加速服务平台 编辑下面/etc/docker/daemon.json文件为 {"registry-mirrors": ["https://docker.xuanyuan.me"] }同时可以进入轩…...

tree 树组件大数据卡顿问题优化
问题背景 项目中有用到树组件用来做文件目录,但是由于这个树组件的节点越来越多,导致页面在滚动这个树组件的时候浏览器就很容易卡死。这种问题基本上都是因为dom节点太多,导致的浏览器卡顿,这里很明显就需要用到虚拟列表的技术&…...

音视频——I2S 协议详解
I2S 协议详解 I2S (Inter-IC Sound) 协议是一种串行总线协议,专门用于在数字音频设备之间传输数字音频数据。它由飞利浦(Philips)公司开发,以其简单、高效和广泛的兼容性而闻名。 1. 信号线 I2S 协议通常使用三根或四根信号线&a…...
