【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割6(数据预处理)
由于之前哔站作者整理的LUNA16数据处理方式过于的繁琐,于是,本文就对LUNA16数据做一个新的整理,最终得到的数据和形式是差不多的。但是,主要不同的是代码逻辑比较的简单,便于理解。
对于数据集的学习,可以去参考这里:【LIDC-IDRI】 CT 肺结节 XML 标记特征良恶性标签PKL转储(一)
步骤和中心内容,包括一下几个部分:
masks生成:从xml文件中,抽取出对应序列series的结节标记位置坐标(可能一个结节多人多次标注),生成对应的mask数组文件,大小与图像数组大小一致;- 肺实质提取操作:从肺区分割的数据中,与原始图像和
mask图做乘积操作,非肺区部分进行填充,或者去除操作均可; resample操作:根据spacing,进行resample操作,可以在zyx三个维度进行resample,也可以仅仅在z方向进行resample操作位1mm(这个我在论文中看到有类似这样做的);- 根据
mask,获取结节的zyx中心点坐标,和半径。
至此,我们将收获以下几个文件:
- 包含
ct的图像数据; - 对应的
mask数据; - 记录
zyx中心点坐标,和半径的文件。
相比于luna16给出的数据形式,目前的数据就比较好理解,和方便查看了。无论是可视化,还是后续的数据处理和训练,都更加的直观、明了。后面就会针对这部分,一一进行展开。
由于代码量还是比较大,处理的东西,和涉及的文件比较多,可能会几个篇章展开。本篇就先对xml文件进行处理,转出来,以便于查看。这里涉及到xml文件的格式,和处理,就单独开一篇,链接去参考:【医学影像数据处理】 XML 文件格式处理汇总
一、xml文件转储
1.1、认识标注文件xml
对于LIDC-IDRI数据集中,xml文件内各个字段表示什么意思的介绍,可以参考我的另一篇文章,点击这里:【LIDC-IDRI】 CT 肺结节 XML 标记特征良恶性标签PKL转储(一)
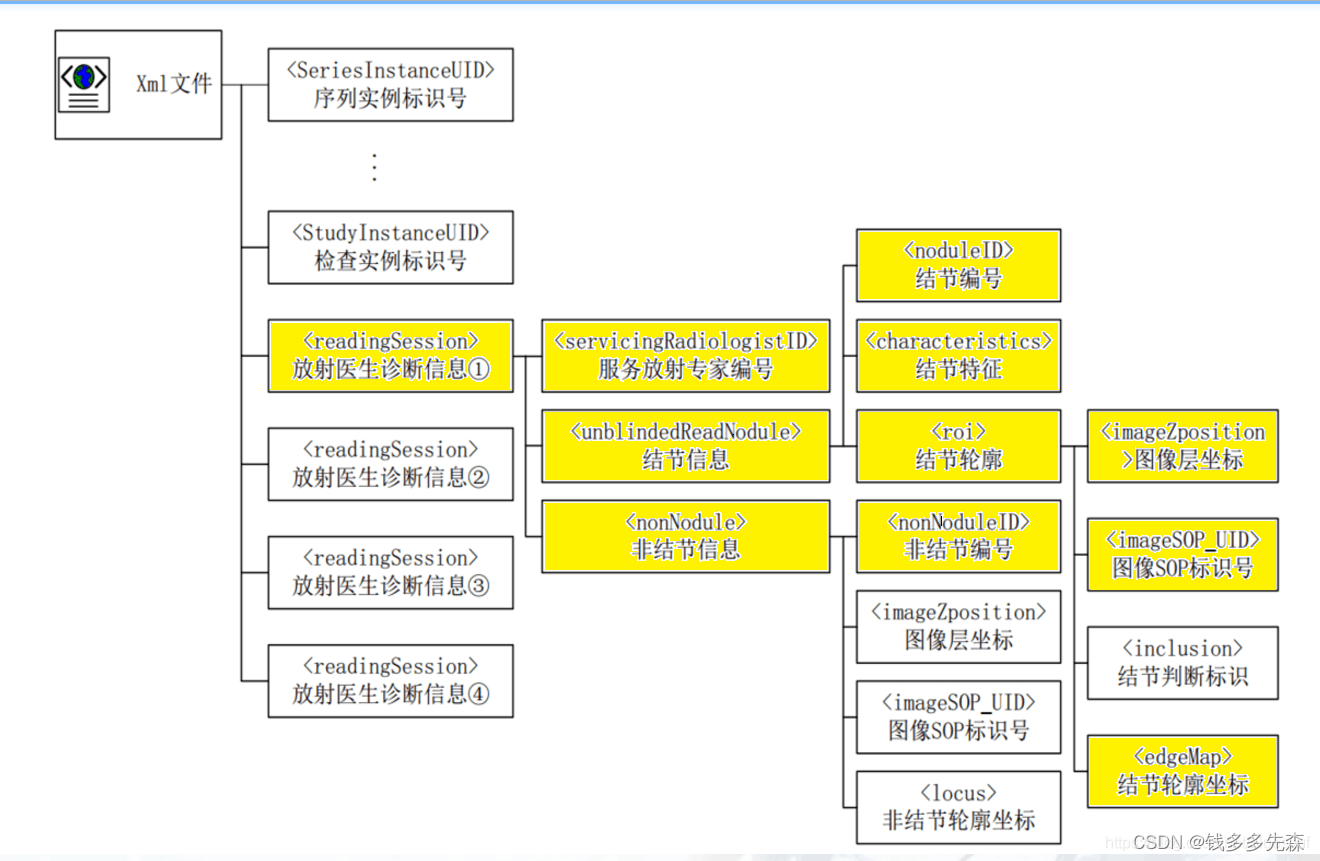
在这篇文章里面,着重介绍了这个数据的结构,以及xml各个记录的tag是什么意思。相信你看完,对这个数据集的处理,有更深的理解。
其中大部分代码都是跟上面这个链接介绍和获取的内容是一样的,可以参考这个GitHub:NoduleNet - utils -LIDC
有些内容没有介绍到,简单做个补充
ResponseHeader:这个是头部分,记录了这个病例(也就是单个病人的CT图像)的信息。
为了方便查看,和学习xml文件,可以参考这篇文章:【医学影像数据处理】 XML 文件格式处理汇总。我们就采用其中xml转字典的形式,方便我们查看。下面就展示了转字典后的前后部分内容对比,如下:
原始xml的数据形式,节选了其中的一小段,展示如下:
<?xml version="1.0" encoding="UTF-8"?>
<LidcReadMessage uid="1.3.6.1.4.1.14519.5.2.1.6279.6001.1308168927505.0" xmlns="http://www.nih.gov" xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" xsi:schemaLocation="http://www.nih.gov http://troll.rad.med.umich.edu/lidc/LidcReadMessage.xsd"><ResponseHeader><Version>1.7</Version><MessageId>1148851</MessageId><DateRequest>2005-11-03</DateRequest><TimeRequest>12:25:10</TimeRequest><RequestingSite>removed</RequestingSite><ServicingSite>removed</ServicingSite><TaskDescription>Second unblinded read</TaskDescription><CtImageFile>removed</CtImageFile><SeriesInstanceUid>1.3.6.1.4.1.14519.5.2.1.6279.6001.131939324905446238286154504249</SeriesInstanceUid><StudyInstanceUID>1.3.6.1.4.1.14519.5.2.1.6279.6001.303241414168367763244410429787</StudyInstanceUID><DateService>2005-11-03</DateService><TimeService>12:25:40</TimeService><ResponseDescription>1 - Reading complete</ResponseDescription><ResponseComments></ResponseComments></ResponseHeader>
转换成dictionary字典后的形式。(更便于查看了)
{"LidcReadMessage": {"@uid": "1.3.6.1.4.1.14519.5.2.1.6279.6001.1308168927505.0","@xmlns": "http://www.nih.gov","@xmlns:xsi": "http://www.w3.org/2001/XMLSchema-instance","@xsi:schemaLocation": "http://www.nih.gov http://troll.rad.med.umich.edu/lidc/LidcReadMessage.xsd","ResponseHeader": {"Version": "1.7","MessageId": "1148851","DateRequest": "2005-11-03","TimeRequest": "12:25:10","RequestingSite": "removed","ServicingSite": "removed","TaskDescription": "Second unblinded read","CtImageFile": "removed","SeriesInstanceUid": "1.3.6.1.4.1.14519.5.2.1.6279.6001.131939324905446238286154504249","StudyInstanceUID": "1.3.6.1.4.1.14519.5.2.1.6279.6001.303241414168367763244410429787","DateService": "2005-11-03","TimeService": "12:25:40","ResponseDescription": "1 - Reading complete","ResponseComments": null},
}
1.2、xml综合记录转为按series的npy文件
LIDC-IDRI有1018个检查,在标记文件夹tcia-lidc-xml中6 个文件夹中,有1318 个xml文件。并且,这些xml文件的名称,和图像的序列名称不是一一对应的。
所以,就需要现将xml文件内标注的信息,给重新整理出来,转为人能轻易看懂和理解的内容。并且,标注文件如果能与图像文件是一一对应的,那么后续的处理也会方便了许多。
这一小节做的事情,就是将xml文件,给抽取出来,留下关心的内容,其他不重要的,不关心的内容暂时不管。
下面是处理的代码,主要的步骤如下概述:
- 遍历所有的
xml文件,一一处理; - 对单个
xml文件,解析出seriesuid和标注的结节坐标; - 存储到以
seriesuid命名的npy文件,存储的内容就是一个个结节坐标。
完整代码如下:
from tqdm import tqdm
import sys
import os
import numpy as npfrom pylung.utils import find_all_files
from pylung.annotation import parsedef xml2mask(xml_file):header, annos = parse(xml_file) # get one xml infoctr_arrs = []for i, reader in enumerate(annos):for j, nodule in enumerate(reader.nodules):ctr_arr = []for k, roi in enumerate(nodule.rois):z = roi.zfor roi_xy in roi.roi_xy:ctr_arr.append([z, roi_xy[1], roi_xy[0]]) # [[[z, y, x], [z, y, x]]]ctr_arrs.append(ctr_arr)seriesuid = header.series_instance_uidreturn seriesuid, ctr_arrsdef annotation2masks(annos_dir, save_dir):# get all xml file pathfiles = find_all_files(annos_dir, '.xml')for f in tqdm(files, total=len(files)):print(f)try:seriesuid, masks = xml2mask(f)np.save(os.path.join(save_dir, '%s' % (seriesuid)), masks) # save xml 3D coor [[z, y, x], [z, y, x]]except:print("Unexpected error:", sys.exc_info()[0])if __name__ == '__main__':annos_dir = './LUNA16/annotation/LIDC-XML-only/tcia-lidc-xml' # .xmlctr_arr_save_dir = './LUNA16/annotation/noduleCoor' # 保存每个注释器解析的中间结节mask的地方os.makedirs(ctr_arr_save_dir, exist_ok=True)# xml信息,转储npy(临时文件)annotation2masks(annos_dir, ctr_arr_save_dir)
下面打开一个·npy·文件进行查看,记录的内容如下,是所有医生对这个序列标注的所有结节的polygon坐标点:
[list([[-299.8, 206, 42], [-299.8, 207, 41], [-299.8, 208, 41], [-299.8, 209, 40], [-299.8, 210, 40], [-299.8, 211, 41], [-299.8, 212, 41], [-299.8, 213, 42], [-299.8, 214, 42], [-299.8, 215, 43], [-299.8, 216, 44], [-299.8, 216, 45], [-299.8, 215, 46], [-299.8, 215, 47], [-299.8, 215, 48], [-299.8, 214, 49], [-299.8, 213, 49], [-299.8, 212, 49], [-299.8, 211, 49], [-299.8, 210, 49], [-299.8, 209, 49], [-299.8, 208, 48], [-299.8, 207, 47], [-299.8, 207, 46], [-299.8, 206, 45], [-299.8, 206, 44], [-299.8, 206, 43], [-299.8, 206, 42], [-298.0, 206, 46], [-298.0, 207, 45], [-298.0, 207, 44], [-298.0, 208, 43], [-298.0, 209, 42], [-298.0, 209, 41], [-298.0, 210, 40], [-298.0, 211, 40], [-298.0, 212, 39], [-298.0, 213, 40], [-298.0, 214, 41], [-298.0, 215, 42], [-298.0, 215, 43], [-298.0, 216, 44], [-298.0, 216, 45], [-298.0, 216, 46], [-298.0, 216, 47], [-298.0, 215, 48], [-298.0, 214, 48], [-298.0, 213, 48], [-298.0, 212, 48], [-298.0, 211, 48], [-298.0, 210, 48], [-298.0, 209, 48], [-298.0, 208, 48], [-298.0, 207, 47], [-298.0, 206, 46], [-296.2, 209, 42], [-296.2, 210, 41], [-296.2, 211, 40], [-296.2, 212, 40], [-296.2, 213, 41], [-296.2, 214, 42], [-296.2, 215, 43], [-296.2, 216, 44], [-296.2, 216, 45], [-296.2, 216, 46], [-296.2, 216, 47], [-296.2, 216, 48], [-296.2, 215, 49], [-296.2, 214, 49], [-296.2, 213, 49], [-296.2, 212, 49], [-296.2, 211, 48], [-296.2, 210, 47], [-296.2, 209, 46], [-296.2, 209, 45], [-296.2, 209, 44], [-296.2, 209, 43], [-296.2, 209, 42]])list([[-227.8, 151, 405], [-227.8, 152, 404], [-227.8, 153, 403], [-227.8, 154, 402], [-227.8, 155, 402], [-227.8, 156, 402], [-227.8, 157, 403], [-227.8, 157, 404], [-227.8, 157, 405], [-227.8, 158, 406], [-227.8, 158, 407], [-227.8, 158, 408], [-227.8, 157, 409], [-227.8, 156, 409], [-227.8, 155, 409], [-227.8, 154, 408], [-227.8, 153, 408], [-227.8, 152, 407], [-227.8, 151, 406], [-227.8, 151, 405], [-226.0, 152, 405], [-226.0, 153, 404], [-226.0, 154, 404], [-226.0, 155, 403], [-226.0, 156, 404], [-226.0, 157, 405], [-226.0, 157, 406], [-226.0, 157, 407], [-226.0, 156, 408], [-226.0, 155, 408], [-226.0, 154, 408], [-226.0, 153, 408], [-226.0, 152, 407], [-226.0, 152, 406], [-226.0, 152, 405]])list([[-226.0, 158, 407], [-226.0, 157, 408], [-226.0, 156, 409], [-226.0, 155, 409], [-226.0, 154, 409], [-226.0, 153, 409], [-226.0, 152, 408], [-226.0, 151, 407], [-226.0, 152, 406], [-226.0, 153, 405], [-226.0, 153, 404], [-226.0, 154, 403], [-226.0, 155, 402], [-226.0, 156, 402], [-226.0, 157, 403], [-226.0, 158, 404], [-226.0, 158, 405], [-226.0, 158, 406], [-226.0, 158, 407], [-227.8, 159, 407], [-227.8, 158, 408], [-227.8, 157, 409], [-227.8, 156, 410], [-227.8, 155, 410], [-227.8, 154, 410], [-227.8, 153, 409], [-227.8, 152, 408], [-227.8, 151, 407], [-227.8, 151, 406], [-227.8, 151, 405], [-227.8, 152, 404], [-227.8, 153, 403], [-227.8, 154, 402], [-227.8, 155, 402], [-227.8, 156, 402], [-227.8, 157, 403], [-227.8, 158, 404], [-227.8, 158, 405], [-227.8, 158, 406], [-227.8, 159, 407]])list([[-296.2, 214, 46], [-296.2, 213, 47], [-296.2, 212, 47], [-296.2, 211, 47], [-296.2, 210, 46], [-296.2, 209, 45], [-296.2, 208, 44], [-296.2, 208, 43], [-296.2, 208, 42], [-296.2, 209, 41], [-296.2, 210, 42], [-296.2, 211, 42], [-296.2, 212, 43], [-296.2, 213, 44], [-296.2, 214, 45], [-296.2, 214, 46], [-298.0, 216, 47], [-298.0, 215, 48], [-298.0, 214, 49], [-298.0, 213, 49], [-298.0, 212, 49], [-298.0, 211, 49], [-298.0, 210, 49], [-298.0, 209, 48], [-298.0, 208, 47], [-298.0, 207, 46], [-298.0, 207, 45], [-298.0, 207, 44], [-298.0, 208, 43], [-298.0, 208, 42], [-298.0, 209, 41], [-298.0, 210, 41], [-298.0, 211, 41], [-298.0, 212, 41], [-298.0, 213, 41], [-298.0, 214, 42], [-298.0, 215, 43], [-298.0, 216, 44], [-298.0, 216, 45], [-298.0, 216, 46], [-298.0, 216, 47], [-299.8, 216, 50], [-299.8, 215, 51], [-299.8, 214, 51], [-299.8, 213, 50], [-299.8, 212, 50], [-299.8, 211, 50], [-299.8, 210, 49], [-299.8, 209, 48], [-299.8, 208, 47], [-299.8, 207, 46], [-299.8, 207, 45], [-299.8, 207, 44], [-299.8, 208, 43], [-299.8, 209, 42], [-299.8, 210, 42], [-299.8, 211, 41], [-299.8, 212, 41], [-299.8, 213, 42], [-299.8, 214, 42], [-299.8, 215, 43], [-299.8, 216, 44], [-299.8, 216, 45], [-299.8, 216, 46], [-299.8, 216, 47], [-299.8, 216, 48], [-299.8, 216, 49], [-299.8, 216, 50]])list([[-226.0, 158, 407], [-226.0, 157, 408], [-226.0, 156, 409], [-226.0, 155, 409], [-226.0, 154, 409], [-226.0, 153, 409], [-226.0, 152, 409], [-226.0, 151, 409], [-226.0, 151, 408], [-226.0, 151, 407], [-226.0, 151, 406], [-226.0, 151, 405], [-226.0, 152, 404], [-226.0, 152, 403], [-226.0, 153, 403], [-226.0, 154, 402], [-226.0, 154, 401], [-226.0, 155, 401], [-226.0, 156, 401], [-226.0, 157, 401], [-226.0, 157, 402], [-226.0, 158, 403], [-226.0, 158, 404], [-226.0, 158, 405], [-226.0, 158, 406], [-226.0, 158, 407], [-227.8, 159, 407], [-227.8, 158, 408], [-227.8, 158, 409], [-227.8, 157, 409], [-227.8, 156, 410], [-227.8, 155, 410], [-227.8, 154, 409], [-227.8, 153, 409], [-227.8, 152, 409], [-227.8, 151, 408], [-227.8, 151, 407], [-227.8, 151, 406], [-227.8, 151, 405], [-227.8, 151, 404], [-227.8, 152, 403], [-227.8, 152, 402], [-227.8, 153, 401], [-227.8, 154, 401], [-227.8, 155, 401], [-227.8, 156, 401], [-227.8, 157, 401], [-227.8, 158, 402], [-227.8, 158, 403], [-227.8, 159, 404], [-227.8, 159, 405], [-227.8, 159, 406], [-227.8, 159, 407]])list([[-296.2, 215, 47], [-296.2, 214, 48], [-296.2, 213, 48], [-296.2, 212, 48], [-296.2, 211, 48], [-296.2, 210, 47], [-296.2, 209, 47], [-296.2, 208, 46], [-296.2, 208, 45], [-296.2, 207, 44], [-296.2, 207, 43], [-296.2, 208, 42], [-296.2, 209, 42], [-296.2, 210, 42], [-296.2, 211, 42], [-296.2, 212, 43], [-296.2, 213, 43], [-296.2, 214, 44], [-296.2, 215, 45], [-296.2, 215, 46], [-296.2, 215, 47], [-298.0, 216, 47], [-298.0, 215, 48], [-298.0, 214, 49], [-298.0, 214, 50], [-298.0, 213, 50], [-298.0, 212, 50], [-298.0, 211, 49], [-298.0, 210, 49], [-298.0, 209, 48], [-298.0, 208, 48], [-298.0, 207, 47], [-298.0, 207, 46], [-298.0, 207, 45], [-298.0, 207, 44], [-298.0, 207, 43], [-298.0, 207, 42], [-298.0, 207, 41], [-298.0, 208, 41], [-298.0, 209, 41], [-298.0, 210, 41], [-298.0, 211, 41], [-298.0, 212, 41], [-298.0, 213, 41], [-298.0, 214, 41], [-298.0, 215, 42], [-298.0, 215, 43], [-298.0, 216, 44], [-298.0, 216, 45], [-298.0, 216, 46], [-298.0, 216, 47], [-299.8, 217, 46], [-299.8, 216, 47], [-299.8, 216, 48], [-299.8, 215, 49], [-299.8, 214, 50], [-299.8, 213, 50], [-299.8, 212, 50], [-299.8, 211, 50], [-299.8, 210, 50], [-299.8, 209, 49], [-299.8, 208, 48], [-299.8, 208, 47], [-299.8, 207, 46], [-299.8, 207, 45], [-299.8, 207, 44], [-299.8, 208, 43], [-299.8, 209, 42], [-299.8, 209, 41], [-299.8, 210, 41], [-299.8, 211, 41], [-299.8, 212, 41], [-299.8, 213, 41], [-299.8, 214, 42], [-299.8, 215, 42], [-299.8, 215, 43], [-299.8, 216, 44], [-299.8, 217, 45], [-299.8, 217, 46], [-301.6, 214, 45], [-301.6, 213, 46], [-301.6, 212, 47], [-301.6, 211, 47], [-301.6, 210, 46], [-301.6, 209, 45], [-301.6, 210, 44], [-301.6, 211, 43], [-301.6, 212, 43], [-301.6, 213, 44], [-301.6, 214, 45]])list([[-296.2, 209, 43], [-296.2, 209, 44], [-296.2, 210, 45], [-296.2, 211, 46], [-296.2, 212, 47], [-296.2, 212, 48], [-296.2, 213, 48], [-296.2, 214, 48], [-296.2, 215, 47], [-296.2, 215, 46], [-296.2, 215, 45], [-296.2, 214, 44], [-296.2, 213, 43], [-296.2, 212, 43], [-296.2, 211, 43], [-296.2, 210, 43], [-296.2, 209, 43], [-298.0, 208, 42], [-298.0, 208, 43], [-298.0, 208, 44], [-298.0, 208, 45], [-298.0, 208, 46], [-298.0, 208, 47], [-298.0, 209, 47], [-298.0, 210, 48], [-298.0, 211, 48], [-298.0, 211, 49], [-298.0, 212, 49], [-298.0, 213, 48], [-298.0, 214, 48], [-298.0, 215, 47], [-298.0, 216, 46], [-298.0, 216, 45], [-298.0, 216, 44], [-298.0, 215, 43], [-298.0, 214, 43], [-298.0, 213, 42], [-298.0, 212, 42], [-298.0, 212, 41], [-298.0, 211, 41], [-298.0, 210, 41], [-298.0, 209, 42], [-298.0, 208, 42], [-299.8, 210, 43], [-299.8, 209, 43], [-299.8, 208, 44], [-299.8, 207, 44], [-299.8, 207, 45], [-299.8, 207, 46], [-299.8, 208, 47], [-299.8, 209, 48], [-299.8, 210, 49], [-299.8, 211, 49], [-299.8, 212, 49], [-299.8, 213, 50], [-299.8, 214, 49], [-299.8, 215, 48], [-299.8, 215, 47], [-299.8, 216, 46], [-299.8, 216, 45], [-299.8, 215, 44], [-299.8, 215, 43], [-299.8, 214, 43], [-299.8, 214, 42], [-299.8, 213, 42], [-299.8, 212, 41], [-299.8, 211, 41], [-299.8, 210, 42], [-299.8, 210, 43]])] <class 'numpy.ndarray'>
二、标记次数和mask数组生成
生成npy文件并不是此次标注信息的最终结果,有以下几个原因:
xml文件内标注的结节坐标是多个医生分别标注的,所以会存在标注上的重叠(也就是一个结节被多个医生重复标注,很多是背靠背标注,也不知道其他医生标注了什么)。所以需要对多人标注的内容进行处理,留下最终的结节坐标;- 只是坐标点,还需要生成和
image一样shape,相互对应的mask文件。
根据上面几个原因,生成最终mask文件,就需要经历以下几个步骤:
- 标记的结节坐标点,需要将
hu z到instanceNum处理,对应的图像上; - 对多个医生标注的结节,进行处理,根据
iou重叠规则,留下最终的结节; - 留下的结节坐标,绘制到
mask上,存储下来。
实现代码如下:
import nrrd
import SimpleITK as sitk
import cv2
import os
import numpy as npdef load_itk_image(filename):"""Return img array and [z,y,x]-ordered origin and spacing"""# sitk.ReadImage返回的image的shape是x、y、zitkimage = sitk.ReadImage(filename)numpyImage = sitk.GetArrayFromImage(itkimage)numpyOrigin = np.array(list(reversed(itkimage.GetOrigin())))numpySpacing = np.array(list(reversed(itkimage.GetSpacing())))return numpyImage, numpyOrigin, numpySpacingdef arrs2mask(img_dir, ctr_arr_dir, save_dir):cnt = 0consensus = {1: 0, 2: 0, 3: 0, 4: 0} # 一致意见# generate save documentfor k in consensus.keys():if not os.path.exists(os.path.join(save_dir, str(k))):os.makedirs(os.path.join(save_dir, str(k)))for f in os.listdir(img_dir):if f.endswith('.mhd'):pid = f[:-4]print('pid:', pid)# ctimg, origin, spacing = load_itk_image(os.path.join(img_dir, '%s.mhd' % (pid)))# mask coor npyctr_arrs = np.load(os.path.join(ctr_arr_dir, '%s.npy' % (pid)), allow_pickle=True)cnt += len(ctr_arrs)nodule_masks = []# 依次标注结节处理for ctr_arr in ctr_arrs:z_origin = origin[0]z_spacing = spacing[0]ctr_arr = np.array(ctr_arr)# ctr_arr[:, 0] z轴方向值,由hu z到instanceNum [-50, -40, -30]-->[2, 3, 4]ctr_arr[:, 0] = np.absolute(ctr_arr[:, 0] - z_origin) / z_spacing # 对数组中的每一个元素求其绝对值。np.abs是这个函数的简写ctr_arr = ctr_arr.astype(np.int32)print(ctr_arr)# 每一个标注的结节,都会新临时生成一个与img一样大小的mask文件mask = np.zeros(img.shape)# 遍历标注层的 z 轴序列for z in np.unique(ctr_arr[:, 0]): # 去除其中重复的元素 ,并按元素 由小到大排序ctr = ctr_arr[ctr_arr[:, 0] == z][:, [2, 1]]ctr = np.array([ctr], dtype=np.int32)mask[z] = cv2.fillPoly(mask[z], ctr, color=(1,))nodule_masks.append(mask)i = 0visited = []d = {}masks = []while i < len(nodule_masks):# If mached before, then no need to create new maskif i in visited:i += 1continuesame_nodules = []mask1 = nodule_masks[i]same_nodules.append(mask1)d[i] = {}d[i]['count'] = 1d[i]['iou'] = []# Find annotations pointing to the same nodule# 当前结节mask[i],与其后面的所有结节,依次求ioufor j in range(i + 1, len(nodule_masks)):# if not overlapped with previous added nodulesif j in visited:continuemask2 = nodule_masks[j]iou = float(np.logical_and(mask1, mask2).sum()) / np.logical_or(mask1, mask2).sum()# 如果iou超过阈值,则当前第i个mask记为被重复标记一次if iou > 0.4:visited.append(j)same_nodules.append(mask2)d[i]['count'] += 1d[i]['iou'].append(iou)masks.append(same_nodules)i += 1print(visited)exit()# only 4 people, check up 4 datafor k, v in d.items():if v['count'] > 4:print('WARNING: %s: %dth nodule, iou: %s' % (pid, k, str(v['iou'])))v['count'] = 4consensus[v['count']] += 1# number of consensusnum = np.array([len(m) for m in masks])num[num > 4] = 4 # 最多4次,超过4次重复标记的,记为4次if len(num) == 0:continue# Iterate from the nodules with most consensusfor n in range(num.max(), 0, -1):mask = np.zeros(img.shape, dtype=np.uint8)for i, index in enumerate(np.where(num >= n)[0]):same_nodules = masks[index]m = np.logical_or.reduce(same_nodules)mask[m] = i + 1 # 区分不同的结节,不同的结节给与不同的数值,依次增加(如果是分割,可以直接都给1,或者最后统一处理为1也可以)nrrd.write(os.path.join(save_dir, str(n), pid+'.nrrd'), mask) # maskprint(consensus)print(cnt)if __name__ == '__main__':img_dir = r'./LUNA16/image_combined' # datactr_arr_save_dir = r'./LUNA16/annotation/noduleCoor' # 保存每个注释器解析的中间结节mask的地方noduleMask_save_dir = r'./LUNA16/nodule_masks' # 保存合并结节掩码的文件夹# 对转储的临时文件,生成maskarrs2mask(img_dir, ctr_arr_save_dir, noduleMask_save_dir)
至此,和image一样shape的mask是生成了。下面用itk-snap打开查看处理后的结果,如下所示:

属于分别打开image和mask的nrrd图像,mhd格式的image,转nrrd,可以参考下面的代码:
nii_path = os.path.join(r'./LUNA16/image_combined', '1.3.6.1.4.1.14519.5.2.1.6279.6001.184412674007117333405073397832.mhd')
image = itk.array_from_image(itk.imread(nii_path))nrrd.write(r'./image.nrrd', image)
三、总结
lidc-idri的数据集内的数据格式,都是我们不常遇到的数据形式,尤其是mhd文件的raw文件,同时表示一个数据的两个不同部分,也是很少遇到的。
但是对于初学者来说,理解这种数据形式,还是有些陌生,这部分相信通过本系列可以有较深的理解。与此同时,本篇还存储为nrrd文件,这是我比较喜欢的数组存储格式,理解的好理解和简单。
到这里,你就收获了一个新的一一对应关系。这样比你看xml文件,理解起来会简单很多。下一节,我们就对初步得到的image和mask,与肺区分割结合,进一步进行精细化处理。resample操作,调整数据到统一的尺度。
相关文章:

【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割6(数据预处理)
由于之前哔站作者整理的LUNA16数据处理方式过于的繁琐,于是,本文就对LUNA16数据做一个新的整理,最终得到的数据和形式是差不多的。但是,主要不同的是代码逻辑比较的简单,便于理解。 对于数据集的学习,可以…...
硬件加速器及其深度神经网络模型的性能指标理解
前言: 现如今,深度神经网络模型和硬件加速器,如GPU、TPU等的关系可谓是“不分彼此”,随着模型参数的增加,硬件加速器成为了训练、推理深度神经网络不可或缺的一个工具,而近年来硬件加速器的发展也得益于加速…...

嵌入式每日500(4)231104 (Flash类型定义、Flash常量定义、Flash函数)
文章目录 1.Flash类型定义(两个结构体)2.Flash常量定义(3种)3.Flash函数(31个,FLASH分为两个区,一个是普通的存储空间,一个是选项字节OB,函数名里带OB的就是对选项字节空…...

21款奔驰GLC300L升级23P驾驶辅助 出行更加的安全
驾驶辅助和自动驾驶的区别就是需要人为去接管,虽然车辆会根据道路自己行驶,弯道上也能居中自动修正行驶,长时间不接管方向盘,系统会提示人为接管,这就是奔驰的23P驾驶辅助系统, 很多车友升级23P驾驶辅助系…...
【小黑嵌入式系统第七课】PSoC® 5LP 开发套件(CY8CKIT-050B )——PSoC® 5LP主芯片、I/O系统、GPIO控制LED流水灯的实现
上一课: 【小黑嵌入式系统第六课】嵌入式系统软件设计基础——C语言简述、程序涉及规范、多任务程序设计、状态机建模(FSM)、模块化设计、事件触发、时间触发 文章目录 一、PSoC 5LP主芯片二、PSoC 5LP I/O系统(1) I/O系统特性(2) I/O系统怎样运作?1、I/…...

深度学习简史
一、说明 为了学习好深度学习,有必要知道深度学习的整个成长历史,可以说,深度学习的发展历程伴随整个人工智能的成长历程,本文将介绍这个历程的关键性历史节点。 二、深度学习的旅程 2.1 深度学习的当前认知 深度学习是机器学习的…...

CSRF 和 XSS 是什么
在Web开发中,安全性是至关重要的。然而,随着网络攻击技术的不断演进,跨站请求伪造(CSRF)和跨站脚本攻击(XSS)成为了最常见和具有破坏力的网络安全威胁之一。本文将介绍CSRF和XSS的概念、原理以及…...

亚信科技发布“电信级”核心交易数据库AntDB7.0,助力政企“信”创未来!
昨日,亚信科技AntDB数据库 7.0产品线上发布会成功举办,数千位关注亚信科技、关注国产数据库,致力于推动数据库行业变革的专家、客户热情参与,并对发布会及产品给予高度评价。 新增两大技术特性 作为我国最早一批独立研发的通用型…...
硬件调试-电源纹波测量
硬件调试-电源纹波测量 Fang XS.1452512966qq.com如果有错误,希望被指出,学习技术的路难免会磕磕绊绊量的积累引起质的变化感谢酸奶大佬提供的硬件技术指导; 电源纹波 百度百科定义如下: 纹波是由于直流稳定电源的电压波动而造…...

【洛谷算法题】P5710-数的性质【入门2分支结构】
👨💻博客主页:花无缺 欢迎 点赞👍 收藏⭐ 留言📝 加关注✅! 本文由 花无缺 原创 收录于专栏 【洛谷算法题】 文章目录 【洛谷算法题】P5710-数的性质【入门2分支结构】🌏题目描述🌏输入格式&a…...
arcgis图上添加发光效果!
看完本文, 你可以不借助外部图片素材, 让你的图纸符号表达出你想要的光! 我们以之前的某个项目图纸为例,来介绍下让符号发光的技术! 第一步—底图整理 准备好栅格影像底图、行政边界的矢量数据,确保“数据合适、位置正确、边界吻合”。 确定好图纸的大小、出图比例、投…...

[MySQL]——SQL预编译、动态sql
键盘敲烂,年薪30万🌈 目录 一、SQL的预编译 📕一条SQL语句的执行过程 📕弊端 📕预编译SQL的优势 📕两种参数占位符 📕小结 二、动态SQL 📕概念介绍: Ǵ…...

安装ifconfig命令(两步搞定)
如果你输入ifconfig显示没有这条命令,最可能的原因就是你在安装CentOS,先择的是最小安装模式,默认不安装ifconfig等命令, 解决办法:1. yum安装ifconfig这个命令包。命令:yum search ifconfig 2.通过yum …...

【蓝桥杯 第十届省赛Java B组】真题训练(A - H)H待更新
目录 A、组队 - 看图一眼出答案 B、不同子串 - 字符串模拟 set去重 C、数列求值 - 模拟取余 D、数的分解 - 三重暴力 E、迷宫 - bfs 判断路径 F、特别数的和 - 弱智模拟 G、外卖店优先级 - map 暴力(90%通过率) H、人物相关性分析 - A、组队 -…...

【牛客题】二进制求和 <模拟>
给定两个用字符串表示的二进制数,返回他们的和。 数据范围:字符串长度满足 1 ≤ n ≤ 1 0 5 10^5 105 ,字符串中只含有 0 和 1,且保证除 0 以外的二进制数没有前导零的情况。 示例输入: "101","1&qu…...

Error:Only idle or expired IP address can be disabled.
华为数通设备,在配置DHCP排除地址时可能会出现以下情况 Error:Only idle or expired IP address can be disabled. 这是因为地址已经被分配出去,所以现在无法进行排除 解决方法 1.先进入接口关闭DHCP [CE2-GigabitEthernet0/0/1]undo dhcp select g…...

Xubuntu16.04系统中create_ap开启5G网络的踩坑记录
Xubuntu16.04系统中安装create_ap创建无线AP: https://blog.csdn.net/qq_45445740/article/details/133972642?spm1001.2014.3001.5501 目录 1.create_ap.conf 配置文件解析2.关于信号强度和延时2.1 信号强度2.2 信号延时2.3 网络延时测试工具推荐——PingPlotter …...
8. 一文快速学懂常用工具——Linux命令(上)
本章讲解知识点 引言 指令学习 本专栏适合于软件开发刚入职的学生或人士,有一定的编程基础,帮助大家快速掌握工作中必会的工具和指令。本专栏针对面试题答案进行了优化,尽量做到好记、言简意赅。如专栏内容有错漏,欢迎在评论区指…...

@RestController注解说明
在Spring框架中,RestController注解是一个非常重要的注解,它用于将一个类标记为RESTful风格的控制器。本文将详细介绍RestController注解的作用和用法,并提供示例以帮助读者更好地理解和使用它。 RestController的作用 RestController注解是…...

Excel中行列范围的转换
将 行:1,4-5,8,11 列:a,c-e,f 这种写法转换成单元格地址的方法。 public static Tuple<List<int>, List<string>> ConvertRowColumn(string rowRep, string colRep){List<int> rowIdxs new List<int>();rowRep rowRep.…...

Docker 离线安装指南
参考文章 1、确认操作系统类型及内核版本 Docker依赖于Linux内核的一些特性,不同版本的Docker对内核版本有不同要求。例如,Docker 17.06及之后的版本通常需要Linux内核3.10及以上版本,Docker17.09及更高版本对应Linux内核4.9.x及更高版本。…...
Linux 文件类型,目录与路径,文件与目录管理
文件类型 后面的字符表示文件类型标志 普通文件:-(纯文本文件,二进制文件,数据格式文件) 如文本文件、图片、程序文件等。 目录文件:d(directory) 用来存放其他文件或子目录。 设备…...
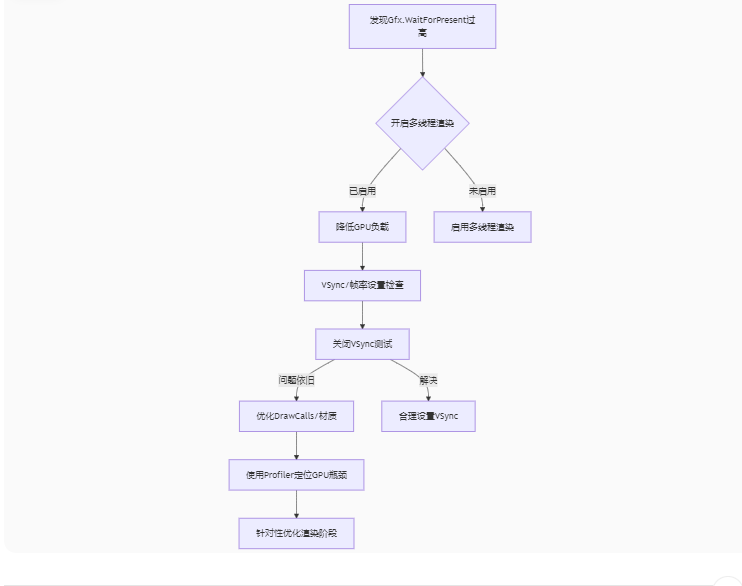
Unity3D中Gfx.WaitForPresent优化方案
前言 在Unity中,Gfx.WaitForPresent占用CPU过高通常表示主线程在等待GPU完成渲染(即CPU被阻塞),这表明存在GPU瓶颈或垂直同步/帧率设置问题。以下是系统的优化方案: 对惹,这里有一个游戏开发交流小组&…...

SciencePlots——绘制论文中的图片
文章目录 安装一、风格二、1 资源 安装 # 安装最新版 pip install githttps://github.com/garrettj403/SciencePlots.git# 安装稳定版 pip install SciencePlots一、风格 简单好用的深度学习论文绘图专用工具包–Science Plot 二、 1 资源 论文绘图神器来了:一行…...

AtCoder 第409场初级竞赛 A~E题解
A Conflict 【题目链接】 原题链接:A - Conflict 【考点】 枚举 【题目大意】 找到是否有两人都想要的物品。 【解析】 遍历两端字符串,只有在同时为 o 时输出 Yes 并结束程序,否则输出 No。 【难度】 GESP三级 【代码参考】 #i…...
渗透实战PortSwigger靶场-XSS Lab 14:大多数标签和属性被阻止
<script>标签被拦截 我们需要把全部可用的 tag 和 event 进行暴力破解 XSS cheat sheet: https://portswigger.net/web-security/cross-site-scripting/cheat-sheet 通过爆破发现body可以用 再把全部 events 放进去爆破 这些 event 全部可用 <body onres…...

Qwen3-Embedding-0.6B深度解析:多语言语义检索的轻量级利器
第一章 引言:语义表示的新时代挑战与Qwen3的破局之路 1.1 文本嵌入的核心价值与技术演进 在人工智能领域,文本嵌入技术如同连接自然语言与机器理解的“神经突触”——它将人类语言转化为计算机可计算的语义向量,支撑着搜索引擎、推荐系统、…...
P3 QT项目----记事本(3.8)
3.8 记事本项目总结 项目源码 1.main.cpp #include "widget.h" #include <QApplication> int main(int argc, char *argv[]) {QApplication a(argc, argv);Widget w;w.show();return a.exec(); } 2.widget.cpp #include "widget.h" #include &q…...

智能仓储的未来:自动化、AI与数据分析如何重塑物流中心
当仓库学会“思考”,物流的终极形态正在诞生 想象这样的场景: 凌晨3点,某物流中心灯火通明却空无一人。AGV机器人集群根据实时订单动态规划路径;AI视觉系统在0.1秒内扫描包裹信息;数字孪生平台正模拟次日峰值流量压力…...

蓝桥杯3498 01串的熵
问题描述 对于一个长度为 23333333的 01 串, 如果其信息熵为 11625907.5798, 且 0 出现次数比 1 少, 那么这个 01 串中 0 出现了多少次? #include<iostream> #include<cmath> using namespace std;int n 23333333;int main() {//枚举 0 出现的次数//因…...
