PSP - 从头搭建 抗原类别 (GPCR) 的 蛋白质结构预测 项目流程
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/134595717
GPCRs(G Protein-Coupled Receptors,G蛋白偶联受体),又称为7次跨膜受体,是细胞信号传导中的重要蛋白质,当膜外的配体作用于该受体时,该受体的膜内部分与G蛋白相互结合激活G蛋白,进而启动不同的信号转导通路,参与多种生理和病理过程,如免疫调节、行为和情绪的调节、感觉的传递、内稳态的调节以及肿瘤的生长与转移等。G 蛋白偶联受体(GPCR)超家族包含约 600-1000个靶标,是已知最大的一类具有治疗价值的分子靶标,目前世界药物市场上至少有三分之一的小分子药物是GPCR的激动剂或拮抗剂。GPCR过表达细胞系已广泛用于药物筛选和自身免疫性疾病的病因和发病机制研究。
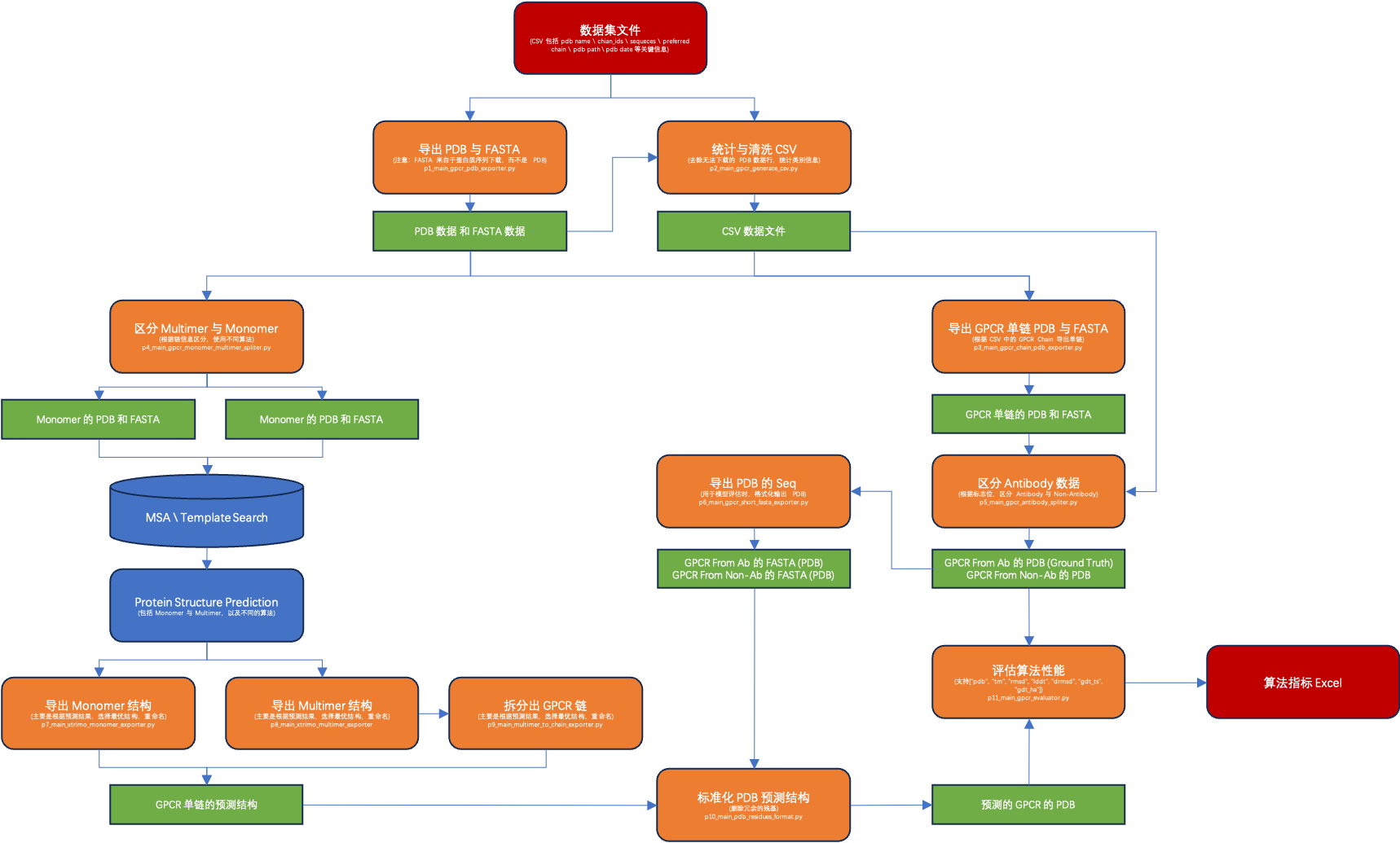
预测流程框架如下:
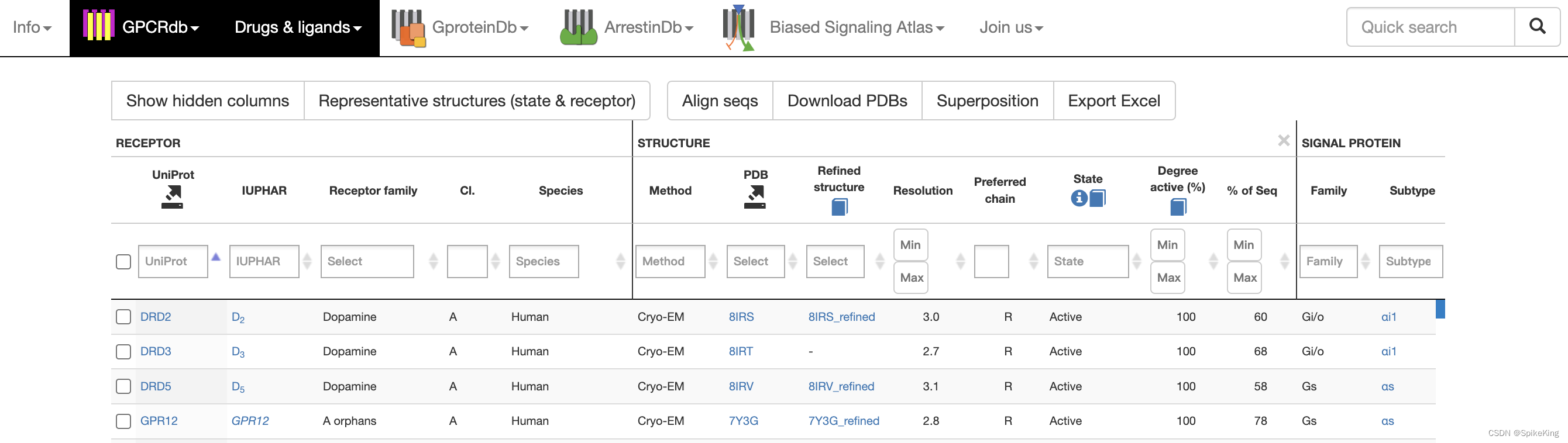
1. GPCR 数据集
GPCR 数据,来自于 GPCR-DB 官网,其中包括 PDB、GPCR chain id、PDB Date 等信息,再从 PDB 中获取 FASTA Sequence 信息,即:
PDB 的 FASTA Sequence,来自于 RCSB 官网,如下:
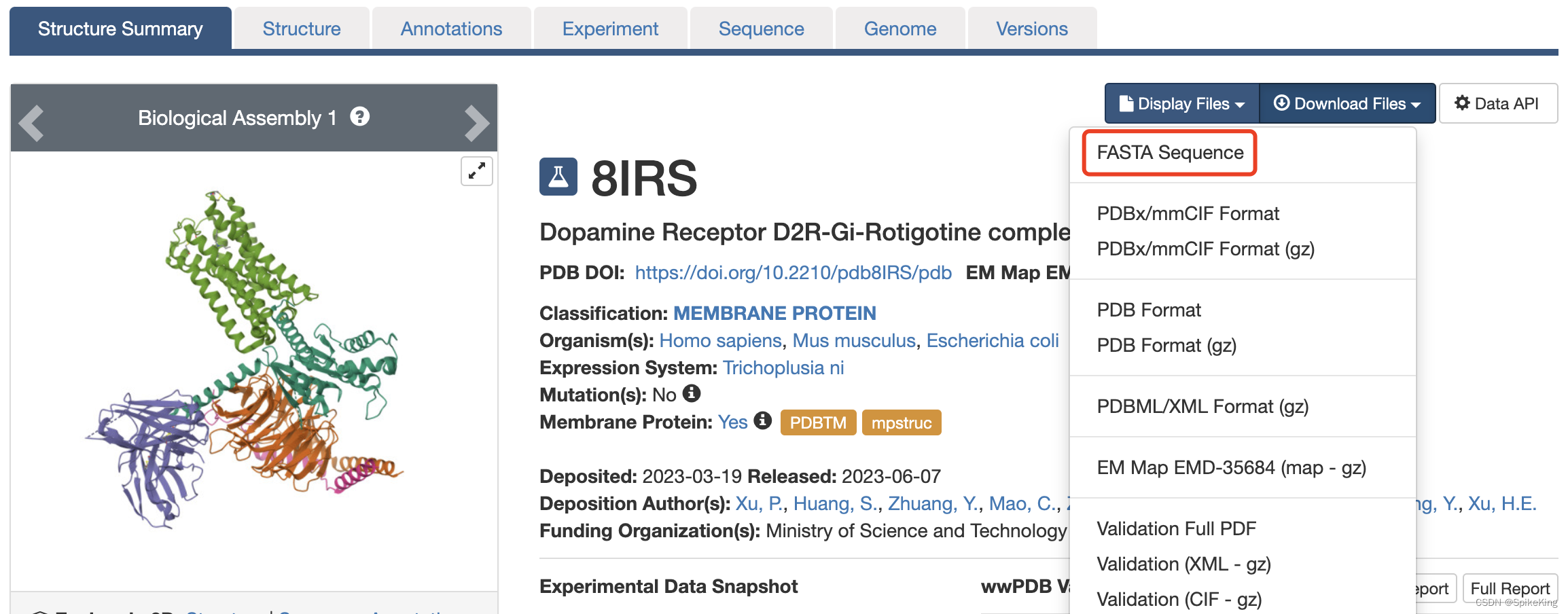
8IRS 的 FASTA 序列,如下:
>8IRS_1|Chain A|Guanine nucleotide-binding protein G(i) subunit alpha-1|Homo sapiens (9606)
MGCTLSAEDKAAVERSKMIDRNLREDGEKAAREVKLLLLGAGESGKNTIVKQMKIIHEAGYSEEECKQYKAVVYSNTIQSIIAIIRAMGRLKIDFGDSARADDARQLFVLAGAAEEGFMTAELAGVIKRLWKDSGVQACFNRSREYQLNDSAAYYLNDLDRIAQPNYIPTQQDVLRTRVKTTGIVETHFTFKDLHFKMFDVGAQRSERKKWIHCFEGVTAIIFCVALSDYDLVLAEDEEMNRMHASMKLFDSICNNKWFTDTSIILFLNKKDLFEEKIKKSPLTICYPEYAGSNTYEEAAAYIQCQFEDLNKRKDTKEIYTHFTCSTDTKNVQFVFDAVTDVIIKNNLKDCGLF
>8IRS_2|Chain B|Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1|Homo sapiens (9606)
MGSLLQSELDQLRQEAEQLKNQIRDARKACADATLSQITNNIDPVGRIQMRTRRTLRGHLAKIYAMHWGTDSRLLVSASQDGKLIIWDSYTTNKVHAIPLRSSWVMTCAYAPSGNYVACGGLDNICSIYNLKTREGNVRVSRELAGHTGYLSCCRFLDDNQIVTSSGDTTCALWDIETGQQTTTFTGHTGDVMSLSLAPDTRLFVSGACDASAKLWDVREGMCRQTFTGHESDINAICFFPNGNAFATGSDDATCRLFDLRADQELMTYSHDNIICGITSVSFSKSGRLLLAGYDDFNCNVWDALKADRAGVLAGHDNRVSCLGVTDDGMAVATGSWDSFLKIWN
>8IRS_3|Chain C[auth E]|ScFv16|Mus musculus (10090)
DVQLVESGGGLVQPGGSRKLSCSASGFAFSSFGMHWVRQAPEKGLEWVAYISSGSGTIYYADTVKGRFTISRDDPKNTLFLQMTSLRSEDTAMYYCVRSIYYYGSSPFDFWGQGTTLTVSSGGGGSGGGGSGGGGSDIVMTQATSSVPVTPGESVSISCRSSKSLLHSNGNTYLYWFLQRPGQSPQLLIYRMSNLASGVPDRFSGSGSGTAFTLTISRLEAEDVGVYYCMQHLEYPLTFGAGTKLELK
>8IRS_4|Chain D[auth G]|Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2|Homo sapiens (9606)
MASNNTASIAQARKLVEQLKMEANIDRIKVSKAAADLMAYCEAHAKEDPLLTPVPASENPFREKKFFCAIL
>8IRS_5|Chain E[auth R]|Soluble cytochrome b562,D(2) dopamine receptor|Escherichia coli (562)
DYKDDDDAKLQTMHHHHHHHHHHHHHHHADLEDNWETLNDNLKVIEKADNAAQVKDALTKMRAAALDAQKATPPKLEDKSPDSPEMKDFRHGFDILVGQIDDALKLANEGKVKEAQAAAEQLKTTRNAYIQKYLASENLYFQGGTMDPLNLSWYDDDLERQNWSRPFNGSDGKADRPHYNYYATLLTLLIAVIVFGNVLVCMAVSREKALQTTTNYLIVSLAVADLLVATLVMPWVVYLEVVGEWKFSRIHCDIFVTLDVMMCTASILNLCAISIDRYTAVAMPMLYNTRYSSKRRVTVMISIVWVLSFTISCPLLFGLNNADQNECIIANPAFVVYSSIVSFYVPFIVTLLVYIKIYIVLRRRRKRVNTKRSSRAFRAHLRAPLKGNCTHPEDMKLCTVIMKSNGSFPVNRRRVEAARRAQELEMEMLSSTSPPERTRYSPIPPSHHQLTLPDPSHHGLHSTPDSPAKPEKNGHAKDHPKIAKIFEIQTMPNGKTRTSLKTMSRRKLSQQKEKKATQMLAIVLGVFIICWLPFFITHILNIHCDCNIPPVLYSAFTWLGYVNSAVNPIIYTTFNIEFRKAFLKILHC
存储的 8irs 序列,即:
pdb,chain,seq,mol,gpcr_chain
8irs,"R,G,E,B,A","DYKDDDDAKLQTMHHHHHHHHHHHHHHHADLEDNWETLNDNLKVIEKADNAAQVKDALTKMRAAALDAQKATPPKLEDKSPDSPEMKDFRHGFDILVGQIDDALKLANEGKVKEAQAAAEQLKTTRNAYIQKYLASENLYFQGGTMDPLNLSWYDDDLERQNWSRPFNGSDGKADRPHYNYYATLLTLLIAVIVFGNVLVCMAVSREKALQTTTNYLIVSLAVADLLVATLVMPWVVYLEVVGEWKFSRIHCDIFVTLDVMMCTASILNLCAISIDRYTAVAMPMLYNTRYSSKRRVTVMISIVWVLSFTISCPLLFGLNNADQNECIIANPAFVVYSSIVSFYVPFIVTLLVYIKIYIVLRRRRKRVNTKRSSRAFRAHLRAPLKGNCTHPEDMKLCTVIMKSNGSFPVNRRRVEAARRAQELEMEMLSSTSPPERTRYSPIPPSHHQLTLPDPSHHGLHSTPDSPAKPEKNGHAKDHPKIAKIFEIQTMPNGKTRTSLKTMSRRKLSQQKEKKATQMLAIVLGVFIICWLPFFITHILNIHCDCNIPPVLYSAFTWLGYVNSAVNPIIYTTFNIEFRKAFLKILHC,MASNNTASIAQARKLVEQLKMEANIDRIKVSKAAADLMAYCEAHAKEDPLLTPVPASENPFREKKFFCAIL,DVQLVESGGGLVQPGGSRKLSCSASGFAFSSFGMHWVRQAPEKGLEWVAYISSGSGTIYYADTVKGRFTISRDDPKNTLFLQMTSLRSEDTAMYYCVRSIYYYGSSPFDFWGQGTTLTVSSGGGGSGGGGSGGGGSDIVMTQATSSVPVTPGESVSISCRSSKSLLHSNGNTYLYWFLQRPGQSPQLLIYRMSNLASGVPDRFSGSGSGTAFTLTISRLEAEDVGVYYCMQHLEYPLTFGAGTKLELK,MGSLLQSELDQLRQEAEQLKNQIRDARKACADATLSQITNNIDPVGRIQMRTRRTLRGHLAKIYAMHWGTDSRLLVSASQDGKLIIWDSYTTNKVHAIPLRSSWVMTCAYAPSGNYVACGGLDNICSIYNLKTREGNVRVSRELAGHTGYLSCCRFLDDNQIVTSSGDTTCALWDIETGQQTTTFTGHTGDVMSLSLAPDTRLFVSGACDASAKLWDVREGMCRQTFTGHESDINAICFFPNGNAFATGSDDATCRLFDLRADQELMTYSHDNIICGITSVSFSKSGRLLLAGYDDFNCNVWDALKADRAGVLAGHDNRVSCLGVTDDGMAVATGSWDSFLKIWN,MGCTLSAEDKAAVERSKMIDRNLREDGEKAAREVKLLLLGAGESGKNTIVKQMKIIHEAGYSEEECKQYKAVVYSNTIQSIIAIIRAMGRLKIDFGDSARADDARQLFVLAGAAEEGFMTAELAGVIKRLWKDSGVQACFNRSREYQLNDSAAYYLNDLDRIAQPNYIPTQQDVLRTRVKTTGIVETHFTFKDLHFKMFDVGAQRSERKKWIHCFEGVTAIIFCVALSDYDLVLAEDEEMNRMHASMKLFDSICNNKWFTDTSIILFLNKKDLFEEKIKKSPLTICYPEYAGSNTYEEAAAYIQCQFEDLNKRKDTKEIYTHFTCSTDTKNVQFVFDAVTDVIIKNNLKDCGLF",R
注意:Chain ID 优先使用 auth 值,其次使用默认的 Chain 值,例如 Chain D[auth G] 是 G,Chain A 是 A。
获取 CSV 文件 gpcr_dataset.csv,字段包括:pdb、chain、seq、gpcr_chain 等关键字段。
2. 导出 PDB 与 FASTA 数据
根据数据文件,导出 GPCR 相关的复合物 PDB 与 多链 FASTA,再根据 GPCR 链 (Preferred Chain) 导出单体 PDB 与 单链 FASTA。
2.1 导出 PDB 复合物与蛋白质序列
调用 p1_main_gpcr_pdb_exporter.py,提取 GPCR 复合物结构与序列。
python3 gpcr/p1_main_gpcr_pdb_exporter.py \
-i gpcr/gpcr_data_filter.csv \
-p gpcr/gpcr_complex_v2/pdb_complex_446 \
-f gpcr/gpcr_complex_v2/fasta_complex_446
存储的 PDB 的格式与 FASTA 的格式,建议保留 PDB 名称、链名、链长、全部序列长度,即:
- PDB:
8IW9_A234_B338_C53_R309_S231_N128_1293.pdb - FASTA:
8IW9_A362_B377_C59_N128_R348_S285_1559.fasta
注意:全长的 FASTA 序列预测 PDB 结构,优于从 PDB 中提取的 FASTA 序列预测的结构,原因是 PDB 结构中缺失一些残基位置。
GPCR 复合物的输出位置,包括 PDB 与 FASTA,即:
gpcr/gpcr_complex_v2/pdb_complex_446/
gpcr/gpcr_complex_v2/fasta_complex_446/
8IW9_A362_B377_C59_N128_R348_S285_1559.fasta 序列:
>A
MMGCTLSAEDKAAVERSKMIEKQLQKDKQVYRATHRLLLLGADNSGKSTIVKQMRIYHVNGYSEEECKQYKAVVYSNTIQSIIAIIRAMGRLKIDFGDSARADDARQLFVLAGAAEEGFMTAELAGVIKRLWKDSGVQACFNRSREYQLNDSAAYYLNDLDRIAQPNYIPTQQDVLRTRVKTSGIFETKFQVDKVNFHMFDVGAQRDERRKWIQCFNDVTAIIFVVDSSDYNRLQEALNDFKSIWNNRWLRTISVILFLNKQDLLAEKVLAGKSKIEDYFPEFARYTTPEDATPEPGEDPRVTRAKYFIRDEFLRISTASGDGRHYCYPHFTCSVDTENARRIFNDCRDIIQRMHLRQYELL
>B
MHHHHHHGSLLQSELDQLRQEAEQLKNQIRDARKACADATLSQITNNIDPVGRIQMRTRRTLRGHLAKIYAMHWGTDSRLLVSASQDGKLIIWDSYTTNKVHAIPLRSSWVMTCAYAPSGNYVACGGLDNICSIYNLKTREGNVRVSRELAGHTGYLSCCRFLDDNQIVTSSGDTTCALWDIETGQQTTTFTGHTGDVMSLSLAPDTRLFVSGACDASAKLWDVREGMCRQTFTGHESDINAICFFPNGNAFATGSDDATCRLFDLRADQELMTYSHDNIICGITSVSFSKSGRLLLAGYDDFNCNVWDALKADRAGVLAGHDNRVSCLGVTDDGMAVATGSWDSFLKIWNGSSGGGGSGGGGSSGVSGWRLFKKIS
>C
NTASIAQARKLVEQLKMEANIDRIKVSKAAADLMAYCEAHAKEDPLLTPVPASENPFRE
>N
QVQLQESGGGLVQPGGSLRLSCAASGFTFSNYKMNWVRQAPGKGLEWVSDISQSGASISYTGSVKGRFTISRDNAKNTLYLQMNSLKPEDTAVYYCARCPAPFTRDCFDVTSTTYAYRGQGTQVTVSS
>R
MTSDFSPEPPMELCYENVNGSCIKSSYAPWPRAILYGVLGLGALLAVFGNLLVIIAILHFKQLHTPTNFLVASLACADFLVGVTVMPFSTVRSVESCWYFGESYCKFHTCFDTSFCFASLFHLCCISIDRYIAVTDPLTYPTKFTVSVSGLCIALSWFFSVTYSFSIFYTGANEEGIEELVVALTCVGGCQAPLNQNWVLLCFLLFFLPTVVMVFLYGRIFLVAKYQARKIEGTANQAQASSESYKERVAKRERKAAKTLGIAMAAFLVSWLPYIIDAVIDAYMNFITPAYVYEILVWCVYYNSAMNPLIYAFFYPWFRKAIKLIVSGKVFRADSSTTNLFSEEAGAG
>S
MLLVNQSHQGFNKEHTSKMVSAIVLYVLLAAAAHSAFAVQLVESGGGLVQPGGSRKLSCSASGFAFSSFGMHWVRQAPEKGLEWVAYISSGSGTIYYADTVKGRFTISRDDPKNTLFLQMTSLRSEDTAMYYCVRSIYYYGSSPFDFWGQGTTLTVSAGGGGSGGGGSGGGGSADIVMTQATSSVPVTPGESVSISCRSSKSLLHSNGNTYLYWFLQRPGQSPQLLIYRMSNLASGVPDRFSGSGSGTAFTLTISRLEAEDVGVYYCMQHLEYPLTFGAGTKLEL
2.2 统计与清洗 CSV 文件
调用 p2_main_gpcr_generate_csv.py,根据 FASTA 文件,在原文件中,去除错误的 Case,保留现有的 Case,即:
python3 gpcr/p2_main_gpcr_generate_csv.py \
-i gpcr/gpcr_data_filter.csv \
-f gpcr/gpcr_complex_v2/fasta_complex_446 \
-o gpcr/gpcr_complex_v2/gpcr_info_446.csv
GPCR 复合物是否包含 抗体(antibody) 链的数据类型:
{'antibody': 340, 'no antibody': 106}
更新的 GPCR 信息文件:
pdb,ab,chains,gpcr,g_len,seqs
7XTC,True,"A,B,G,N,R",R,576,"MGCLGNSKTED..."
2.3 拆分 GPCR 单链的 PDB 与 FASTA
调用 p3_main_gpcr_chain_pdb_exporter.py,根据 GPCR 链拆分 PDB 与 FASTA,即:
python3 gpcr/p3_main_gpcr_chain_pdb_exporter.py \
-i gpcr/gpcr_complex_v2/gpcr_info_446.csv \
-f gpcr/gpcr_complex_v2/fasta_complex_446 \
-p gpcr/gpcr_complex_v2/pdb_complex_446 \
-of gpcr/gpcr_complex_v2/gcpr_chain/fasta \
-op gpcr/gpcr_complex_v2/gcpr_chain/pdb
在拆分 PDB 单链之后,使用格式化 PDB,即保证残基中 CA 只有1个,同时链 ID 转换成 A,有利于后续的结构评估。
输出的 GPCR 链,序列是全长,FASTA 长度大于 PDB 长度,如下:
8IW9_R309.pdb8IW9_R348.fasta
8IW9_R348.fasta 的序列,即:
>R
MTSDFSPEPPMELCYENVNGSCIKSSYAPWPRAILYGVLGLGALLAVFGNLLVIIAILHFKQLHTPTNFLVASLACADFLVGVTVMPFSTVRSVESCWYFGESYCKFHTCFDTSFCFASLFHLCCISIDRYIAVTDPLTYPTKFTVSVSGLCIALSWFFSVTYSFSIFYTGANEEGIEELVVALTCVGGCQAPLNQNWVLLCFLLFFLPTVVMVFLYGRIFLVAKYQARKIEGTANQAQASSESYKERVAKRERKAAKTLGIAMAAFLVSWLPYIIDAVIDAYMNFITPAYVYEILVWCVYYNSAMNPLIYAFFYPWFRKAIKLIVSGKVFRADSSTTNLFSEEAGAG
2.4 GPCR 数据集区分 Monomer 与 Multimer
调用 p4_main_gpcr_monomer_multimer_spliter.py,拆分成 Monomer 与 Multimer 的 FASTA:
python3 gpcr/p4_main_gpcr_monomer_multimer_spliter.py \
-i gpcr/gpcr_complex_v2/fasta_complex_446/ \
-mo gpcr/gpcr_complex_v2/fasta_monomer_36 \
-mu gpcr/gpcr_complex_v2/fasta_multimer_410
确保 Monomer 与 Multimer 的数量之和,等于之前的结构数量,即 36 + 410 = 446,输出如下:
- Monomer 的 FASTA 文件:
8I2H_A682.fasta - Multimer 的 FASTA 文件:
8IW9_A362_B377_C59_N128_R348_S285_1559.fasta
8I2H_A682.fasta 序列如下:
>A
LGSGCHHRICHCSNRVFLCQESKVTEIPSDLPRNAIELRFVLTKLRVIQKGAFSGFGDLEKIEISQNDVLEVIEADVFSNLPKLHEIRIEKANNLLYINPEAFQNLPNLQYLLISNTGIKHLPDVHKIHSLQKVLLDIQDNINIHTIERNSFVGLSFESVILWLNKNGIQEIHNCAFNGTQLDELNLSDNNNLEELPNDVFHGASGPVILDISRTRIHSLPSYGLENLKKLRARSTYNLKKLPTLEKLVALMEASLTYPSHCCAFANWRRQISELHPICNKSILRQEVDYMTQARGQRSSLAEDNESSYSRGFDMTYTEFDYDLCNEVVDVTCSPKPDAFNPCEDIMGYNILRVLIWFISILAITGNIIVLVILTTSQYKLTVPRFLMCNLAFADLCIGIYLLLIASVDIHTKSQYHNYAIDWQTGAGCDAAGFFTVFASELSVYTLTAITLERWHTITHAMQLDCKVQLRHAASVMVMGWIFAFAAALFPIFGISSYMKVSICLPMDIDSPLSQLYVMSLLVLNVLAFVVICGCYIHIYLTVRNPNIVSSSSDTRIAKRMAMLIFTDFLCMAPISFFAISASLKVPLITVSKAKILLVLFHPINSCANPFLYAIFTKNFRRDFFILLSKCGCYEMQAQIYRTETSSTVHNTHPRNGHCSSAPRVTNGSTYILVPLSHLAQN
拆分成 Monomer 与 Multimer 主要原因是蛋白质结构预测算法,区分 Monomer 与 Multimer,使用不同的模型与算法框架,进行预测。
2.5 GPCR 单链区分 Antibody
调用 p5_main_gpcr_antibody_spliter.py,通过标签文件,拆分出 Antibody 与 Non-Antibody 数据集的 PDB 与 FASTA 文件:
python3 gpcr/p5_main_gpcr_antibody_spliter.py \
-i gpcr/gpcr_complex_v2/gpcr_info_446.csv \
-r gpcr/gpcr_complex_v2/gcpr_chain/pdb_446 \
-oa gpcr/gpcr_complex_v2/gcpr_chain/pdb_ab \
-on gpcr/gpcr_complex_v2/gcpr_chain/pdb_nab \
-m pdb
日志:[Info] sample: 446, ab: 340, nab: 106
2.6 从 PDB 中导出已预测残基序列
调用 p6_main_gpcr_short_fasta_exporter.py,从 PDB 中提取 FASTA (短序列),用于后续评估:
python3 gpcr/p6_main_gpcr_short_fasta_exporter.py \
-i gpcr/gpcr_complex_v2/pdb_complex_446 \
-o gpcr/gpcr_complex_v2/fasta_from_pdb_446
PDB 中提取 FASTA 短序列的预测效果,低于真实的长序列,用于后续评估。
输出的 FASTA 序列长度 与 PDB 一致:
- FASTA:
8IW9_A234_B338_C53_R309_S231_N128_1293.fasta - PDB:
8IW9_A234_B338_C53_N128_R309_S231_1293.pdb
3. 搜索 MSA 序列与导出 PDB 结构
搜索 MSA 序列,预测 PDB 结构,导出 PDB 结构,从 Multimer 拆出单链,再根据 FASTA 格式化。
3.1 搜索 MSA
推理 AF2 的 MSA 脚本,需要区分 Monomer 与 Multimer,即:
nohup bash run_alphafold.sh -f gpcr/gpcr_complex_v2/fasta_multimer_410/ -o gpcr/gpcr_complex_v2/fasta_multimer_410_msas -m multimer -h true > nohup.fasta_multimer_410_msas.out &nohup bash run_alphafold.sh -f gpcr/gpcr_complex_v2/fasta_monomer_36/ -o gpcr/gpcr_complex_v2/fasta_monomer_36_msas -m monomer -h true > nohup.fasta_monomer_36_msas.out &nohup bash run_alphafold.sh -f gpcr/gpcr_complex_v2/gcpr_chain/fasta_446 -o gpcr/gpcr_complex_v2/gcpr_chain/fasta_446_msas -m monomer -h true > nohup.gcpr_chain_fasta_msas.out &
使用 MSA 服务推理序列,即:
python msa_main.py -m 0 -f gpcr/gpcr_complex_v2/fasta_multimer_410/ -r mydata/test_fasta_multimer_410.json
python msa_main.py -m 1 -r mydata/test_fasta_multimer_410.jsonpython msa_main.py -m 0 -f gpcr/gpcr_complex_v2/fasta_monomer_36/ -r mydata/test_fasta_monomer_36.json
python msa_main.py -m 1 -r mydata/test_fasta_monomer_36.jsonpython msa_main.py -m 0 -f gpcr/gpcr_complex_v2/gcpr_chain/fasta_446/ -r mydata/test_fasta_446.json
python msa_main.py -m 1 -r mydata/test_fasta_446.json
以及预测 Monomer 与 Multimer 的结构。
3.1 导出 Monomer 的 PDB 结构
调用 p7_main_xtrimo_monomer_exporter.py,导出预测的 Monomer PDB 结构至单个文件夹,用于后续评估:
python3 gpcr/main_xtrimo_monomer_exporter.py \
-i gpcr_protein_no_antibody/ \
-o mydata/gpcr_eval/baseline_nab_local \
-f gpcr/gpcr_complex/gcpr_chain/fasta_non_ab_95/ \
-r mydata/gpcr_eval/baseline_nab_local_format
3.3 导出 Multimer 的 PDB 结构
调用 p8_main_xtrimo_multimer_exporter.py,导出预测的 Multimer PDB 结构至单个文件夹,用于后续评估:
python3 gpcr/main_xtrimo_multimer_exporter.py \
-i mydata/outputs_infer/gpcr_fasta_multimer_392_outputs/ \
-o mydata/gpcr_eval/gpcr_fasta_multimer_392 \
-f gpcr/gpcr_complex/fasta_multimer_392/ \
-r mydata/gpcr_eval/gpcr_fasta_multimer_392_format
再调用 p9_main_multimer_to_chain_exporter.py,从 Multimer 中 导出 GPCR 单链结构,即:
python3 gpcr/p9_main_multimer_to_chain_exporter.py \
-i mydata/gpcr_eval/gpcr_fasta_multimer_392_format \
-o mydata/gpcr_eval/gpcr_chain_multimer_v1 \
-c gpcr/gpcr_complex_v2/gpcr_info_446.csv
Monomer 的单链与 Multimer 导出的单链,都需要根据 PDB 序列去除冗余残基,只保留与 PDB 一致的残基。
调用 p10_main_pdb_residues_format.py,预测 PDB 结构的序列,匹配目标 PDB 结构的序列,标准化格式,用于之后的单链评估,即:
python3 gpcr/p10_main_pdb_residues_format.py \
-i mydata/gpcr_eval/gpcr_chain_redundancy_multimer \
-o mydata/gpcr_eval/gpcr_chain_redundancy_multimer_format \
-f gpcr/gpcr_complex_v2/fasta_from_pdb_446
4. 评估模型性能
调用 p11_main_gpcr_evaluator.py 评估预测结果,支持 ["pdb", "tm", "rmsd", "lddt", "drmsd", "gdt_ts", "gdt_ha"]
- 输入文件夹
-m - 实验名称
-n - 真实结构文件夹
-t - 输出 xls 文件
-o
即:
python3 gpcr/p11_main_gpcr_evaluator.py \
-m mydata/gpcr_eval/gpcr_chain_redundancy_multimer_format \
-n nab_multimer_redundancy \
-t mydata/gpcr_targets/pdb_non_ab_95_format \
-o mydata/gpcr_res/
输出效果:
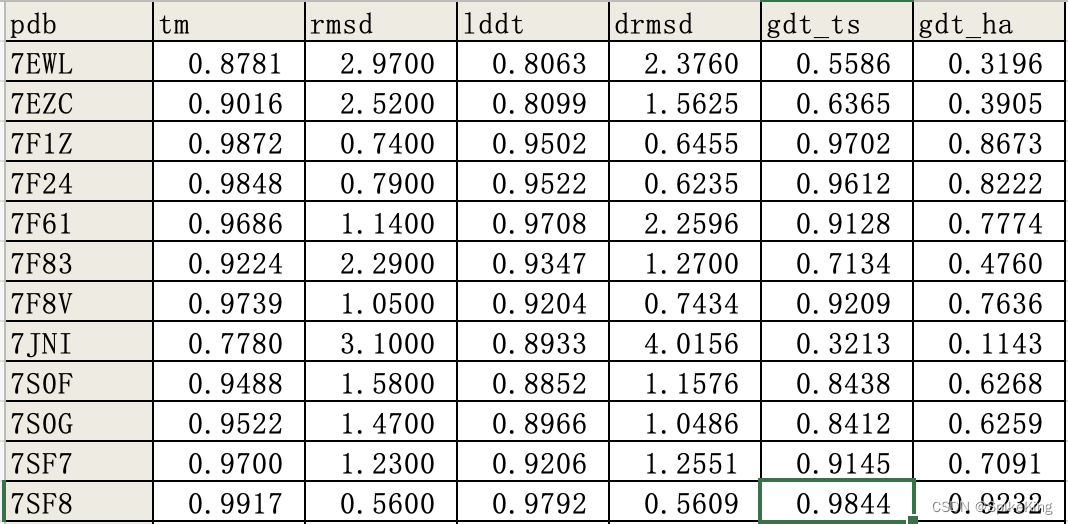
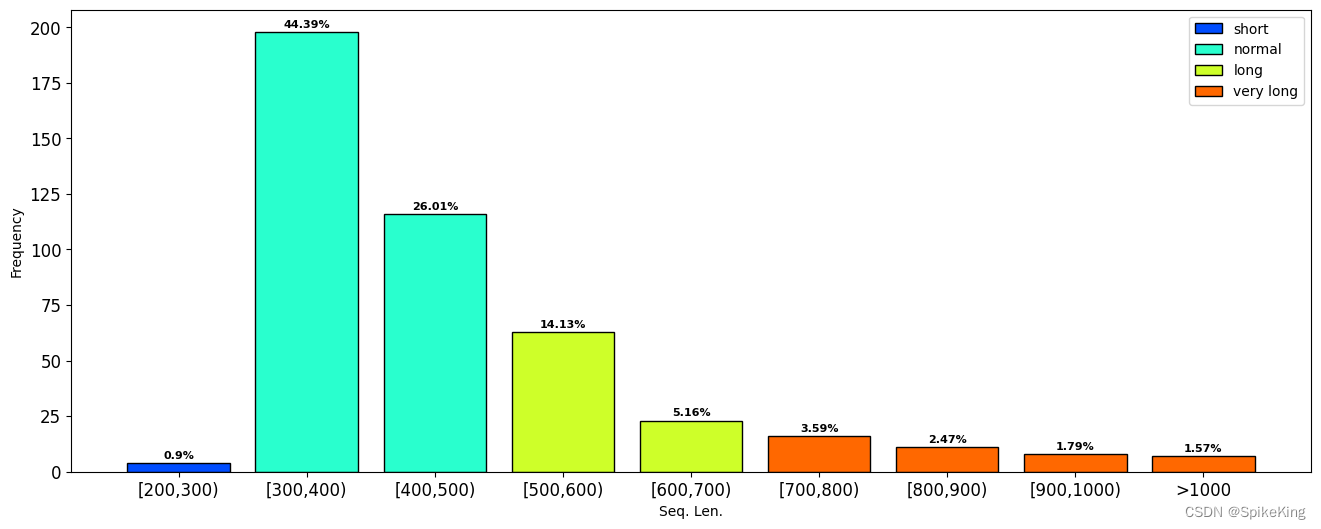
调用 p12_main_gpcr_info_analysis.py 统计信息,支持展示序列长度:
python3 gpcr/p12_main_gpcr_info_analysis.py \
-i gpcr/gpcr_complex_v2/gpcr_info_446.csv \
-o mydata/gpcr/gpcr_images \
-k g_len
日志:
[Info] seq len range: 283 ~ 1543
[Info] len > 20: 446, len < 20: 0
[Info] value_counts: 300: 198 (44.3946%), 400: 116 (26.009%), 500: 63 (14.1256%), 600: 23 (5.157%), 700: 16 (3.5874%), 800: 11 (2.4664%), 900: 8 (1.7937%), 1000: 7 (1.5695%), 200: 4 (0.8969%), sum: 446
效果:
相关文章:

PSP - 从头搭建 抗原类别 (GPCR) 的 蛋白质结构预测 项目流程
欢迎关注我的CSDN:https://spike.blog.csdn.net/ 本文地址:https://spike.blog.csdn.net/article/details/134595717 GPCRs(G Protein-Coupled Receptors,G蛋白偶联受体),又称为7次跨膜受体,是细…...

城市NOA加速落地,景联文科技高质量数据标注助力感知系统升级
当前,自动驾驶技术的演进正在经历着从基础L2到L3过渡的重要阶段,其中NOA(自动辅助导航驾驶)扮演着至关重要的角色。城市NOA(L2.9)作为城市场景下的NOA,被看作是车企向更高阶自动驾驶迈进的必经之…...
是什么?)
控制反转(IoC)是什么?
文章目录 控制反转(Inversion of Control,IoC)传统的程序设计中:应用程序控制程序流程控制反转设计中:由框架或容器控制程序流程IoC 的作用 举例生活例子软件工程例子 控制反转(Inversion of Control&#…...
Redisson分布式锁源码解析、集群环境存在的问题
一、使用Redisson步骤 Redisson各个锁基本所用Redisson各个锁基本所用Redisson各个锁基本所用 二、源码解析 lock锁 1) 基本思想: lock有两种方法 一种是空参 另一种是带参 * 空参方法:会默认调用看门狗的过期时间30*1000&…...

2016年10月4日 Go生态洞察:HTTP追踪介绍
🌷🍁 博主猫头虎(🐅🐾)带您 Go to New World✨🍁 🦄 博客首页——🐅🐾猫头虎的博客🎐 🐳 《面试题大全专栏》 🦕 文章图文…...

分布式篇---第四篇
系列文章目录 文章目录 系列文章目录前言一、分布式ID生成有几种方案?二、幂等解决方法有哪些?三、常见负载均衡算法有哪些?前言 前些天发现了一个巨牛的人工智能学习网站,通俗易懂,风趣幽默,忍不住分享一下给大家。点击跳转到网站,这篇文章男女通用,看懂了就去分享给…...
从零开始的C++(十九)
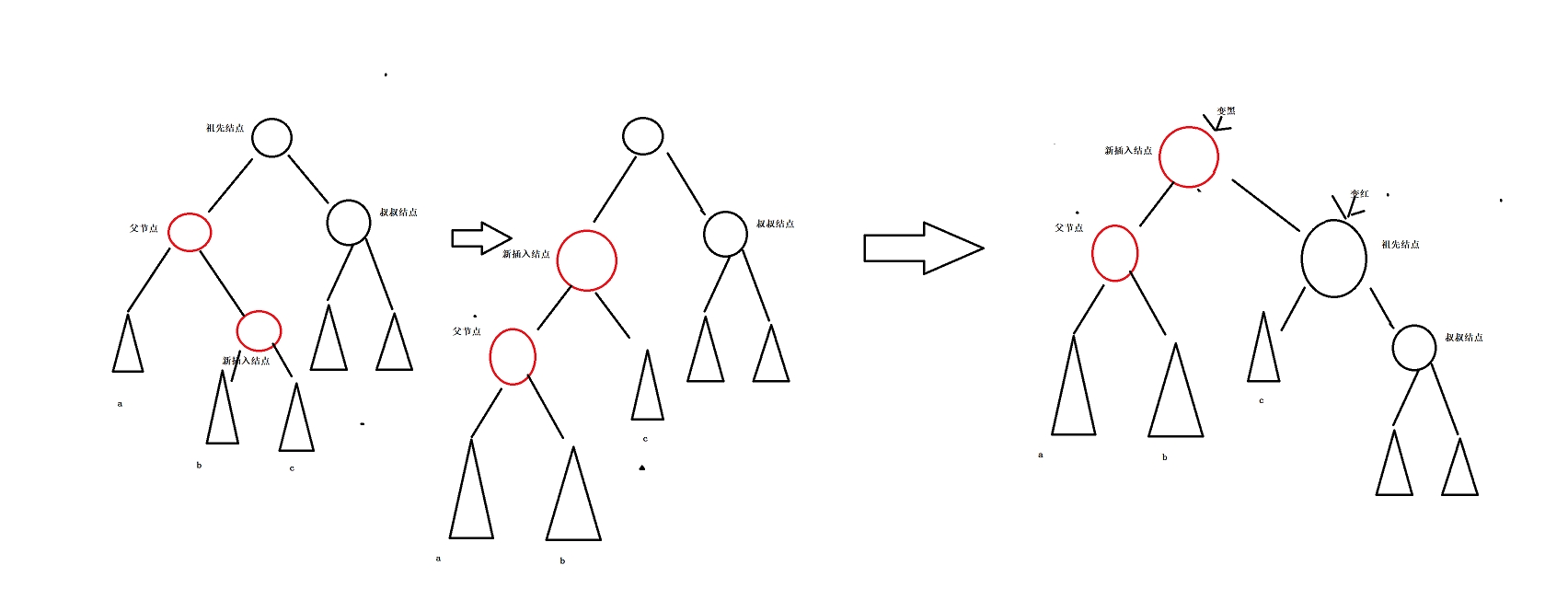
红黑树: 一种接近平衡的二叉树,平衡程度低于搜索二叉树。 特点: 1.根节点为黑 2.黑色结点的子结点可以是红色结点或黑色结点。 3.红色结点的子结点只能是黑色结点。 4.每个结点到其所有叶子结点的路径的黑色结点个数相同。 5.指向空的…...
opencv-使用 Haar 分类器进行面部检测
Haar 分类器是一种用于对象检测的方法,最常见的应用之一是面部检测。Haar 分类器基于Haar-like 特征,这些特征可以通过计算图像中的积分图来高效地计算。 在OpenCV中,Haar 分类器被广泛用于面部检测。以下是一个简单的使用OpenCV进行面部检测…...

C++纯虚函数和抽象类 制作饮品案例(涉及知识点:继承,多态,实例化继承抽象类的子类,多文件实现项目)
一.纯虚函数的由来 在多态中,通常父类中虚函数的实现是毫无意义的,主要都是调用子类重写的内容。例如: #include<iostream>using namespace std;class AbstractCalculator { public:int m_Num1;int m_Num2;virtual int getResult(){r…...

什么是网关和链路追踪,以及怎么使用?
在云计算中,网关和链路追踪也是非常重要的。云服务提供商通常会提供网关和链路追踪等相关服务来帮助用户管理和优化云计算网络。 云计算中的网关通常包括以下几种: 虚拟网络网关:用于将云中不同虚拟网络之间相互连接,实现云内不同…...

git 文件被莫名其妙的或略且无论如何都查不到哪个.gitignore文件忽略的
先说解决办法:git check-ignore -v [文件路径] 这个命令会返回一个忽略规则,以及该规则在哪个文件中定义的,该规则使得指定的文件被忽略。 1.遇到的问题 同项目组,其他同学都可以正常的提交.meta文件,我的提交就出现以…...
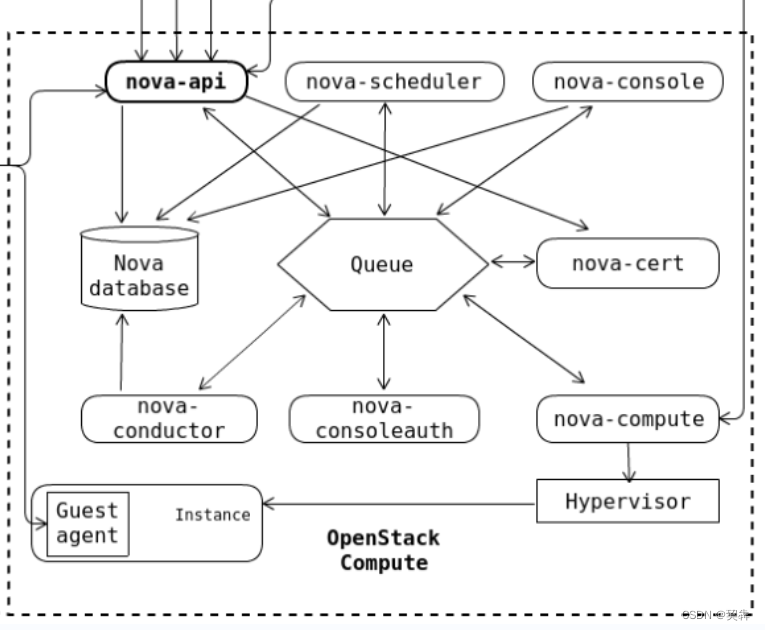
nova组件简介
目录 组件关系图 controller节点 openstack-nova-api.service: openstack-nova-conductor.service: openstack-nova-consoleauth.service: openstack-nova-novncproxy.service: openstack-nova-scheduler.service: openstack-nova-conductor.service详解 作用和功能&…...

【Vue】响应式与数据劫持
Vue.js 是一个渐进式的 JavaScript 框架,其中最重要的一个特性就是响应式(Reactivity)。Vue 借助数据劫持(Data Observation)技术实现了对数据的响应式更新,当数据发生变化时,它会自动重新渲染视…...

Modbus RTU转Profinet网关连接PLC与变频器通讯在机床上应用案例
背景:以前在机床加工车间里,工人们忙碌地操作着各种机床设备。为了使整个生产过程更加高效、流畅,进行智能化改造。 方案:在机床上,PLC通过Modbus RTU转Profinet网关连接变频器进行通讯:PLC作为整个生产线…...

Autoware 整体架构
Autoware 整体架构 测试...

【maven】手动指定jar推送
说明 为了推送第三方的jar,有时需要指定对应的jar推送到私有仓库。 示例 mvn deploy:deploy-file --settings /home/xxx/.m2/settings.xml -DgroupIdgroupId的值 -DartifactIdartifactId的值 -Dversionjar包的版本号 -Dpackagingjar -Dfilejar的路径 -Durlhttp:/…...

算法---定长子串中元音的最大数目
题目 给你字符串 s 和整数 k 。 请返回字符串 s 中长度为 k 的单个子字符串中可能包含的最大元音字母数。 英文中的 元音字母 为(a, e, i, o, u)。 示例 1: 输入:s “abciiidef”, k 3 输出:3 解释:…...
美国汽车零部件巨头 AutoZone 遭遇网络攻击
Security Affairs 网站披露,美国汽车配件零售商巨头 AutoZone 称其成为了 Clop MOVEit 文件传输网络攻击的受害者,导致大量数据泄露。 AutoZone 是美国最大的汽车零配件售后市场经销商之一,在美国、墨西哥、波多黎各、巴西和美属维尔京群岛经…...

WPF面试题入门篇
入门篇[2] 1. 谈谈什么是WPF? WPF(Windows Presentation Foundation)是微软公司开发的一种用于创建Windows应用程序的用户界面框架。它是.NET Framework的一部分,提供了一种基于XAML(可扩展应用程序标记语言…...
opencv-ORB检测
ORB(Oriented FAST and Rotated BRIEF)是一种图像特征检测和描述算法,结合了 FAST 关键点检测器和 BRIEF 描述子的优点。ORB 算法具有良好的性能,特别适用于实时应用,如目标追踪、相机定位等。 以下是 ORB 算法的一般…...

KubeSphere 容器平台高可用:环境搭建与可视化操作指南
Linux_k8s篇 欢迎来到Linux的世界,看笔记好好学多敲多打,每个人都是大神! 题目:KubeSphere 容器平台高可用:环境搭建与可视化操作指南 版本号: 1.0,0 作者: 老王要学习 日期: 2025.06.05 适用环境: Ubuntu22 文档说…...

RestClient
什么是RestClient RestClient 是 Elasticsearch 官方提供的 Java 低级 REST 客户端,它允许HTTP与Elasticsearch 集群通信,而无需处理 JSON 序列化/反序列化等底层细节。它是 Elasticsearch Java API 客户端的基础。 RestClient 主要特点 轻量级ÿ…...
XCTF-web-easyupload
试了试php,php7,pht,phtml等,都没有用 尝试.user.ini 抓包修改将.user.ini修改为jpg图片 在上传一个123.jpg 用蚁剑连接,得到flag...
)
Java 语言特性(面试系列2)
一、SQL 基础 1. 复杂查询 (1)连接查询(JOIN) 内连接(INNER JOIN):返回两表匹配的记录。 SELECT e.name, d.dept_name FROM employees e INNER JOIN departments d ON e.dept_id d.dept_id; 左…...

iOS 26 携众系统重磅更新,但“苹果智能”仍与国行无缘
美国西海岸的夏天,再次被苹果点燃。一年一度的全球开发者大会 WWDC25 如期而至,这不仅是开发者的盛宴,更是全球数亿苹果用户翘首以盼的科技春晚。今年,苹果依旧为我们带来了全家桶式的系统更新,包括 iOS 26、iPadOS 26…...
CentOS下的分布式内存计算Spark环境部署
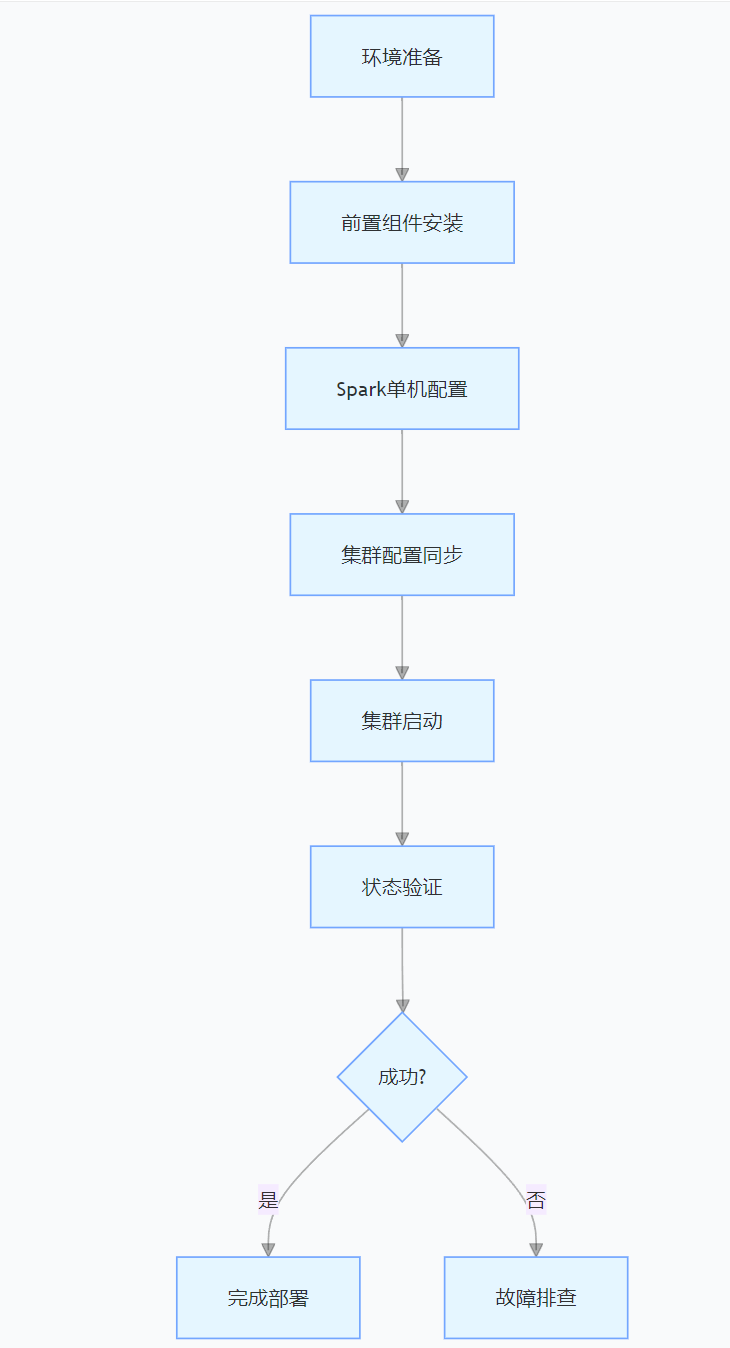
一、Spark 核心架构与应用场景 1.1 分布式计算引擎的核心优势 Spark 是基于内存的分布式计算框架,相比 MapReduce 具有以下核心优势: 内存计算:数据可常驻内存,迭代计算性能提升 10-100 倍(文档段落:3-79…...

Qt Http Server模块功能及架构
Qt Http Server 是 Qt 6.0 中引入的一个新模块,它提供了一个轻量级的 HTTP 服务器实现,主要用于构建基于 HTTP 的应用程序和服务。 功能介绍: 主要功能 HTTP服务器功能: 支持 HTTP/1.1 协议 简单的请求/响应处理模型 支持 GET…...
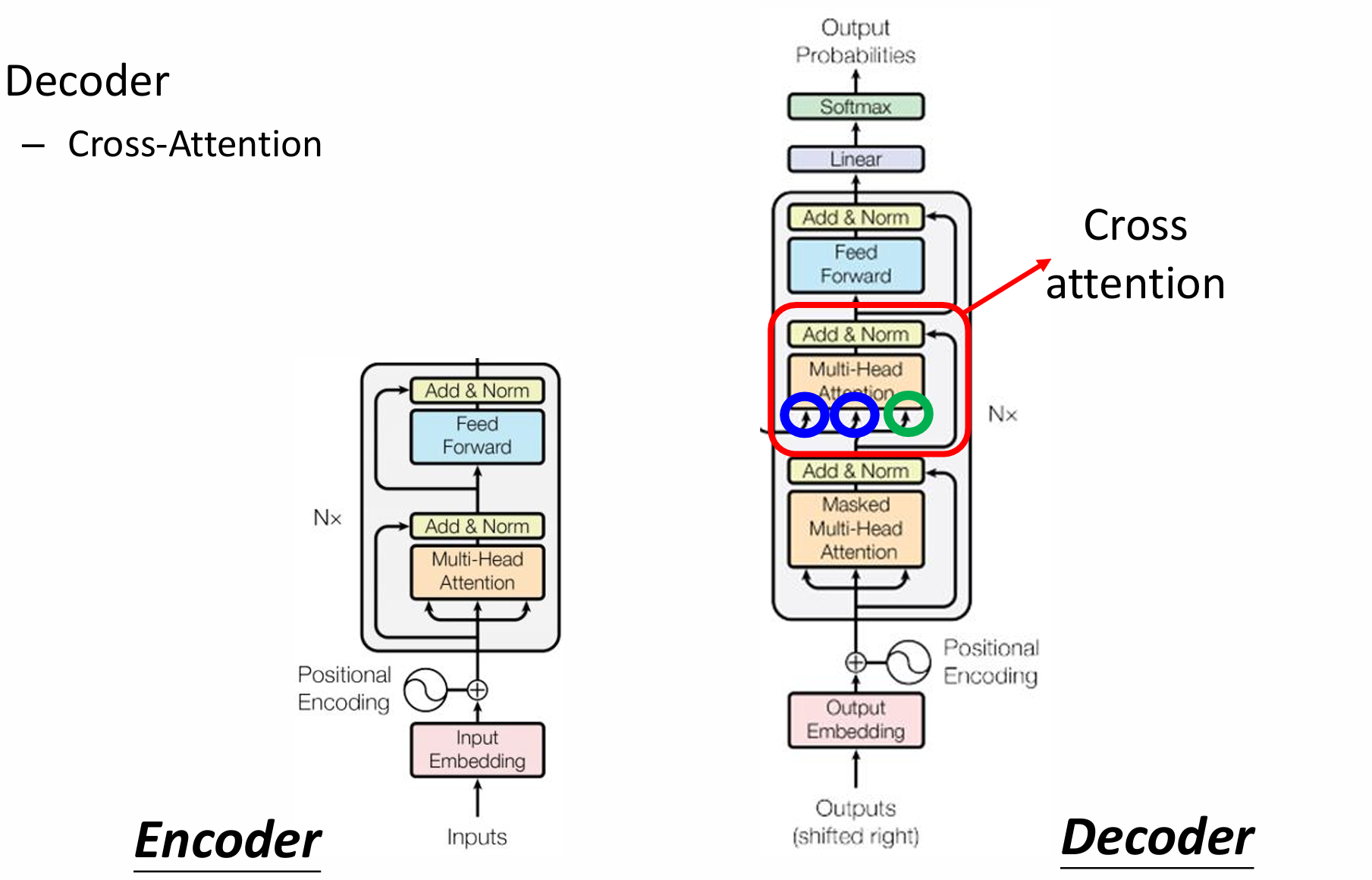
自然语言处理——Transformer
自然语言处理——Transformer 自注意力机制多头注意力机制Transformer 虽然循环神经网络可以对具有序列特性的数据非常有效,它能挖掘数据中的时序信息以及语义信息,但是它有一个很大的缺陷——很难并行化。 我们可以考虑用CNN来替代RNN,但是…...

Android15默认授权浮窗权限
我们经常有那种需求,客户需要定制的apk集成在ROM中,并且默认授予其【显示在其他应用的上层】权限,也就是我们常说的浮窗权限,那么我们就可以通过以下方法在wms、ams等系统服务的systemReady()方法中调用即可实现预置应用默认授权浮…...

力扣-35.搜索插入位置
题目描述 给定一个排序数组和一个目标值,在数组中找到目标值,并返回其索引。如果目标值不存在于数组中,返回它将会被按顺序插入的位置。 请必须使用时间复杂度为 O(log n) 的算法。 class Solution {public int searchInsert(int[] nums, …...
