机器学习之DeepSequence软件使用学习3-预测突变效应
import theano
import numpy as np
import sys
import pandas as pd
import scipy
#scipy 模块是 Python 中用于科学计算和数据分析的重要模块之一。它包含了许多高级的数学函数和工具,包括数值积分、优化、线性代数、统计等。
from scipy.stats import spearmanr
#scipy.stats模块是SciPy库中用于统计分析的模块,它提供了许多用于概率分布和统计测试的函数。%matplotlib inline
import matplotlib.pyplot as plt
我们将介绍加载模型和预测突变影响的基本函数。
下载预训练参数。
请首先使用 download_pretrained.sh 脚本下载预训练参数。
加载模型。
sys.path.insert(0, "../DeepSequence")import model
import helper
import train
突变影响预测。
突变影响预测辅助函数始终针对比对中的焦点序列。我们可以单独请求预测突变效应。
为了获得可靠的突变效应预测结果,我们建议从模型中取 Monte Carlo 500-2000 个样本(使用 N_pred_iterations 参数)。
我们可以预测单个、双重、三重突变等的影响。突变以元组列表的形式组织,其中元组为(Uniprot位置,野生型氨基酸,突变氨基酸)。
PABP
首先让我们加载一个模型。我们不需要在这里计算序列权重,因为我们不是在训练模型,而且在 CPU 上进行这项计算可能会很慢。
在 "Explore model parameters.ipynb" 笔记本中,helper.py 代码被修改以预先指定 DataHelper 类使用的数据集。然而,我们可以传入一个比对名称和一些额外参数,这样就不必修改 helper.py 文件。
data_params = {"alignment_file":"datasets/PABP_YEAST_hmmerbit_plmc_n5_m30_f50_t0.2_r115-210_id100_b48.a2m"}pabp_data_helper = helper.DataHelper(alignment_file=data_params["alignment_file"],working_dir=".",#指定当前程序的工作目录为当前所在的目录。calc_weights=False#不计算权重)model_params = {"batch_size" : 100,"encode_dim_zero" : 1500,"encode_dim_one" : 1500,"decode_dim_zero" : 100,"decode_dim_one" : 500,"n_patterns" : 4,"n_latent" : 30,"logit_p" : 0.001,"sparsity" : "logit","encode_nonlin" : "relu","decode_nonlin" : "relu","final_decode_nonlin": "sigmoid","output_bias" : True,"final_pwm_scale" : True,"conv_pat" : True,"d_c_size" : 40}pabp_vae_model = model.VariationalAutoencoder(pabp_data_helper,batch_size = model_params["batch_size"],encoder_architecture = [model_params["encode_dim_zero"],model_params["encode_dim_one"]],decoder_architecture = [model_params["decode_dim_zero"],model_params["decode_dim_one"]],n_latent = model_params["n_latent"],n_patterns = model_params["n_patterns"],convolve_patterns = model_params["conv_pat"],conv_decoder_size = model_params["d_c_size"],logit_p = model_params["logit_p"],sparsity = model_params["sparsity"],encode_nonlinearity_type = model_params["encode_nonlin"],decode_nonlinearity_type = model_params["decode_nonlin"],final_decode_nonlinearity = model_params["final_decode_nonlin"],output_bias = model_params["output_bias"],final_pwm_scale = model_params["final_pwm_scale"],working_dir = ".")print ("Model built")
我们查看一下datasets文件夹下的PABP_YEAST_hmmerbit_plmc_n5_m30_f50_t0.2_r115-210_id100_b48.a2m文件,显示有456123行,每三行为一组数据,表明有152041组数据
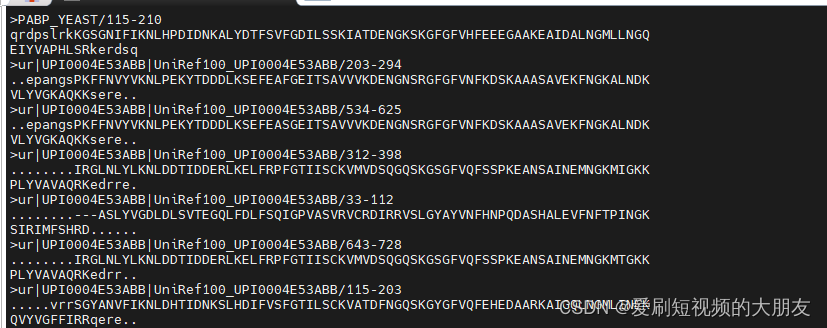 可以看到,比对文件是使用的类似于FASTA格式的氨基酸序列。
可以看到,比对文件是使用的类似于FASTA格式的氨基酸序列。
Encoding sequences
Neff = 151528.0
Data Shape = (151528, 82, 20)
Model built
加载预训练模型在 ‘params’ 文件夹中的参数。
file_prefix = "PABP_YEAST"
pabp_vae_model.load_parameters(file_prefix=file_prefix)
print ("Parameters loaded")
Parameters loaded
print (pabp_data_helper.delta_elbo(pabp_vae_model,[(126,"G","A")], N_pred_iterations=500))
-2.03463650668
print (pabp_data_helper.delta_elbo(pabp_vae_model,[(126,"G","A"), (137,"I","P")], N_pred_iterations=500))
-10.8308351474
注意,这里我单独计算了(137,“I”,“P”)的elbo,计算结果如下
>>> print (pabp_data_helper.delta_elbo(pabp_vae_model,[(137,"I","P")], N_pred_iterations=500))
-9.21612828741
可以看到,计算的结果不是每个突变的简单叠加。
print (pabp_data_helper.delta_elbo(pabp_vae_model,[(126,"G","A"), (137,"I","P"), (155,"S","A")], N_pred_iterations=500))
-16.058655309
这里我将同样的程序跑了三遍,但结果有略微出入。
>>> print (pabp_data_helper.delta_elbo(pabp_vae_model,[(126,"G","A"), (137,"I","P"), (155,"S","A")], N_pred_iterations=500))
-15.8832967199
>>> print (pabp_data_helper.delta_elbo(pabp_vae_model,[(126,"G","A"), (137,"I","P"), (155,"S","A")], N_pred_iterations=500))
-15.7348273612
>>> print (pabp_data_helper.delta_elbo(pabp_vae_model,[(126,"G","A"), (137,"I","P"), (155,"S","A")], N_pred_iterations=500))
-15.6141800809
我又计算了(155,“S”,“A”)的单独的突变影响,结果如下:
>>> print (pabp_data_helper.delta_elbo(pabp_vae_model,[(155,"S","A")], N_pred_iterations=500))
-4.43953605237
可见虽然不是每个位点突变的简单相加,但也近似于相加的结果。
我们可以预测所有单个突变的影响。优选使用此函数及以下函数,因为它们能够利用对突变数据进行小批量处理所带来的加速优势。
pabp_full_matr_mutant_name_list, pabp_full_matr_delta_elbos \= pabp_data_helper.single_mutant_matrix(pabp_vae_model, N_pred_iterations=500)
print (pabp_full_matr_mutant_name_list[0], pabp_full_matr_delta_elbos[0])
('K123A', 0.5887526915685584)
我们还可以以批处理模式从文件中预测突变的影响。
pabp_custom_matr_mutant_name_list, pabp_custom_matr_delta_elbos \= pabp_data_helper.custom_mutant_matrix("mutations/PABP_YEAST_Fields2013-singles.csv", \pabp_vae_model, N_pred_iterations=500)print (pabp_custom_matr_mutant_name_list[12], pabp_custom_matr_delta_elbos[12])
('N127D', -6.426795215037501)
我们也可以编写一个快速的函数来从一个突变文件计算 Spearman 系数(rho)。
def generate_spearmanr(mutant_name_list, delta_elbo_list, mutation_filename, phenotype_name):measurement_df = pd.read_csv(mutation_filename, sep=',')mutant_list = measurement_df.mutant.tolist()expr_values_ref_list = measurement_df[phenotype_name].tolist()mutant_name_to_pred = {mutant_name_list[i]:delta_elbo_list[i] for i in range(len(delta_elbo_list))}# If there are measurements wt_list = []preds_for_spearmanr = []measurements_for_spearmanr = []for i,mutant_name in enumerate(mutant_list):expr_val = expr_values_ref_list[i]# Make sure we have made a prediction for that mutantif mutant_name in mutant_name_to_pred:multi_mut_name_list = mutant_name.split(':')# If there is no measurement for that mutant, pass over itif np.isnan(expr_val):pass# If it was a codon change, add it to the wt vals to averageelif mutant_name[0] == mutant_name[-1] and len(multi_mut_name_list) == 1:wt_list.append(expr_values_ref_list[i])# If it is labeled as the wt sequence, add it to the average listelif mutant_name == 'wt' or mutant_name == 'WT':wt_list.append(expr_values_ref_list[i])else:measurements_for_spearmanr.append(expr_val)preds_for_spearmanr.append(mutant_name_to_pred[mutant_name])if wt_list != []:measurements_for_spearmanr.append(np.mean(average_wt_list))preds_for_spearmanr.append(0.0)num_data = len(measurements_for_spearmanr)spearman_r, spearman_pval = spearmanr(measurements_for_spearmanr, preds_for_spearmanr)print ("N: "+str(num_data)+", Spearmanr: "+str(spearman_r)+", p-val: "+str(spearman_pval))
generate_spearmanr(pabp_custom_matr_mutant_name_list, pabp_custom_matr_delta_elbos, \"mutations/PABP_YEAST_Fields2013-singles.csv", "log")
N: 1188, Spearmanr: 0.6509305755221257, p-val: 4.0800344026520655e-144
PDZ
data_params = {"alignment_file":"datasets/DLG4_RAT_hmmerbit_plmc_n5_m30_f50_t0.2_r300-400_id100_b50.a2m"}pdz_data_helper = helper.DataHelper(alignment_file=data_params["alignment_file"],working_dir=".",calc_weights=False)pdz_vae_model = model.VariationalAutoencoder(pdz_data_helper,batch_size = model_params["batch_size"],encoder_architecture = [model_params["encode_dim_zero"],model_params["encode_dim_one"]],decoder_architecture = [model_params["decode_dim_zero"],model_params["decode_dim_one"]],n_latent = model_params["n_latent"],n_patterns = model_params["n_patterns"],convolve_patterns = model_params["conv_pat"],conv_decoder_size = model_params["d_c_size"],logit_p = model_params["logit_p"],sparsity = model_params["sparsity"],encode_nonlinearity_type = model_params["encode_nonlin"],decode_nonlinearity_type = model_params["decode_nonlin"],final_decode_nonlinearity = model_params["final_decode_nonlin"],output_bias = model_params["output_bias"],final_pwm_scale = model_params["final_pwm_scale"],working_dir = ".")print ("Model built")file_prefix = "DLG4_RAT"
pdz_vae_model.load_parameters(file_prefix=file_prefix)print ("Parameters loaded\n\n")pdz_custom_matr_mutant_name_list, pdz_custom_matr_delta_elbos \= pdz_data_helper.custom_mutant_matrix("mutations/DLG4_RAT_Ranganathan2012.csv", \pdz_vae_model, N_pred_iterations=500)generate_spearmanr(pdz_custom_matr_mutant_name_list, pdz_custom_matr_delta_elbos, \"mutations/DLG4_RAT_Ranganathan2012.csv", "CRIPT")
Encoding sequences
Neff = 102246.0
Data Shape = (102246, 84, 20)
Model built
Parameters loadedN: 1577, Spearmanr: 0.6199244929585085, p-val: 4.31636475994128e-168
B-lactamase
对于包含更多待预测突变的较大蛋白质,运行时间可能会更长。针对这种情况,我们建议使用支持 GPU 的计算。
data_params = {"dataset":"BLAT_ECOLX"}blat_data_helper = helper.DataHelper(dataset=data_params["dataset"],working_dir=".",calc_weights=False)blat_vae_model = model.VariationalAutoencoder(blat_data_helper,batch_size = model_params["batch_size"],encoder_architecture = [model_params["encode_dim_zero"],model_params["encode_dim_one"]],decoder_architecture = [model_params["decode_dim_zero"],model_params["decode_dim_one"]],n_latent = model_params["n_latent"],n_patterns = model_params["n_patterns"],convolve_patterns = model_params["conv_pat"],conv_decoder_size = model_params["d_c_size"],logit_p = model_params["logit_p"],sparsity = model_params["sparsity"],encode_nonlinearity_type = model_params["encode_nonlin"],decode_nonlinearity_type = model_params["decode_nonlin"],final_decode_nonlinearity = model_params["final_decode_nonlin"],output_bias = model_params["output_bias"],final_pwm_scale = model_params["final_pwm_scale"],working_dir = ".")print ("Model built")file_prefix = "BLAT_ECOLX"
blat_vae_model.load_parameters(file_prefix=file_prefix)print ("Parameters loaded\n\n")blat_custom_matr_mutant_name_list, blat_custom_matr_delta_elbos \= blat_data_helper.custom_mutant_matrix("mutations/BLAT_ECOLX_Ranganathan2015.csv", \blat_vae_model, N_pred_iterations=500)generate_spearmanr(blat_custom_matr_mutant_name_list, blat_custom_matr_delta_elbos, \"mutations/BLAT_ECOLX_Ranganathan2015.csv", "2500")
Encoding sequences
Neff = 8355.0
Data Shape = (8355, 253, 20)
Model built
Parameters loadedN: 4807, Spearmanr: 0.743886370415797, p-val: 0.0
相关文章:

机器学习之DeepSequence软件使用学习3-预测突变效应
import theano import numpy as np import sys import pandas as pd import scipy #scipy 模块是 Python 中用于科学计算和数据分析的重要模块之一。它包含了许多高级的数学函数和工具,包括数值积分、优化、线性代数、统计等。 from scipy.stats import spearmanr #…...
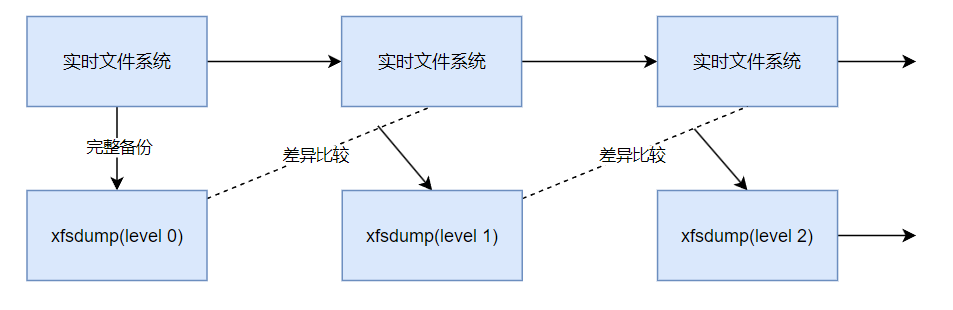
Linux文件与文件系统的压缩
文章目录 Linux文件与文件系统的压缩Linux系统常见的压缩命令gzip,zcat/zmore/zless/zgrepbzip2,bzcat/bzmore/bzless/bzgreppxz,xzcat/xzmore/xzless/xzgrepgzip,bzip2,xz压缩时间对比打包命令:tar打包命令…...

ubuntu 中进入python 编辑如何退出到命令行
文章目录 在Python解释器(交互式命令行)中,你可以使用 exit()函数或 CtrlD(在Unix/Linux/macOS上)或 CtrlZ然后输入 Enter(在Windows上)来退出Python解释器并返回到命令行。 以下是具体的步骤&a…...
2024.3.12 C++
1.思维导图 2.自己封装一个矩形类(Rect),拥有私有属性:宽度(width)、高度(height),定义公有成员函数: 初始化函数:void init(int w, int h)更改宽度的函数:set_w(int w)更改高度的函数:set_h(int h) 输出该矩形的周长和面积函数:void show() #include <iostream…...

飞塔防火墙开局百篇——002.FortiGate上网配置——透明模式配置(Transparent)
透明模式配置 开启透明模式创建策略 在不改变现有网络拓扑前提下,将防火墙NGFW以透明模式部署到网络中,放在路由器和交换机之间,防火墙为透明模式,对内网网段192.168.1.0/24的上网进行4~7层的安全防护。 登陆FortiGate防火墙界面&…...

代码随想录算法训练营第52天|300.最长递增子序列 674.最长连续递增序列 718.最长重复子数组
300.最长递增子序列 这道题还挺简单的,咱们设置dp[i]表示到第i个数字时的递增子序列的最长的值,那么dp[i]就要遍历从0到i-1的数,也就是看看当前这个数字是否比前面的数字大,如果大的话就看看现在的子序列长度是否会长于前面那个数…...

分享一些开源的游戏仓库
1.CnC_Remastered_Collection 红色警戒95版本 https://github.com/electronicarts/CnC_Remastered_Collection gitee仓库分流:https://gitee.com/loswdarmy/CnC_Remastered_Collection 2.Far-Cry-1-Source-Full 孤岛惊魂1 https://github.com/StrongPC123/Far-Cry-…...
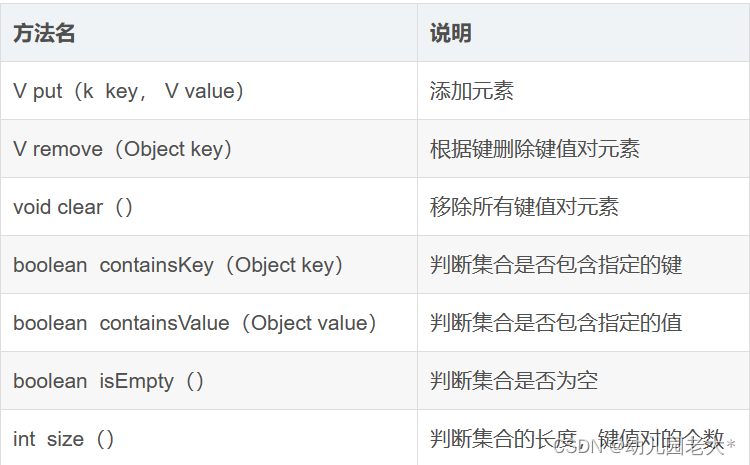
Java详解:单列 | 双列集合 | Collections类
○ 前言: 在开发实践中,我们需要一些能够动态增长长度的容器来保存我们的数据,java中为了解决数据存储单一的情况,java中就提供了不同结构的集合类,可以让我们根据不同的场景进行数据存储的选择,如Java中提…...

Centos7 使用docker来部署mondb
参考官方手册: https://www.mongodb.com/docs/manual/tutorial/install-mongodb-community-with-docker/#std-label-docker-mongodb-community-install 使用脚本快速安装docker curl -fsSL https://get.docker.com -o get-docker.sh | bash get-docker.sh使用 Doc…...
)
Java SE入门及基础(35)
接口 1. 概念 在软件工程中,软件与软件的交互很重要,这就需要一个约定。每个程序员都应该能够编写实现这样的约定。接口就是对约定的描述。 In the Java programming language, an interface is a reference type, similar to a class, that can con…...

基于YOLOv8/YOLOv7/YOLOv6/YOLOv5的常见车型识别系统(Python+PySide6界面+训练代码)
摘要:本文深入探讨了如何应用深度学习技术开发一个先进的常见车型识别系统。该系统核心采用最新的YOLOv8算法,并与早期的YOLOv7、YOLOv6、YOLOv5等版本进行性能比较,主要评估指标包括mAP和F1 Score等。详细解析了YOLOv8的工作机制,…...

Sqoop 学习
参考视频 大数据Sqoop教程丨从零开始讲解大数据业务及数据采集和迁移需求_哔哩哔哩_bilibili 介绍 Sqoop是Hadoop生态体系和RDBMS(关系型数据库)体系之间传送数据的一种工具 Hadop生态系统:HDFS,Hbase,Hive等 RDBMS包…...

Ollama 只安装 Ollama,本地快速部署谷歌开源大模型Gemma(基于Ollama)
参考:本地快速部署谷歌开源大模型Gemma(基于Ollama) - 知乎 确保系统更新: Bash sudo apt update && sudo apt upgrade 需要先下载Ollama,版本要求0.1.26及以上 运行curl -fsSL https://ollama.com/install.sh | sh 监听 Ollama API 接…...
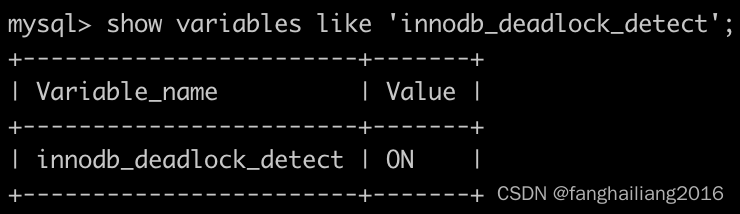
一条 sql 语句可能导致的表锁和行锁以及死锁检测
锁 MDL 当对一个表做增删改查操作的时候,加 MDL 读锁;当要对表做结构变更操作的时候,加 MDL 写锁 ALTER TABLE tbl_name NOWAIT add column ... ALTER TABLE tbl_name WAIT N add column ... …...
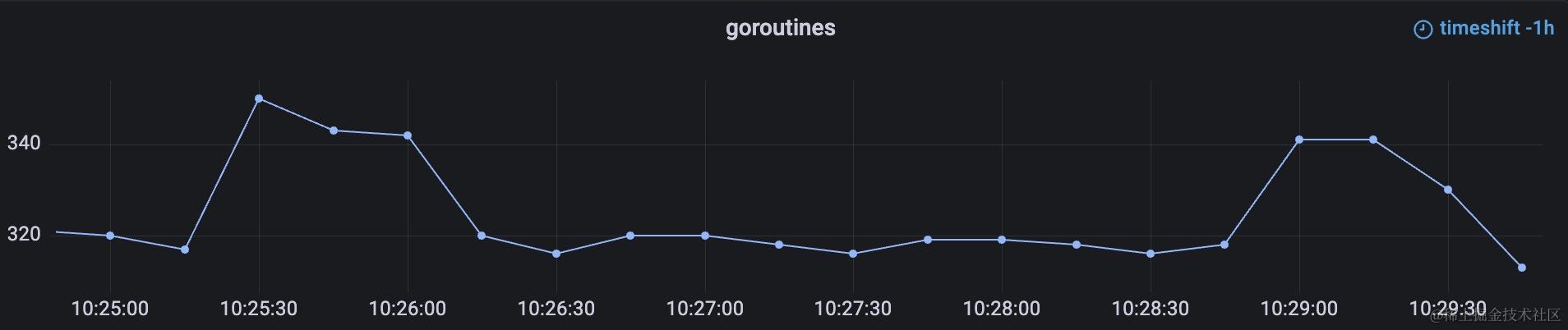
prometheus 原理(架构,promql表达式,描点原理)
大家好,我是蓝胖子,提到监控指标,不得不说prometheus,今天这篇文章我会对prometheus 的架构设计,promql表达式原理和监控图表的绘图原理进行详细的解释。来让大家对prometheus的理解更加深刻。 架构设计 先来看看&am…...
)
Linux的目录结构(介绍主要的)
/:根目录,文件系统的起点,包含了所有目录和文件 /bin:存放基本的可执行命令,如ls,cp,rm /lib:主要存放动态链接库 /opt:供第三方软件安装的目录,通常将软件…...
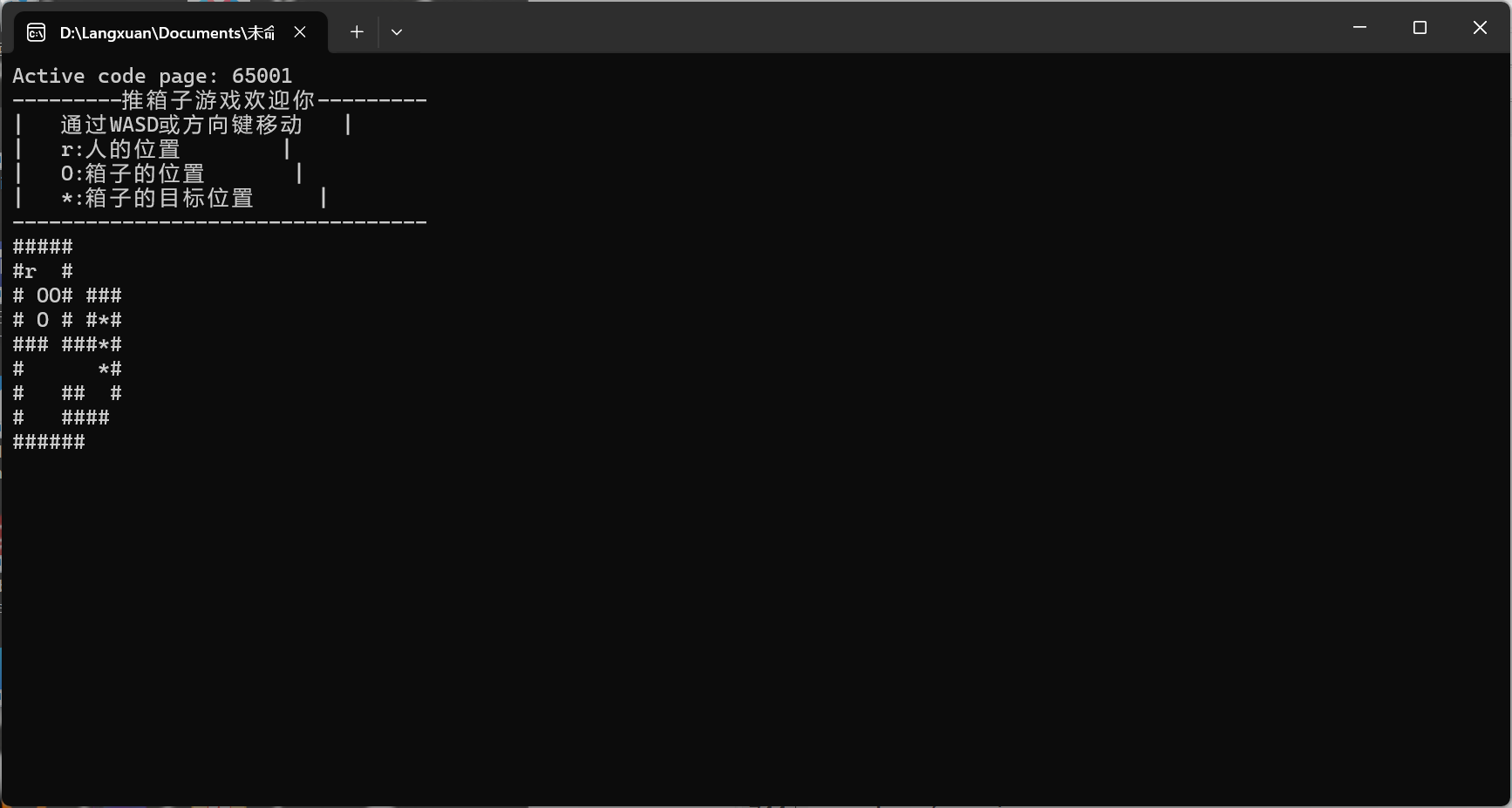
推房子游戏c++
这段代码是一个推箱子游戏的实现。游戏中有一个地图,地图上有墙壁、人、箱子和目标位置。玩家通过键盘输入WASD或方向键来控制人物的移动,目标是将所有的箱子推到相应的目标位置上。 代码中的dt数组表示地图,每个位置上的字符表示对应的元素…...
docker学习入门篇
1、docker简介 docker官网: www.docker.com dockerhub官网: hub.docker.com docker文档官网:docs.docker.com Docker是基于Go语言实现的云开源项目。 Docker的主要目标是:Build, Ship and Run Any App, Anywhere(构建&…...

【Spring Boot 3】动态注入和移除Bean
【Spring Boot 3】动态注入和移除Bean 背景介绍开发环境开发步骤及源码工程目录结构总结动态注入Bean的方法动态移除Bean的方法注意事项背景 软件开发是一门实践性科学,对大多数人来说,学习一种新技术不是一开始就去深究其原理,而是先从做出一个可工作的DEMO入手。但在我个…...
555经典电路
1、555介绍: 555 定时器是一种模拟和数字功能相结合的中规模集成器件。一般用双极性工艺制作的称为 555,用 CMOS 工艺制作的称为 7555,除单定时器外,还有对应的双定时器 556/7556。555 定时器的电源电压范围宽,可在 4…...
:OpenBCI_GUI:从环境搭建到数据可视化(下))
脑机新手指南(八):OpenBCI_GUI:从环境搭建到数据可视化(下)
一、数据处理与分析实战 (一)实时滤波与参数调整 基础滤波操作 60Hz 工频滤波:勾选界面右侧 “60Hz” 复选框,可有效抑制电网干扰(适用于北美地区,欧洲用户可调整为 50Hz)。 平滑处理&…...
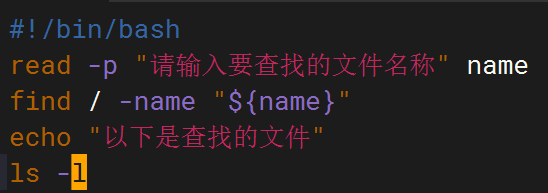
shell脚本--常见案例
1、自动备份文件或目录 2、批量重命名文件 3、查找并删除指定名称的文件: 4、批量删除文件 5、查找并替换文件内容 6、批量创建文件 7、创建文件夹并移动文件 8、在文件夹中查找文件...

Xen Server服务器释放磁盘空间
disk.sh #!/bin/bashcd /run/sr-mount/e54f0646-ae11-0457-b64f-eba4673b824c # 全部虚拟机物理磁盘文件存储 a$(ls -l | awk {print $NF} | cut -d. -f1) # 使用中的虚拟机物理磁盘文件 b$(xe vm-disk-list --multiple | grep uuid | awk {print $NF})printf "%s\n"…...

Java数值运算常见陷阱与规避方法
整数除法中的舍入问题 问题现象 当开发者预期进行浮点除法却误用整数除法时,会出现小数部分被截断的情况。典型错误模式如下: void process(int value) {double half = value / 2; // 整数除法导致截断// 使用half变量 }此时...

AI+无人机如何守护濒危物种?YOLOv8实现95%精准识别
【导读】 野生动物监测在理解和保护生态系统中发挥着至关重要的作用。然而,传统的野生动物观察方法往往耗时耗力、成本高昂且范围有限。无人机的出现为野生动物监测提供了有前景的替代方案,能够实现大范围覆盖并远程采集数据。尽管具备这些优势…...

CRMEB 中 PHP 短信扩展开发:涵盖一号通、阿里云、腾讯云、创蓝
目前已有一号通短信、阿里云短信、腾讯云短信扩展 扩展入口文件 文件目录 crmeb\services\sms\Sms.php 默认驱动类型为:一号通 namespace crmeb\services\sms;use crmeb\basic\BaseManager; use crmeb\services\AccessTokenServeService; use crmeb\services\sms\…...

Kafka主题运维全指南:从基础配置到故障处理
#作者:张桐瑞 文章目录 主题日常管理1. 修改主题分区。2. 修改主题级别参数。3. 变更副本数。4. 修改主题限速。5.主题分区迁移。6. 常见主题错误处理常见错误1:主题删除失败。常见错误2:__consumer_offsets占用太多的磁盘。 主题日常管理 …...
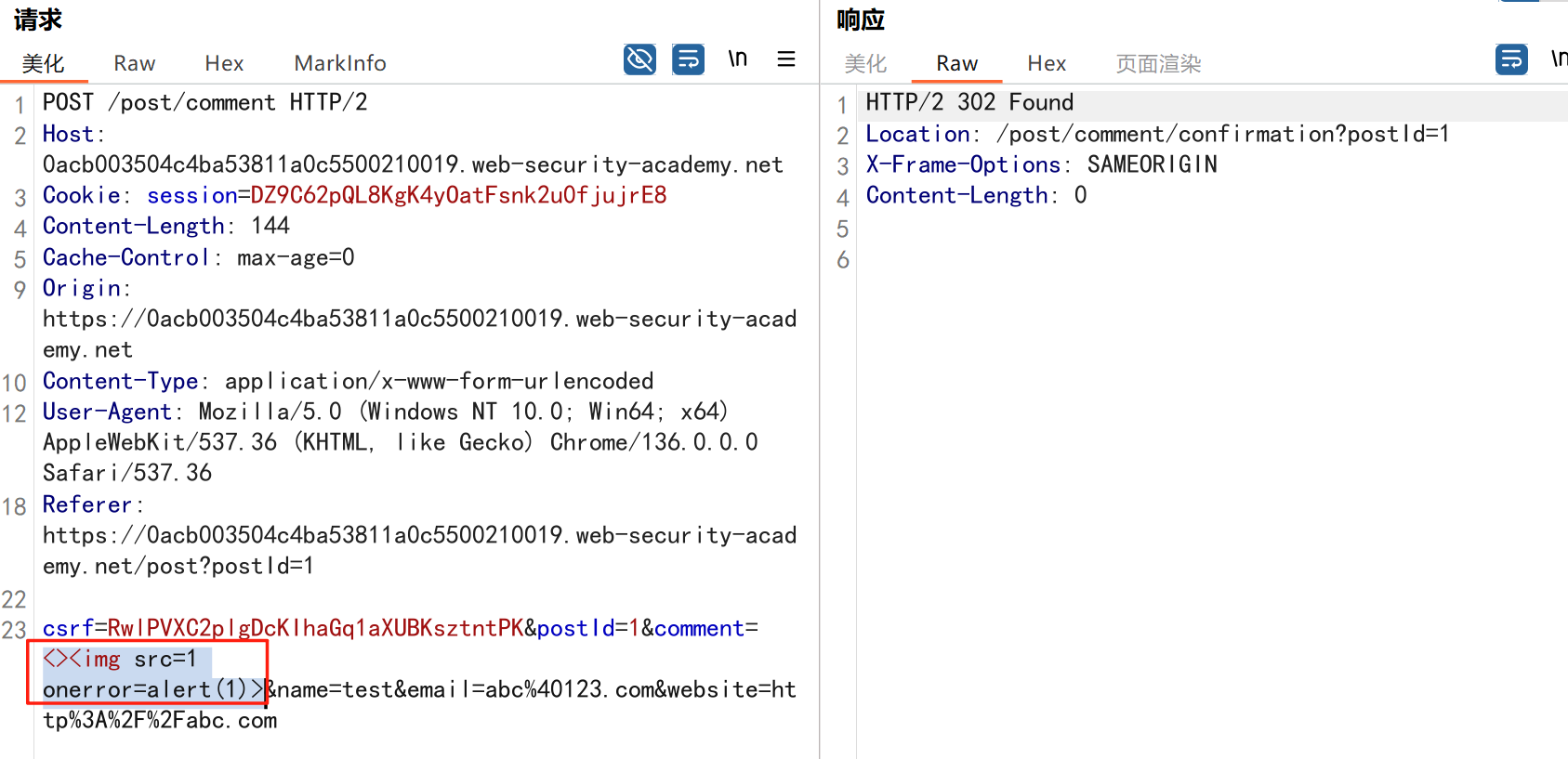
渗透实战PortSwigger靶场:lab13存储型DOM XSS详解
进来是需要留言的,先用做简单的 html 标签测试 发现面的</h1>不见了 数据包中找到了一个loadCommentsWithVulnerableEscapeHtml.js 他是把用户输入的<>进行 html 编码,输入的<>当成字符串处理回显到页面中,看来只是把用户输…...

数据结构:递归的种类(Types of Recursion)
目录 尾递归(Tail Recursion) 什么是 Loop(循环)? 复杂度分析 头递归(Head Recursion) 树形递归(Tree Recursion) 线性递归(Linear Recursion)…...

内窥镜检查中基于提示的息肉分割|文献速递-深度学习医疗AI最新文献
Title 题目 Prompt-based polyp segmentation during endoscopy 内窥镜检查中基于提示的息肉分割 01 文献速递介绍 以下是对这段英文内容的中文翻译: ### 胃肠道癌症的发病率呈上升趋势,且有年轻化倾向(Bray等人,2018&#x…...
