GEO生信数据挖掘(四)数据清洗(离群值处理、低表达基因、归一化、log2处理)
检索到目标数据集后,开始数据挖掘,本文以阿尔兹海默症数据集GSE1297为例
目录
离群值处理
删除 低表达基因
函数归一化,矫正差异
数据标准化—log2处理
完整代码
上节围绕着探针ID和基因名称做了一些清洗工作,还做了重复值检查,空值删除操作。
#查看重复值
table(duplicated(matrix$Gene.Symbol))#去掉缺失值
matrix_na = na.omit(matrix) #基因名称为空删除
matrix_final = matrix_na[matrix_na$Gene.Symbol != "",]离群值处理
用于处理异常值,将超出一定范围的值替换为中位数,以减少异常值对后续分析的影响。
#数据离群处理
#处理极端值
#定义向量极端值处理函数
#用于处理异常值,将超出一定范围的值替换为中位数,以减少异常值对后续分析的影响。
dljdz=function(x) {DOWNB=quantile(x,0.25)-1.5*(quantile(x,0.75)-quantile(x,0.25))UPB=quantile(x,0.75)+1.5*(quantile(x,0.75)-quantile(x,0.25))x[which(x<DOWNB)]=quantile(x,0.5)x[which(x>UPB)]=quantile(x,0.5)return(x)
}#第一列设置为行名
matrix_leave=matrix_finalboxplot(matrix_leave,outline=FALSE, notch=T, las=2) ##出箱线图
dim(matrix_leave)#处理离群值
matrix_leave_res=apply(matrix_leave,2,dljdz)boxplot(matrix_leave_res,outline=FALSE, notch=T, las=2) ##出箱线图
dim(matrix_leave_res)
删除 低表达基因
方案1 :仅去除在所有样本里表达量都为零的基因(或者平均值小于1)
方案2:仅保留在一半(50%,75%...自己选择)以上样本里表达的基因
#删除 低表达基因#仅去除在所有样本里表达量都为零的基因(平均值小于1)# 计算基因表达矩阵的平均值
gene_avg <- apply(matrix_final, 1, mean)
# 根据阈值筛选低表达基因
filtered_genes_1 <- matrix_final[gene_avg >= 1, ] # 表达量平均值小于1的过滤
dim(filtered_genes_1)#+================================
#常用过滤标准2(推荐):
#仅保留在一半以上样本里表达的基因# 计算基因表达矩阵每个基因的平均值
gene_means <- rowMeans(matrix_final)# 计算基因平均值的排序百分位数
gene_percentiles <- rank(gene_means) / length(gene_means)# 获取阈值
threshold <- 0.25 # 删除后25%的阈值
#threshold <- 0.5 # 删除后50%的阈值
# 根据阈值筛选低表达基因
filtered_genes_2 <- matrix_final[gene_percentiles > threshold, ]# 打印筛选后的基因表达矩阵
dim(filtered_genes_2)boxplot(filtered_genes_2,outline=FALSE, notch=T, las=2) ##出箱线图
#dim(filtered_genes)#+删除 低表达基因 得到filtered_genes_1 删除平均表达小于1的 得到filtered_genes_2 删除后25%的函数归一化,矫正差异
#1.归一化不是绝对必要的,但是推荐进行归一化。
#有重复的样本中,应该不具备生物学意义的外部因素会影响单个样品的表达,
#例如中第一批制备的样品会总体上表达高于第二批制备的样品,假设所有样品表达值的范围和分布都应当相似,
#需要进行归一化来确保整个实验中每个样本的表达分布都相似。
#2.归一化要在log2标准化之前做
library(limma) exprSet=normalizeBetweenArrays(filtered_genes_2)boxplot(exprSet,outline=FALSE, notch=T, las=2) ##出箱线图## 这步把矩阵转换为数据框很重要
class(exprSet) ##注释:此时数据的格式是矩阵(Matrix)
exprSet <- as.data.frame(exprSet)数据标准化—log2处理
如果表达量的数值在50以内,通常是经过log2转化后的。如果数字在几百几千,则是未经转化的。
GSE数据集的注释部分会有说明,常见的标准化处理方法有3种:RMA算法、GC-RMA算法、MAS5算法
#标准化 表达矩阵自动log2化
qx <- as.numeric(quantile(exprSet, c(0., 0.25, 0.5, 0.75, 0.99, 1.0), na.rm=T))
LogC <- (qx[5] > 100) ||(qx[6]-qx[1] > 50 && qx[2] > 0) ||(qx[2] > 0 && qx[2] < 1 && qx[4] > 1 && qx[4] < 2)## 开始判断
if (LogC) { exprSet [which(exprSet <= 0)] <- NaN## 取log2exprSet_clean <- log2(exprSet)print("log2 transform finished")
}else{print("log2 transform not needed")
}筛选后的基因表达矩阵的箱线图
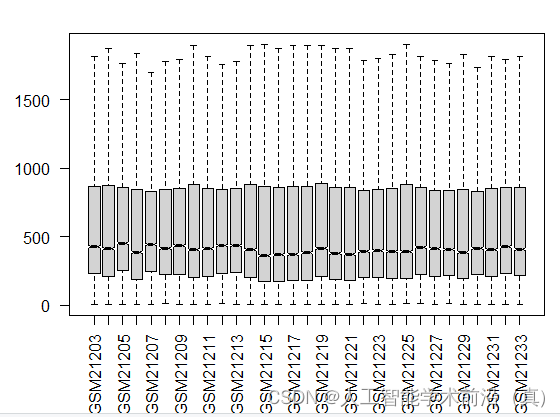
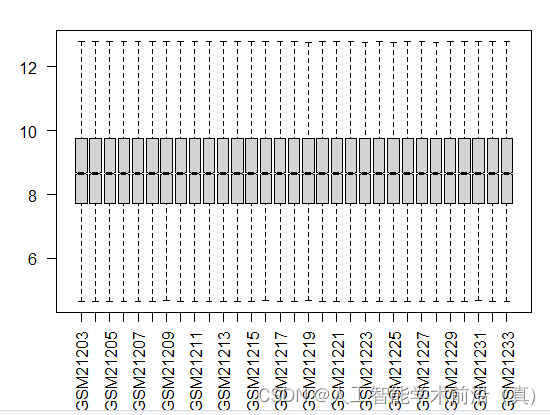
经过数据清洗后的箱线图
完整代码
# 安装并加载GEOquery包
library(GEOquery)# 指定GEO数据集的ID
gse_id <- "GSE1297"# 使用getGEO函数获取数据集的基础信息
gse_info <- getGEO(gse_id, destdir = ".", AnnotGPL = F ,getGPL = F) # Failed to download ./GPL96.soft.gz!# 提取基因表达矩阵
expression_data <- exprs(gse_info[[1]])# 提取注释信息
#annotation <- featureData(gse_info[[1]])#查看平台文件列名
colnames(annotation)#打印项目文件列表
dir() # 读取芯片平台文件txt
platform_file <- read.delim("GPL96-57554.txt", header = TRUE, sep = "\t", comment.char = "#")#查看平台文件列名
colnames(platform_file)# 假设芯片平台文件中有两列,一列是探针ID,一列是基因名
#probe_names <- platform_file$ID
#gene_symbols <- platform_file$Gene.Symbol
platform_file_set=platform_file[,c(1,11)]#一个探针对应多个基因名,保留第一个基因名
ids = platform_file_set
library(tidyverse)
test_function <- apply(ids,1,function(x){paste(x[1],str_split(x[2],'///',simplify=T),sep = "...")})
x = tibble(unlist(test_function))colnames(x) <- "ttt"
ids <- separate(x,ttt,c("ID","Gene.Symbol"),sep = "\\...")
dim(ids)#将Matrix格式表达矩阵转换为data.frame格式
exprSet <- data.frame(expression_data)
dim(exprSet)#给表达矩阵新增加一列ID
exprSet$ID <- rownames(exprSet) # 得到表达矩阵,行名为ID,需要转换,新增一列
dim(exprSet)
#矩阵表达文件和平台文件有相同列‘ID’,使用merge函数合并
express <- merge(x = exprSet, y = ids, by.x = "ID")#删除探针ID列
express$ID =NULLdim(express) matrix = express
dim(matrix)
#查看多少个基因重复了
table(duplicated(matrix$Gene.Symbol))#把重复的Symbol取平均值
matrix <- aggregate(.~Gene.Symbol, matrix, mean) ##把重复的Symbol取平均值
row.names(matrix) <- matrix$Gene.Symbol #把行名命名为SYMBOLdim(matrix)matrix_na = na.omit(matrix) #去掉缺失值
dim(matrix_na)matrix_final = matrix_na[matrix_na$Gene.Symbol != "",]
dim(matrix_final)matrix_final <- subset(matrix_final, select = -1) #删除Symbol列(一般是第一列)
dim(matrix_final)
#+ 经过注释、探针名基因名处理、删除基因名为空值、删除缺失值 得到最终 matrix_final
#+==================================================================================
#+========================================================================================#+==================================================================================
#+========================================================================================
#+增加#(2)离群值处理#数据离群处理
#处理极端值
#定义向量极端值处理函数
#用于处理异常值,将超出一定范围的值替换为中位数,以减少异常值对后续分析的影响。
dljdz=function(x) {DOWNB=quantile(x,0.25)-1.5*(quantile(x,0.75)-quantile(x,0.25))UPB=quantile(x,0.75)+1.5*(quantile(x,0.75)-quantile(x,0.25))x[which(x<DOWNB)]=quantile(x,0.5)x[which(x>UPB)]=quantile(x,0.5)return(x)
}#第一列设置为行名
matrix_leave=matrix_finalboxplot(matrix_leave,outline=FALSE, notch=T, las=2) ##出箱线图
dim(matrix_leave)#处理离群值
matrix_leave_res=apply(matrix_leave,2,dljdz)boxplot(matrix_leave_res,outline=FALSE, notch=T, las=2) ##出箱线图
dim(matrix_leave_res)#+增加#(2)离群值处理
#+==================================================================================
#+========================================================================================#+==================================================================================
#+========================================================================================
#删除 低表达基因#仅去除在所有样本里表达量都为零的基因(平均值小于1)# 计算基因表达矩阵的平均值
gene_avg <- apply(matrix_final, 1, mean)
# 根据阈值筛选低表达基因
filtered_genes_1 <- matrix_final[gene_avg >= 1, ] # 表达量平均值小于1的过滤
dim(filtered_genes_1)#+================================
#常用过滤标准2(推荐):
#仅保留在一半以上样本里表达的基因# 计算基因表达矩阵每个基因的平均值
gene_means <- rowMeans(matrix_final)# 计算基因平均值的排序百分位数
gene_percentiles <- rank(gene_means) / length(gene_means)# 获取阈值
threshold <- 0.25 # 删除后25%的阈值
#threshold <- 0.5 # 删除后50%的阈值
# 根据阈值筛选低表达基因
filtered_genes_2 <- matrix_final[gene_percentiles > threshold, ]# 打印筛选后的基因表达矩阵
dim(filtered_genes_2)boxplot(filtered_genes_2,outline=FALSE, notch=T, las=2) ##出箱线图
#dim(filtered_genes)#+删除 低表达基因 得到filtered_genes_1 删除平均表达小于1的 得到filtered_genes_2 删除后25%的
#+==================================================================================
#+========================================================================================#+==================================================================================
#+========================================================================================
### limma的函数归一化,矫正差异 ,表达矩阵自动log2化#1.归一化不是绝对必要的,但是推荐进行归一化。
#有重复的样本中,应该不具备生物学意义的外部因素会影响单个样品的表达,
#例如中第一批制备的样品会总体上表达高于第二批制备的样品,假设所有样品表达值的范围和分布都应当相似,
#需要进行归一化来确保整个实验中每个样本的表达分布都相似。
#2.归一化要在log2标准化之前做library(limma) exprSet=normalizeBetweenArrays(filtered_genes_2)boxplot(exprSet,outline=FALSE, notch=T, las=2) ##出箱线图## 这步把矩阵转换为数据框很重要
class(exprSet) ##注释:此时数据的格式是矩阵(Matrix)
exprSet <- as.data.frame(exprSet)#标准化 表达矩阵自动log2化
qx <- as.numeric(quantile(exprSet, c(0., 0.25, 0.5, 0.75, 0.99, 1.0), na.rm=T))
LogC <- (qx[5] > 100) ||(qx[6]-qx[1] > 50 && qx[2] > 0) ||(qx[2] > 0 && qx[2] < 1 && qx[4] > 1 && qx[4] < 2)## 开始判断
if (LogC) { exprSet [which(exprSet <= 0)] <- NaN## 取log2exprSet_clean <- log2(exprSet)print("log2 transform finished")
}else{print("log2 transform not needed")
}boxplot(exprSet_clean,outline=FALSE, notch=T, las=2) ##出箱线图
### limma的函数归一化,矫正差异 ,表达矩阵自动log2化 得到exprSet_clean
#+==================================================================================
#+========================================================================================# saving all data to the path
save(exprSet_clean, file ="exprSet_clean_75percent_filter.RData")上述处理得到了干净的基因表达矩阵,数据部分已经没有问题,但是在做数据挖掘(差异分析、富集分析等)之前还有一项准备工作,要将数据样本进行分组,即患病组、对照组。
相关文章:

GEO生信数据挖掘(四)数据清洗(离群值处理、低表达基因、归一化、log2处理)
检索到目标数据集后,开始数据挖掘,本文以阿尔兹海默症数据集GSE1297为例 目录 离群值处理 删除 低表达基因 函数归一化,矫正差异 数据标准化—log2处理 完整代码 上节围绕着探针ID和基因名称做了一些清洗工作,还做了重复值检查…...

CI/CD工具中的CI和CD的含义
CI/CD工具中的CI和CD的含义? CI/CD 是现代软件开发方法中广泛使用的一种方法。其中,CI 代表持续集成(Continuous Integration),CD 则有两层含义,一是持续交付(Continuous Delivery)…...

用go获取IPv4地址,WLAN的IPv4地址,本机公网IP地址详解
文章目录 获取IPv4地址获取WLAN的IPv4地址获取本机公网IP地址 获取IPv4地址 下面的代码会打印出本机所有的IPv4地址。这个方法可能会返回多个IP地址,因为一台机器可能有多个网络接口,每个接口可能有一个或多个IP地址。 package mainimport ("fmt&…...

Android自定义Drawable---灵活多变的矩形背景
Android自定义Drawable—灵活多变的矩形背景 在安卓开发中,我们通常需要为不同的按钮设置不同的背景以实现不同的效果,有时还需要这些按钮根据实际情况进行变化。如果采用编写resource中xml文件的形式,就需要重复定义许多只有微小变动的资源…...

ParagonNTFSforMac_15.5.102中文版最受欢迎的NTFS硬盘格式读取工具
Paragon NTFS for Mac是一款可以为您轻松解决Mac平台上不能识别Windows通用的NTFS文件难题,这是一款强大的Mac读写工具,相信在很多时候,Mac用户需要对NTFS文件的移动硬盘进行写入,但是macOS系统默认是不让写入的,使用小…...

Kafka 搭建过程
目录 1.关于Kafka2.Kafka 搭建过程3.参考 本文主要介绍Kafka基本原理,以及搭建过程。 1.关于Kafka Apache Kafka是一个开源的分布式事件流平台,被设计用来实现实时数据流的发布、订阅、存储和处理。 Kafka的主要特性包括: 高吞吐量&#x…...
七、2023.10.1.Linux(一).7
文章目录 1、 Linux中查看进程运行状态的指令、查看内存使用情况的指令、tar解压文件的参数。2、文件权限怎么修改?3、说说常用的Linux命令?4、说说如何以root权限运行某个程序?5、 说说软链接和硬链接的区别?6、说说静态库和动态…...
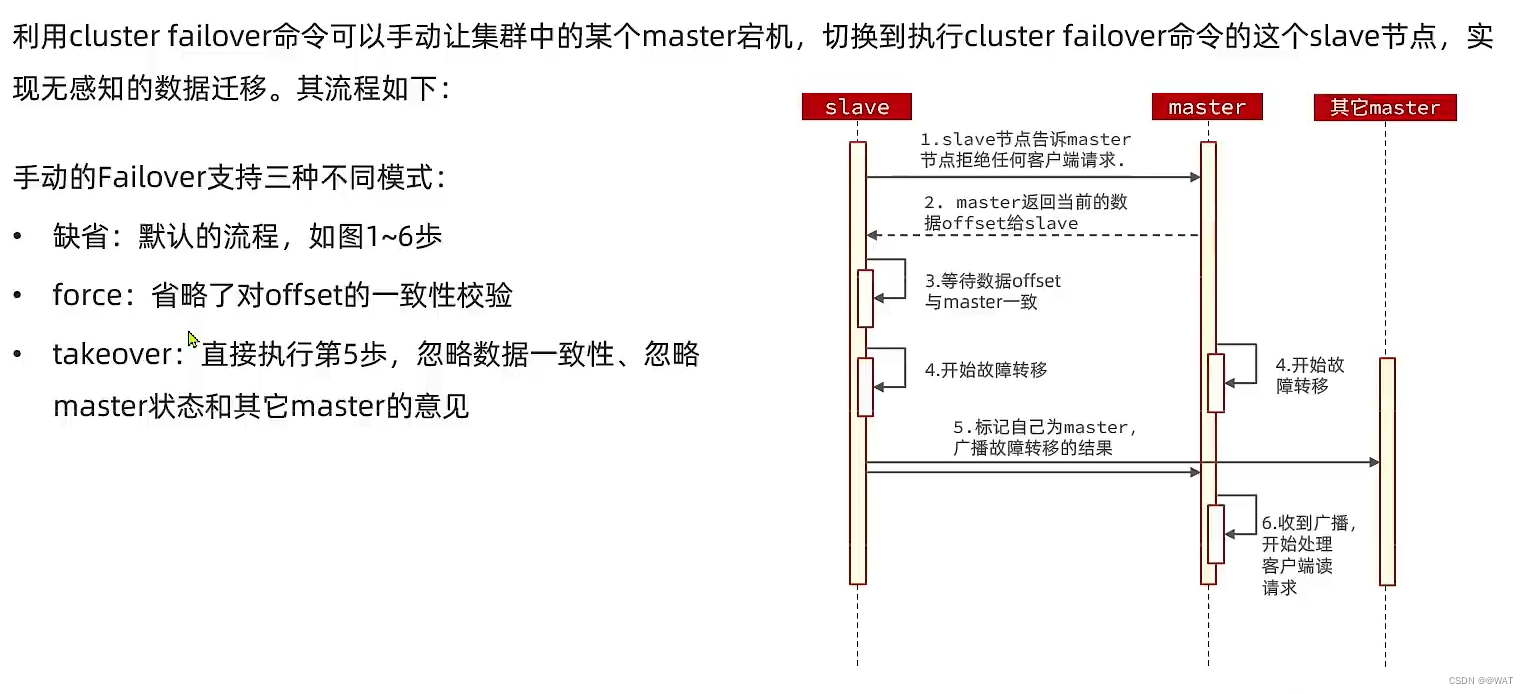
一文教你搞懂Redis集群
一、Redis主从 1.1、搭建主从架构 单节点的Redis的并发能力是有上限的,要进一步的提高Redis的并发能力,据需要大家主从集群,实现读写分离。 共包含三个实例,由于资源有限,所以在一台虚拟机上,开启多个red…...

树上启发式合并 待补
对于每个子树,直接遍历所有轻儿子,继承重儿子 会了板子后,修改维护的东西和莫队是一样的 洛谷 U41492 #include <bits/stdc.h> #define ll long long #define ull unsigned long long constexpr int N1e55; std::vector<int> e…...

minio分布式文件存储
基本介绍 什么是 MinIO MinIO 是一款基于 Go 语言的高性能、可扩展、云原生支持、操作简单、开源的分布式对象存储产品。基于 Apache License v2.0 开源协议,虽然轻量,却拥有着不错的性能。它兼容亚马逊S3云存储服务接口。可以很简单的和其他应…...

Linux新的IO模型io_uring
一、Linux下的网络通信模型 在网络开发的过程中,需要处理好几个问题。首先是通信的内核支持问题;其次是通信的模型问题;最后是框架问题。这些问题在闭源的OS如Windows上,基本上不算什么大问题(因为只能用人家的API&am…...
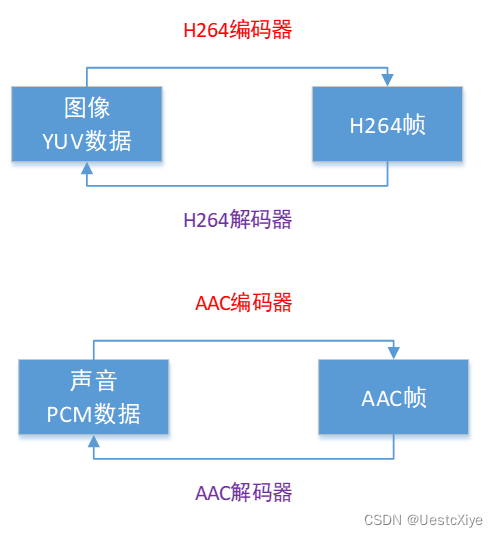
FFmpeg 命令:从入门到精通 | FFmpeg 基本介绍
FFmpeg 命令:从入门到精通 | FFmpeg 基本介绍 FFmpeg 命令:从入门到精通 | FFmpeg 基本介绍FFmpeg 简介FFmpeg 基础知识复用与解复用编解码器码率和帧率 资料 FFmpeg 命令:从入门到精通 | FFmpeg 基本介绍 本系列文章要解决的问题࿱…...

数组篇 第一题:删除排序数组中的重复项
更多精彩内容请关注微信公众号:听潮庭。 第一题:删除排序数组中的重复项 给你一个 非严格递增排列 的数组 nums ,请你 原地 删除重复出现的元素,使每个元素 只出现一次 ,返回删除后数组的新长度。元素的 相对顺序 应…...
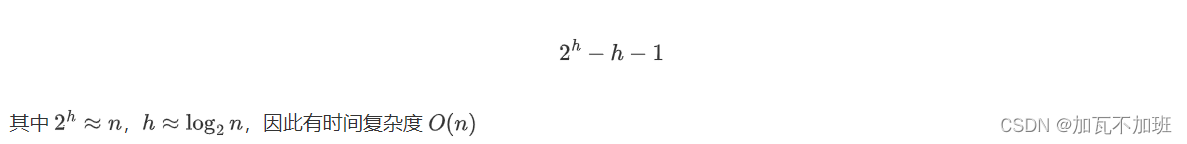
堆的初步认识
在学习本节文章前要先了解:大顶堆与小顶堆: (优先级队列_加瓦不加班的博客-CSDN博客) 堆实现 计算机科学中,堆是一种基于树的数据结构,通常用完全二叉树实现。 什么叫完全二叉树? 答&#x…...

CycleGAN模型之Pytorch实战
一、CycleGAN基本介绍 1. CycleGAN论文:《Unpaired Image-to-Image Translation using Cycle-Consistent Adversarial Networks》 2. 原文代码:https://github.com/junyanz/pytorch-CycleGAN-and-pix2pix 3. 网传精简代码:https://github.com/aitorzip/PyTorch-CycleGAN …...
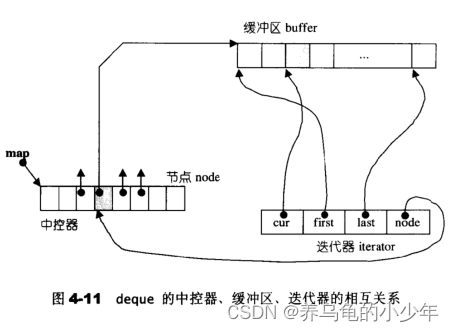
C++(STL容器适配器)
前言: 适配器也称配接器(adapters)在STL组件的灵活组合运用功能上,扮演着轴承、转换器的角色。 《Design Patterns》对adapter的定义如下:将一个class的接口转换为另一个class的接口,使原本因接口不兼容而…...
)
软考 系统架构设计师系列知识点之软件架构风格(7)
接前一篇文章:软考 系统架构设计师系列知识点之软件架构风格(6) 这个十一注定是一个不能放松、保持“紧”的十一。由于报名了全国计算机技术与软件专业技术资格(水平)考试,11月4号就要考试,因此…...

【Vue3】自定义指令
除了 Vue 内置的一系列指令 (比如 v-model 或 v-show) 之外,Vue 还允许你注册自定义的指令 (Custom Directives)。 1. 生命周期钩子函数 一个自定义指令由一个包含类似组件生命周期钩子的对象来定义。钩子函数会接收到指令所绑定元素作为其参数。 在 <script …...

UG\NX CAM二次开发 加工模块获取 UF _ask_application_module
文章作者:代工 来源网站:NX CAM二次开发专栏 简介: UG\NX CAM二次开发 加工模块获取 UF _ask_application_module 代码: void MyClass::do_it() { // TODO: add your code here // 获取NX当前所在的模块 int module_id = 0; // UF_ask_application_module(&…...

借助GPU算力编译Android
借助GPU算力编译Android 借助GPU编译Android代码的意义在于提高编译的效率和速度。传统的CPU编译方式在处理大量代码时可能会遇到性能瓶颈,而GPU编译利用了显卡的并行计算能力,可以同时处理多个任务,加快编译过程。通过利用GPU的并行计算能力,可以将编译过程中的多个任务分…...

手游刚开服就被攻击怎么办?如何防御DDoS?
开服初期是手游最脆弱的阶段,极易成为DDoS攻击的目标。一旦遭遇攻击,可能导致服务器瘫痪、玩家流失,甚至造成巨大经济损失。本文为开发者提供一套简洁有效的应急与防御方案,帮助快速应对并构建长期防护体系。 一、遭遇攻击的紧急应…...
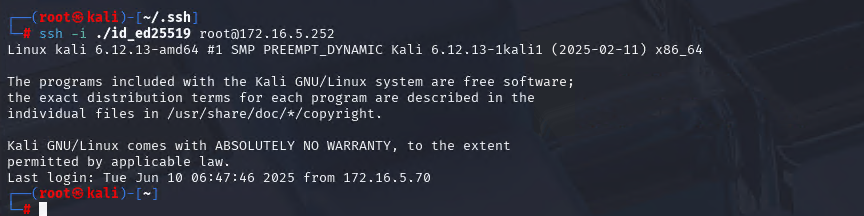
Xshell远程连接Kali(默认 | 私钥)Note版
前言:xshell远程连接,私钥连接和常规默认连接 任务一 开启ssh服务 service ssh status //查看ssh服务状态 service ssh start //开启ssh服务 update-rc.d ssh enable //开启自启动ssh服务 任务二 修改配置文件 vi /etc/ssh/ssh_config //第一…...
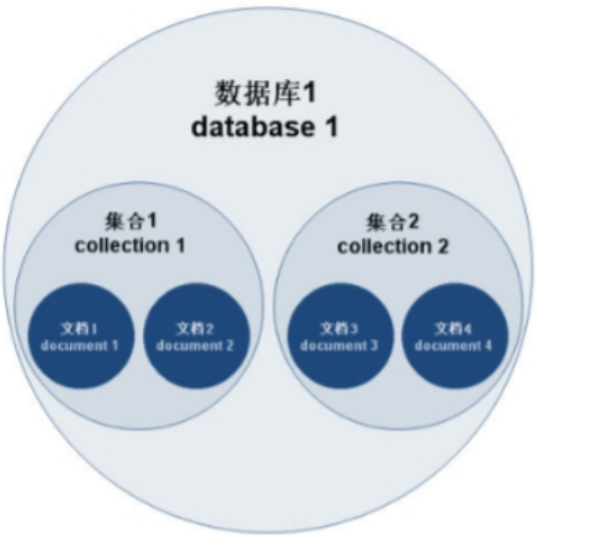
MongoDB学习和应用(高效的非关系型数据库)
一丶 MongoDB简介 对于社交类软件的功能,我们需要对它的功能特点进行分析: 数据量会随着用户数增大而增大读多写少价值较低非好友看不到其动态信息地理位置的查询… 针对以上特点进行分析各大存储工具: mysql:关系型数据库&am…...

QMC5883L的驱动
简介 本篇文章的代码已经上传到了github上面,开源代码 作为一个电子罗盘模块,我们可以通过I2C从中获取偏航角yaw,相对于六轴陀螺仪的yaw,qmc5883l几乎不会零飘并且成本较低。 参考资料 QMC5883L磁场传感器驱动 QMC5883L磁力计…...
Docker 运行 Kafka 带 SASL 认证教程
Docker 运行 Kafka 带 SASL 认证教程 Docker 运行 Kafka 带 SASL 认证教程一、说明二、环境准备三、编写 Docker Compose 和 jaas文件docker-compose.yml代码说明:server_jaas.conf 四、启动服务五、验证服务六、连接kafka服务七、总结 Docker 运行 Kafka 带 SASL 认…...
srs linux
下载编译运行 git clone https:///ossrs/srs.git ./configure --h265on make 编译完成后即可启动SRS # 启动 ./objs/srs -c conf/srs.conf # 查看日志 tail -n 30 -f ./objs/srs.log 开放端口 默认RTMP接收推流端口是1935,SRS管理页面端口是8080,可…...

LLM基础1_语言模型如何处理文本
基于GitHub项目:https://github.com/datawhalechina/llms-from-scratch-cn 工具介绍 tiktoken:OpenAI开发的专业"分词器" torch:Facebook开发的强力计算引擎,相当于超级计算器 理解词嵌入:给词语画"…...
k8s业务程序联调工具-KtConnect
概述 原理 工具作用是建立了一个从本地到集群的单向VPN,根据VPN原理,打通两个内网必然需要借助一个公共中继节点,ktconnect工具巧妙的利用k8s原生的portforward能力,简化了建立连接的过程,apiserver间接起到了中继节…...

【学习笔记】深入理解Java虚拟机学习笔记——第4章 虚拟机性能监控,故障处理工具
第2章 虚拟机性能监控,故障处理工具 4.1 概述 略 4.2 基础故障处理工具 4.2.1 jps:虚拟机进程状况工具 命令:jps [options] [hostid] 功能:本地虚拟机进程显示进程ID(与ps相同),可同时显示主类&#x…...

C#中的CLR属性、依赖属性与附加属性
CLR属性的主要特征 封装性: 隐藏字段的实现细节 提供对字段的受控访问 访问控制: 可单独设置get/set访问器的可见性 可创建只读或只写属性 计算属性: 可以在getter中执行计算逻辑 不需要直接对应一个字段 验证逻辑: 可以…...
