GSVA -- 学习记录
文章目录
- 1.原理简介
- 2. 注意事项
- 3. 功能实现
- 代码实现部分
- 4.可视化
- 5.与GSEA比较
1.原理简介
Gene Set Variation Analysis (GSVA) 基因集变异分析。可以简单认为是样本数据中的基因根据表达量排序后形成了一个rank list,这个rank list 与 预设的gene sets(a set of genes forming a pathway or some other functional unit)进行比较,用KS test计算分布差异(GSVA enrichment score is either the difference between the two sums or the maximum deviation from zero),最大差异作为enrichment score。
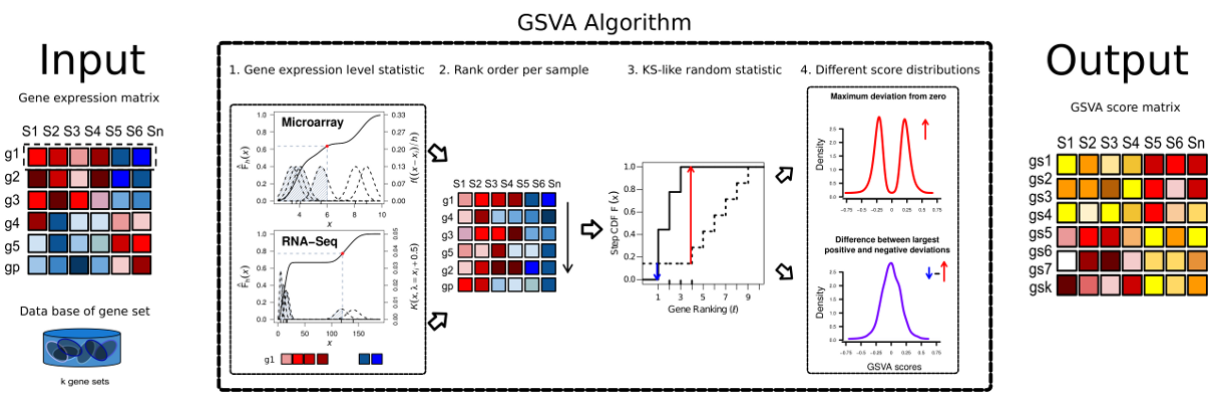
算法主要做了四件事:
1.Kernel estimation of the cumulative density function (kcdf). 实现基因表达量的非参估计,使得样本间基因表达量高低可比。或者就简单认为是一种 rank,只不过rank方法是KS分布计算出来的数值决定的。
2.The expression-level statistic is rank ordered for each sample。同上述,进行样本内gene rank
3.For every gene set, the Kolmogorov-Smirnov-like rank statistic is calculated. rank 后的样本gene set 与预设的gene set 计算分布差异。
4.The GSVA enrichment score is either the difference between the two sums or the maximum deviation from zero. 将第三步计算的分布差异转换成 GSVA enrichment score。
2. 注意事项
-
基因表达量的数值影响gsva的参数设置:
while microarray data, transform values to log2(values) and set paramseter kcdf=“Poisson”
while RNAseq values of expression data is integer count, set paramseter kcdf=“Poisson”
whlie RNAseq values of expression data is log-CPMs, log-RPKMs or log-TPMs , set paramseter kcdf=“Gaussian” -
gsva 函数执行的默认过滤操作:
- matches gene identifiers between gene sets and gene expression values,gene expression matrix中无法match的gene会被剔除
- it discards gene sets that do not meet minimum and maximumgene set size requirements specified with the arguments min.sz and max.sz 。我没搞清楚这个是样本内的gene set 还是预设的gene set大小不符合设置会被过滤掉。
-
得到GSVA ES后如果想进行差异分析时,该如何操作:
unchanged the default argument mx.diff=TRUE to obtain approximately normally distributed ES
and use limma on the resulting GSVA enrichment scores,finally get plots. -
变异分的计算:
two approaches for turning the KS like random walk statistic into an enrichment statistic (ES) (also called GSVA score), the classical maximum deviation method & a normalized ES。classical maximum deviation method的含义 : the maximum deviation from zero of the random walk of the j-th sample with respect to the k-th gene set (gsva函数参数设置: mx.diff =TRUE)
a normalized ES 的含义: 零假设下(no change in pathway activity throughout the sample population)enrichment scores 应该是个正态分布。(个人还是觉得第一种算分方法靠谱)(gsva函数参数设置: mx.diff =FALSE)
3. 功能实现
大致可以实现如下两个目的:
- 单样本内的gene sets 识别,也可以认为得到 gene signatures。
- 多样本ES 比较,识别差异 gene sets,然后聚类,根据gene sets标识样本生物学属性。(单细胞分析中有些人就是这样干的,他们不是基于图的聚类方法 去细胞聚类然后再找DE,最后注释细胞类群。他们让每个细胞与预设的gene sets比较得到 GSVA ES,然后根据ES聚类,从而划分出来细胞类群,然后说这类细胞特化/极化出了啥表型,有啥用,巴拉巴拉一个故事)。
代码实现部分
# 准备表达矩阵
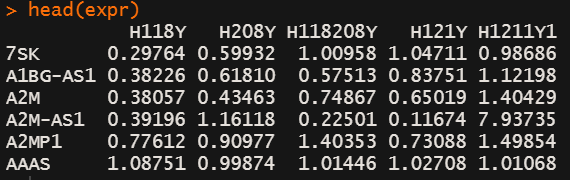
list.files("G:/20240223-project-HY0007-GSVA-analysis-result/")expr <- read.table("../TPM_DE.filter.txt",sep = "\t",header = T,row.names = 1)
head(expr)
expr <- as.matrix(expr) # 需要转换成matrix或者 ExpressionSet object
# 准备预设的gene sets
# install.packages("msigdbr")
library(msigdbr)
## msigdbr包提取下载 先试试KEGG和GO做GSVA分析
##KEGG
KEGG_df_all <- msigdbr(species = "Homo sapiens", # Homo sapiens or Mus musculuscategory = "C2",subcategory = "CP:KEGG")
KEGG_df <- dplyr::select(KEGG_df_all,gs_name,gs_exact_source,gene_symbol)
kegg_list <- split(KEGG_df$gene_symbol, KEGG_df$gs_name) ##按照gs_name给gene_symbol分组##GO
GO_df_all <- msigdbr(species = "Homo sapiens",category = "C5")
GO_df <- dplyr::select(GO_df_all, gs_name, gene_symbol, gs_exact_source, gs_subcat)
GO_df <- GO_df[GO_df$gs_subcat!="HPO",]
go_list <- split(GO_df$gene_symbol, GO_df$gs_name) ##按照gs_name给gene_symbol分组## manual operation
list.files("../")
library(GSEABase) # 读取 Gmt文件myself_geneset <- getGmt("../my_signature.symbols.gmt.txt")
#### GSVA ####
# geneset 1
geneset <- go_list
gsva_mat <- gsva(expr=expr, gset.idx.list=geneset, kcdf="Gaussian" ,#"Gaussian" for logCPM,logRPKM,logTPM, "Poisson" for countsverbose=T, mx.diff =TRUE,# 下游做limma得到差异通路min.sz = 10 # gene sets 少于10个gene的过滤掉)write.csv(gsva_mat,"gsva_go_matrix.csv")# geneset 2
geneset <- kegg_list
gsva_mat <- gsva(expr=expr, gset.idx.list=geneset, kcdf="Gaussian" ,#"Gaussian" for logCPM,logRPKM,logTPM, "Poisson" for countsverbose=T, mx.diff =TRUE,# 下游做limma得到差异通路min.sz = 10 # gene sets 少于10个gene的过滤掉
)write.csv(gsva_mat,"gsva_kegg_matrix.csv")# geneset 3
geneset <- myself_geneset
gsva_mat <- gsva(expr=expr, gset.idx.list=geneset, kcdf="Gaussian" ,#"Gaussian" for logCPM,logRPKM,logTPM, "Poisson" for countsverbose=T, mx.diff =TRUE,# 下游做limma得到差异通路min.sz = 10 # gene sets 少于10个gene的过滤掉
)write.csv(gsva_mat,"gsva_myself_matrix.csv")
# 拿到结果后做可视化分析 一般是火山图和柱状偏差图或者热图
4.可视化
# 先整体来张热图
pheatmap::pheatmap(gsva_mat,cluster_rows = T,show_rownames = F,cluster_cols = F)
# KEGG条目或者GO条目太多了,做个差异分析过滤掉一些不显著差异的
#### 进行limma差异处理 ####
##设定 实验组exp / 对照组ctr
# 不需要进行 limma-trend 或 voom的步骤
library(limma)group_list <- colnames(expr)
design <- model.matrix(~0+factor(group_list))
designcolnames(design) <- levels(factor(group_list))
rownames(design) <- colnames(gsva_mat)
contrast.matrix <- makeContrasts(contrasts=paste0(exp,'-',ctr), #"exp/ctrl"levels = design)fit1 <- lmFit(gsva_mat,design) #拟合模型
fit2 <- contrasts.fit(fit1, contrast.matrix) #统计检验
efit <- eBayes(fit2) #修正summary(decideTests(efit,lfc=1, p.value=0.05)) #统计查看差异结果
tempOutput <- topTable(efit, coef=paste0(exp,'-',ctr), n=Inf)
degs <- na.omit(tempOutput)
write.csv(degs,"gsva_go_degs.results.csv")#### 对GSVA的差异分析结果进行热图可视化 ####
##设置筛选阈值
padj_cutoff=0.05
log2FC_cutoff=log2(2)keep <- rownames(degs[degs$adj.P.Val < padj_cutoff & abs(degs$logFC)>log2FC_cutoff, ])
length(keep)
dat <- gsva_mat[keep[1:50],] #选取前50进行展示degs$significance <- as.factor(ifelse(degs$adj.P.Val < padj_cutoff & abs(degs$logFC) > log2FC_cutoff,ifelse(degs$logFC > log2FC_cutoff ,'UP','DOWN'),'NOT'))# 火山图
this_title <- paste0(' Up : ',nrow(degs[degs$significance =='UP',]) ,'\n Down : ',nrow(degs[degs$significance =='DOWN',]),'\n adj.P.Val <= ',padj_cutoff,'\n FoldChange >= ',round(2^log2FC_cutoff,3))g <- ggplot(data=degs, aes(x=logFC, y=-log10(adj.P.Val),color=significance)) +#点和背景geom_point(alpha=0.4, size=1) +theme_classic()+ #无网格线#坐标轴xlab("log2 ( FoldChange )") + ylab("-log10 ( adj.P.Val )") +#标题文本ggtitle( this_title ) +#分区颜色 scale_colour_manual(values = c('blue','grey','red'))+ #辅助线geom_vline(xintercept = c(-log2FC_cutoff,log2FC_cutoff),lty=4,col="grey",lwd=0.8) +geom_hline(yintercept = -log10(padj_cutoff),lty=4,col="grey",lwd=0.8) +#图例标题间距等设置theme(plot.title = element_text(hjust = 0.5), plot.margin=unit(c(2,2,2,2),'lines'), #上右下左legend.title = element_blank(),legend.position="right")ggsave(g,filename = 'GSVA_go_volcano_padj.pdf',width =8,height =7.5)#### 发散条形图绘制 ####
# install.packages("ggthemes")
# install.packages("ggprism")
library(ggthemes)
library(ggprism)p_cutoff=0.001
degs <- gsva_kegg_degs #载入gsva的差异分析结果
Diff <- rbind(subset(degs,logFC>0)[1:20,], subset(degs,logFC<0)[1:20,]) #选择上下调前20通路
dat_plot <- data.frame(id = row.names(Diff),p = Diff$P.Value,lgfc= Diff$logFC)
dat_plot$group <- ifelse(dat_plot$lgfc>0 ,1,-1) # 将上调设为组1,下调设为组-1
dat_plot$lg_p <- -log10(dat_plot$p)*dat_plot$group # 将上调-log10p设置为正,下调-log10p设置为负
dat_plot$id <- str_replace(dat_plot$id, "KEGG_","");dat_plot$id[1:10]
dat_plot$threshold <- factor(ifelse(abs(dat_plot$p) <= p_cutoff,ifelse(dat_plot$lgfc >0 ,'Up','Down'),'Not'),levels=c('Up','Down','Not'))dat_plot <- dat_plot %>% arrange(lg_p)
dat_plot$id <- factor(dat_plot$id,levels = dat_plot$id)## 设置不同标签数量
low1 <- dat_plot %>% filter(lg_p < log10(p_cutoff)) %>% nrow()
low0 <- dat_plot %>% filter(lg_p < 0) %>% nrow()
high0 <- dat_plot %>% filter(lg_p < -log10(p_cutoff)) %>% nrow()
high1 <- nrow(dat_plot)p <- ggplot(data = dat_plot,aes(x = id, y = lg_p, fill = threshold)) +geom_col()+coord_flip() + scale_fill_manual(values = c('Up'= '#36638a','Not'='#cccccc','Down'='#7bcd7b')) +geom_hline(yintercept = c(-log10(p_cutoff),log10(p_cutoff)),color = 'white',size = 0.5,lty='dashed') +xlab('') + ylab('-log10(P.Value) of GSVA score') + guides(fill="none")+theme_prism(border = T) +theme(axis.text.y = element_blank(),axis.ticks.y = element_blank()) +geom_text(data = dat_plot[1:low1,],aes(x = id,y = 0.1,label = id),hjust = 0,color = 'black') + #黑色标签geom_text(data = dat_plot[(low1 +1):low0,],aes(x = id,y = 0.1,label = id),hjust = 0,color = 'grey') + # 灰色标签geom_text(data = dat_plot[(low0 + 1):high0,],aes(x = id,y = -0.1,label = id),hjust = 1,color = 'grey') + # 灰色标签geom_text(data = dat_plot[(high0 +1):high1,],aes(x = id,y = -0.1,label = id),hjust = 1,color = 'black') # 黑色标签ggsave("GSVA_barplot_pvalue.pdf",p,width = 15,height = 15)
5.与GSEA比较
GSEA与GSVA 计算enrichment scores 方法类似,都是一个gene list 然后和预设的gene sets 比较,计算两者的距离。
距离计算公式都是max distance from zero of KS test.
不同的是GSEA得到的gene list 一般是根据样本间的logFC或者ratio of signal to noise 或者 样本内的relative expression,排序得到 sorted gene list 再与gene sets 计算 enrichment scores。
GSVA 得到gene list 的方法是根据kcdf累积分布计算样本内gene relative expression,然后排序得到 sorted gene list 再与gene sets 计算 enrichment scores。
相关文章:

GSVA -- 学习记录
文章目录 1.原理简介2. 注意事项3. 功能实现代码实现部分 4.可视化5.与GSEA比较 1.原理简介 Gene Set Variation Analysis (GSVA) 基因集变异分析。可以简单认为是样本数据中的基因根据表达量排序后形成了一个rank list,这个rank list 与 预设的gene setsÿ…...

基于Springboot的旅游网管理系统设计与实现(有报告)。Javaee项目,springboot项目。
演示视频: 基于Springboot的旅游网管理系统设计与实现(有报告)。Javaee项目,springboot项目。 项目介绍: 采用M(model)V(view)C(controller)三层…...
Docker基础篇(六) dockerfile体系结构语法
FROM:基础镜像,当前新镜像是基于哪个镜像的 MAINTAINER :镜像维护者的姓名和邮箱地址 RUN:容器构建时需要运行的命令 EXPOSE :当前容器对外暴露出的端口号 WORKDIR:指定在创建容器后,终端默认登…...

【Python编程+数据清洗+Pandas库+数据分析】
数据分析的第一步往往是数据清洗,这个过程关键在于理解、整理和清洗原始数据,为进一步分析做好准备。Python 语言通过Pandas库提供了一系列高效的数据清洗工具。接下来,该文章将通过一个简单的案例演示如何利用 Pandas 进行数据清洗ÿ…...

网络安全之防御保护8 - 11 天笔记
一、内容安全 1、攻击可能只是一个点,防御需要全方面进行 2、IAE引擎 3、DFI和DPI技术 --- 深度检测技术 深度行为检测技术分为:深度包检测技术(DPI)、深度流检测技术(DFI) DPI --- 深度包检测技术 --- 主要针对完整的数据包…...
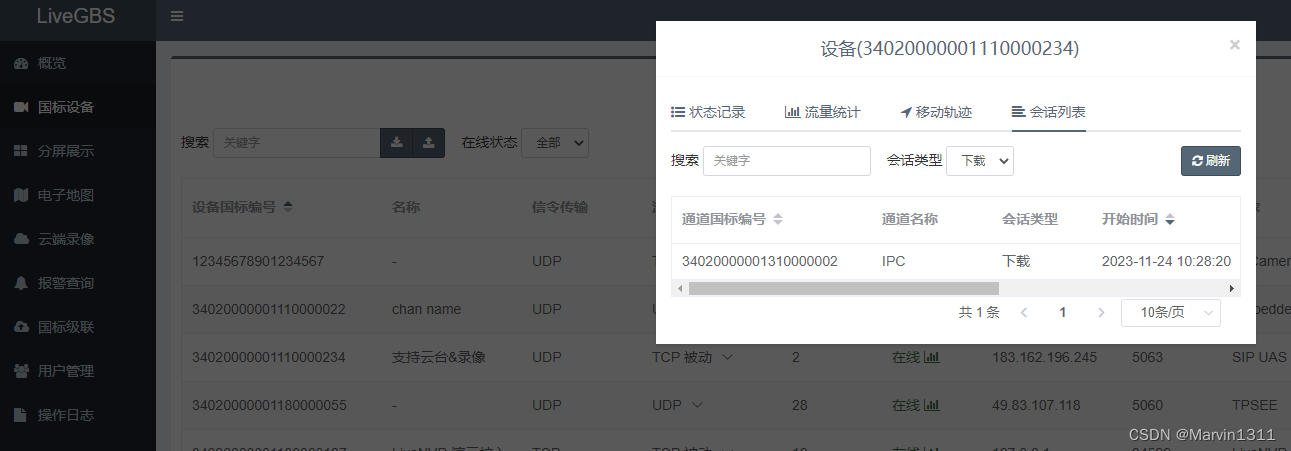
LiveGBS流媒体平台GB/T28181功能-查看国标设备下通道会话列表直播|回放|对讲|播放|录像|级联UDP|TCP|H264|H265会话
LiveGBS流媒体平台GB/T28181功能-查看直播|回放|对讲|播放|录像|级联UDP|TCP|H264|H265会话 1、会话列表2、会话类型3、搭建GB28181视频直播平台 1、会话列表 LiveGBS-> 国标设备-》点击在线状态 点击会话列表 2、会话类型 下拉会话类型可以看到 直播会话、回放会话、下载…...

Python和Jupyter简介
在本notebook中,你将: 1、学习如何使用一个Jupyter notebook 2、快速学习Python语法和科学库 3、学习一些IPython特性,我们将在之后教程中使用。 这是什么? 这是只为你运行在一个个人"容器"中的一个Jupyter noteboo…...
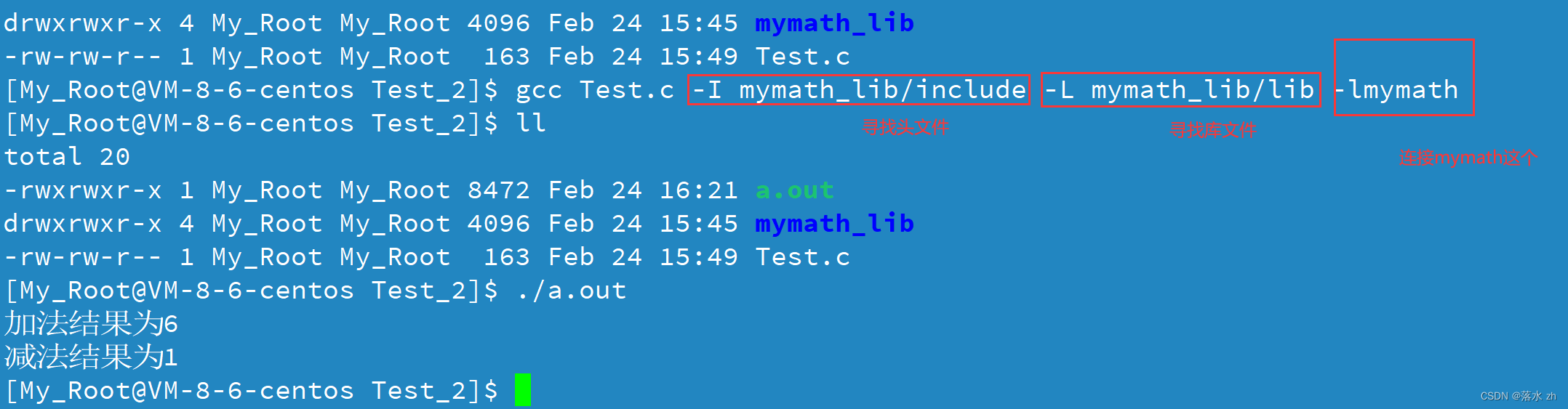
Linux——静态库
Linux——静态库 静态库分析一下 ar指令生成静态库静态库的使用第三方库优化一下 gcc -I(大写的i) -L -l(小写的l),头文件搜索路径,库文件搜索路径,连接库 今天我们来学习静态库的基本知识。 静态库 在了解静态库之前,我们首先来…...
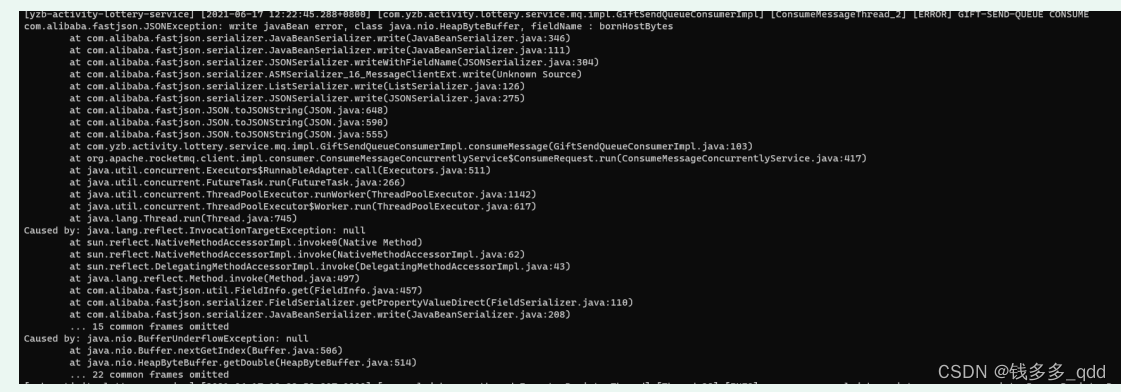
fastjson序列化MessageExt对象问题(1.2.78之前版本)
前言 无论是kafka,还是RocketMq,消费者方法参数中的MessageExt对象不能被 fastjson默认的方式序列化。 一、查看代码 Override public ConsumeConcurrentlyStatus consumeMessage(List<MessageExt> msgs,ConsumeConcurrentlyContext context) {t…...
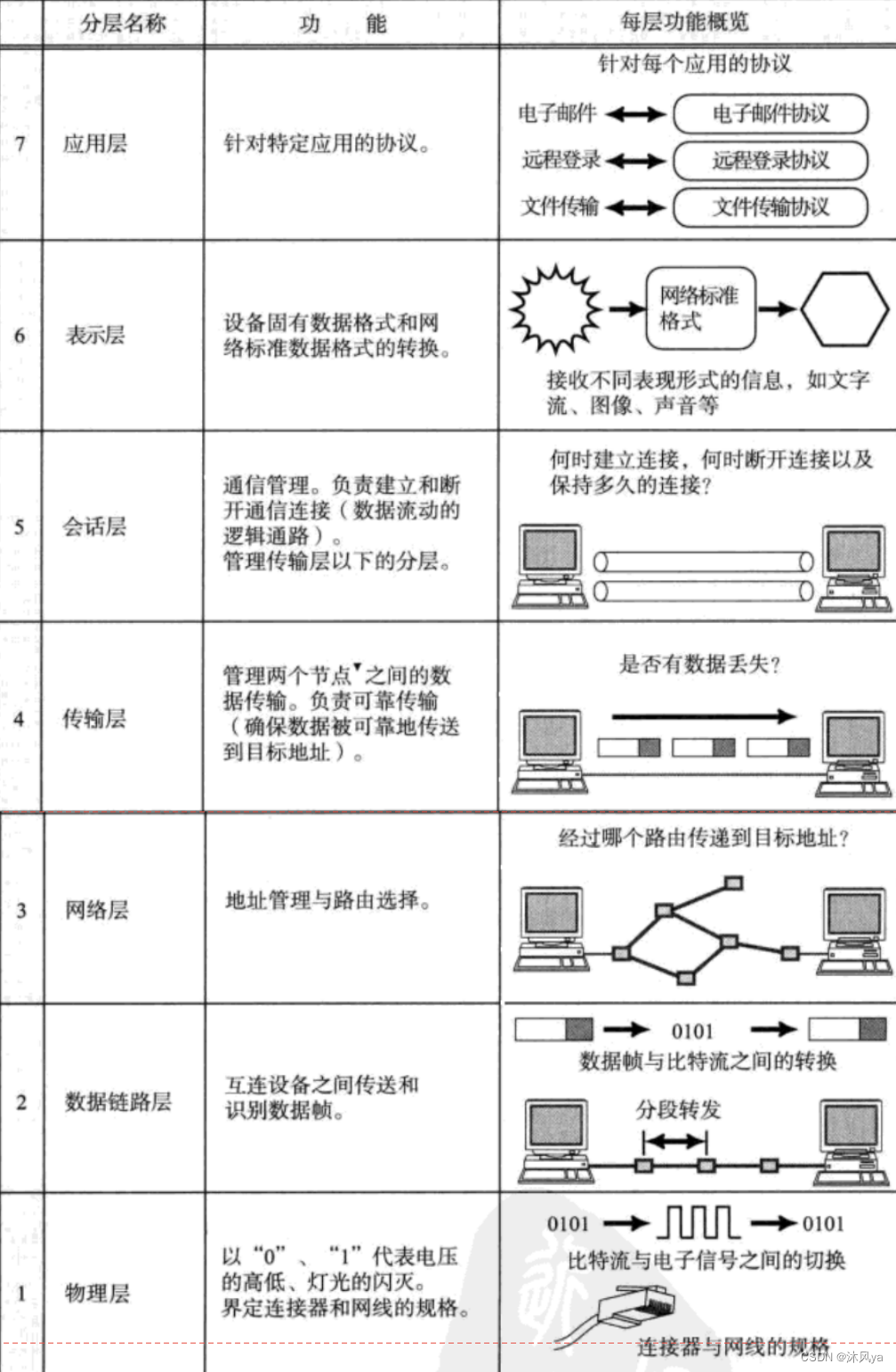
osi模型,tcp/ip模型(名字由来+各层介绍+中间设备介绍)
目录 网络协议如何分层 引入 osi模型 tcp/ip模型 引入 命名由来 介绍 物理层 数据链路层 网络层 传输层 应用层 中间设备 网络协议如何分层 引入 我们已经知道了网络协议是层状结构,接下来就来了解了解下网络协议如何分层 常见的网络协议分层模型是OSI模型 和 …...

ElasticSearch之找到乔丹的空中大灌篮电影
写在前面 本文看一个搜索的实际例子,找到篮球之神乔丹的电影Space Jam,即空中大灌篮。 正式开始之前先来看下要查询的目标文档,以及查询的text: 要查询的目标文档 {..."title": "Space Jam",..."ove…...
)
CSS @符规则(@font-face、@keyframes、@media、@scope等)
随着前端开发的不断发展,CSS 的功能日益强大,其中 规则扮演着举足轻重的角色。它们不仅扩展了 CSS 的功能边界,还为开发者提供了更加灵活和高效的样式定义方式,让我们来一同探索这些强大而实用的 规则吧! font-face …...

uniapp微信小程序解决上方刘海屏遮挡
问题 在有刘海屏的手机上,我们的文字和按钮等可能会被遮挡 应该避免这种情况 解决 const SYSTEM_INFO uni.getSystemInfoSync();export const getStatusBarHeight ()> SYSTEM_INFO.statusBarHeight || 15;export const getTitleBarHeight ()>{if(uni.get…...
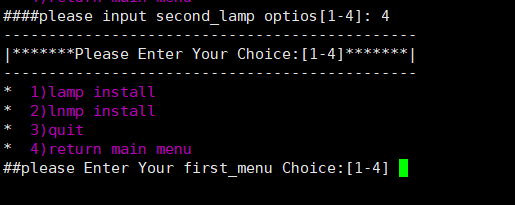
项目:shell实现多级菜单脚本编写
目录 1. 提示 2. 演示效果 2.1. 一级菜单 2.2. 二级菜单 2.3. 执行操作 3. 参考代码 1. 提示 本脚本主要实现多级菜单效果,并没有安装LAMP、LNMP环境,如果要用在实际生成环境中部署LNMP、LAMP环境,只需要简单修改一下就可以了。 2. 演…...
)
Collections常用方法(Java)
Collections常用方法 使用 sort(List<T> list) 对 List 进行排序: List<Integer> numbers new ArrayList<>(Arrays.asList(3, 1, 4, 1, 5, 9, 2, 6)); Collections.sort(numbers); System.out.println("排序后的列表:" …...

Mysql整理-概述
Mysql概述 MySQL是一种流行的开源关系数据库管理系统(RDBMS),它使用结构化查询语言(SQL)来访问、管理和处理数据。它是基于客户端-服务器模型的数据库,意味着数据存储在服务器上,而用户可以通过客户端软件从不同的位置访问这些数据。 MySQL的主要特点包括: 开源软件:M…...

ubuntu+QT+ OpenGL环境搭建和绘图
一,安装OpenGL库 安装OpenGL依赖项:运行sudo apt install libgl1-mesa-glx命令安装OpenGL所需的一些依赖项。 安装OpenGL头文件:运行sudo apt install libgl1-mesa-dev命令来安装OpenGL的头文件。 安装GLUT库:GLUT(Ope…...
)
Vue实现打印功能(vue-print-nb)
1、安装依赖 npm install vue-print-nb --save2、在main.js中引入 import Print from vue-print-nb Vue.use(Print)3、在组件的打印区域标签上加 id“printArea” <div id"printArea"> 打印区域 </div>4、在组件的打印按钮标签上使用指令 v-print“pr…...
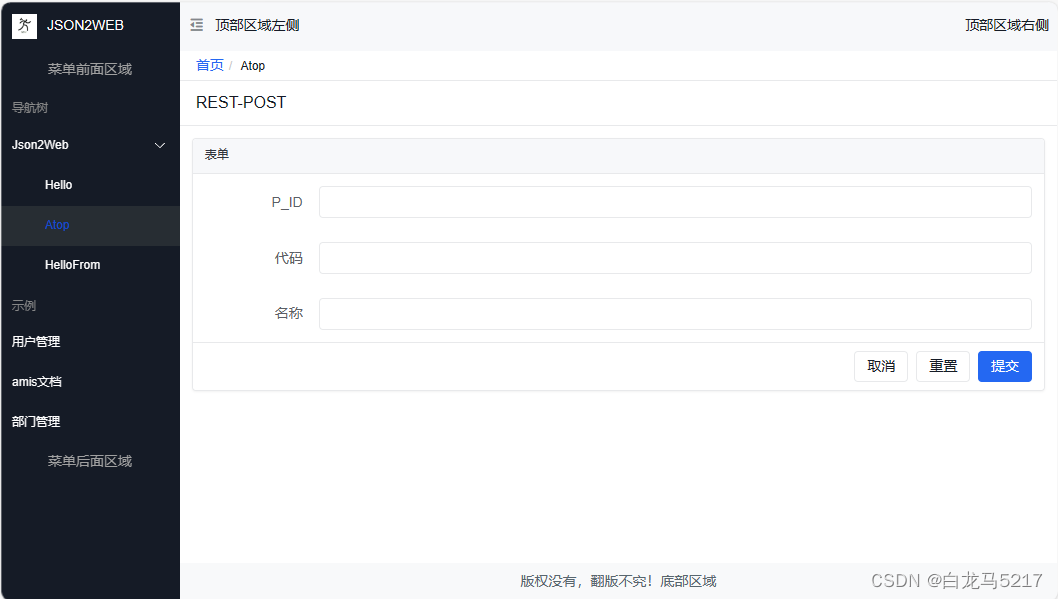
【JSON2WEB】06 JSON2WEB前端框架搭建
【JSON2WEB】01 WEB管理信息系统架构设计 【JSON2WEB】02 JSON2WEB初步UI设计 【JSON2WEB】03 go的模板包html/template的使用 【JSON2WEB】04 amis低代码前端框架介绍 【JSON2WEB】05 前端开发三件套 HTML CSS JavaScript 速成 前端技术路线太多了,知识点更多&…...
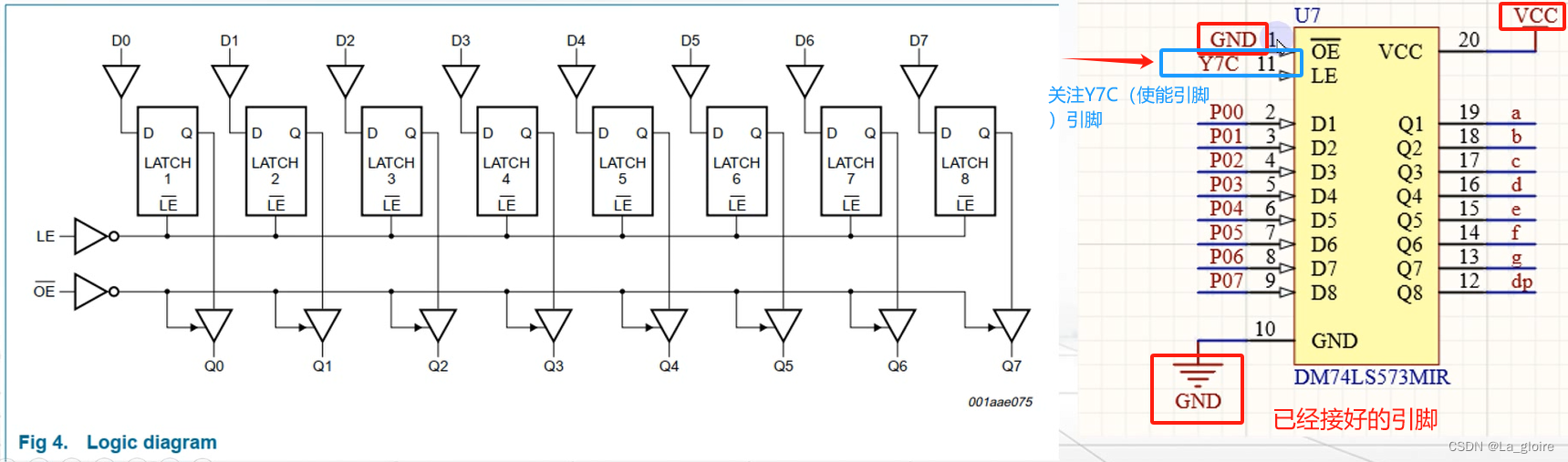
【蓝桥杯单片机入门记录】动态数码管
目录 一、数码管动态显示概述 二、动态数码管原理图 (1)原理图 (2)动态数码管如何与芯片相连 (3)“此器件” ——>锁存器74HC573 三、动态数码管显示例程 (1)例程1…...
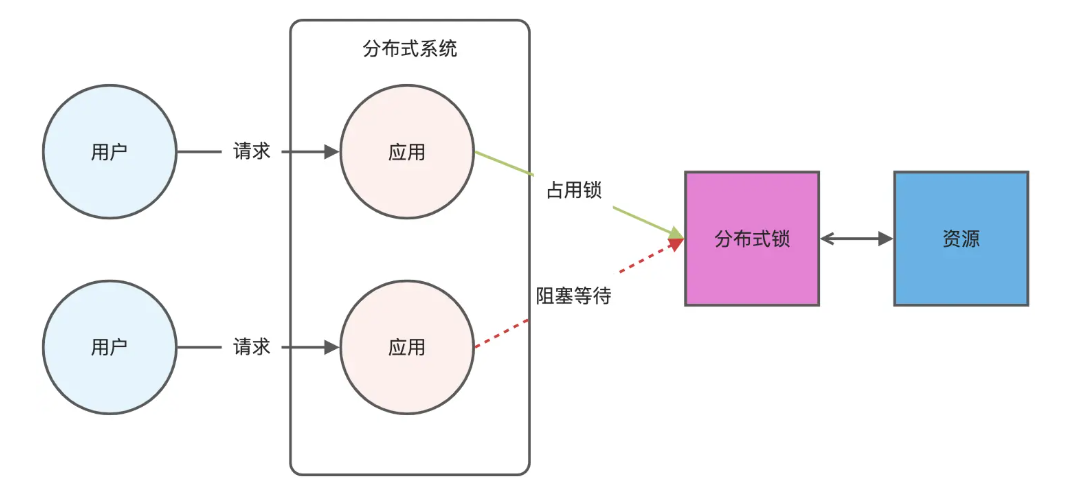
Redis相关知识总结(缓存雪崩,缓存穿透,缓存击穿,Redis实现分布式锁,如何保持数据库和缓存一致)
文章目录 1.什么是Redis?2.为什么要使用redis作为mysql的缓存?3.什么是缓存雪崩、缓存穿透、缓存击穿?3.1缓存雪崩3.1.1 大量缓存同时过期3.1.2 Redis宕机 3.2 缓存击穿3.3 缓存穿透3.4 总结 4. 数据库和缓存如何保持一致性5. Redis实现分布式…...
【第二十一章 SDIO接口(SDIO)】
第二十一章 SDIO接口 目录 第二十一章 SDIO接口(SDIO) 1 SDIO 主要功能 2 SDIO 总线拓扑 3 SDIO 功能描述 3.1 SDIO 适配器 3.2 SDIOAHB 接口 4 卡功能描述 4.1 卡识别模式 4.2 卡复位 4.3 操作电压范围确认 4.4 卡识别过程 4.5 写数据块 4.6 读数据块 4.7 数据流…...

Java - Mysql数据类型对应
Mysql数据类型java数据类型备注整型INT/INTEGERint / java.lang.Integer–BIGINTlong/java.lang.Long–––浮点型FLOATfloat/java.lang.FloatDOUBLEdouble/java.lang.Double–DECIMAL/NUMERICjava.math.BigDecimal字符串型CHARjava.lang.String固定长度字符串VARCHARjava.lang…...

Spring Boot+Neo4j知识图谱实战:3步搭建智能关系网络!
一、引言 在数据驱动的背景下,知识图谱凭借其高效的信息组织能力,正逐步成为各行业应用的关键技术。本文聚焦 Spring Boot与Neo4j图数据库的技术结合,探讨知识图谱开发的实现细节,帮助读者掌握该技术栈在实际项目中的落地方法。 …...
Mobile ALOHA全身模仿学习
一、题目 Mobile ALOHA:通过低成本全身远程操作学习双手移动操作 传统模仿学习(Imitation Learning)缺点:聚焦与桌面操作,缺乏通用任务所需的移动性和灵活性 本论文优点:(1)在ALOHA…...

Java + Spring Boot + Mybatis 实现批量插入
在 Java 中使用 Spring Boot 和 MyBatis 实现批量插入可以通过以下步骤完成。这里提供两种常用方法:使用 MyBatis 的 <foreach> 标签和批处理模式(ExecutorType.BATCH)。 方法一:使用 XML 的 <foreach> 标签ÿ…...

Webpack性能优化:构建速度与体积优化策略
一、构建速度优化 1、升级Webpack和Node.js 优化效果:Webpack 4比Webpack 3构建时间降低60%-98%。原因: V8引擎优化(for of替代forEach、Map/Set替代Object)。默认使用更快的md4哈希算法。AST直接从Loa…...

Redis:现代应用开发的高效内存数据存储利器
一、Redis的起源与发展 Redis最初由意大利程序员Salvatore Sanfilippo在2009年开发,其初衷是为了满足他自己的一个项目需求,即需要一个高性能的键值存储系统来解决传统数据库在高并发场景下的性能瓶颈。随着项目的开源,Redis凭借其简单易用、…...
淘宝扭蛋机小程序系统开发:打造互动性强的购物平台
淘宝扭蛋机小程序系统的开发,旨在打造一个互动性强的购物平台,让用户在购物的同时,能够享受到更多的乐趣和惊喜。 淘宝扭蛋机小程序系统拥有丰富的互动功能。用户可以通过虚拟摇杆操作扭蛋机,实现旋转、抽拉等动作,增…...
【LeetCode】算法详解#6 ---除自身以外数组的乘积
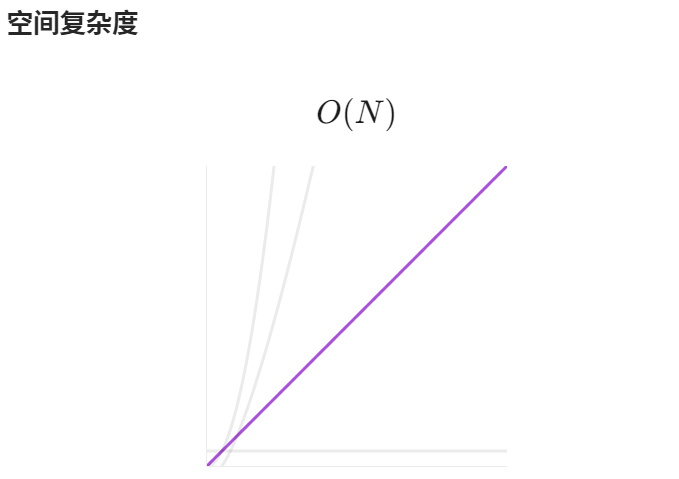
1.题目介绍 给定一个整数数组 nums,返回 数组 answer ,其中 answer[i] 等于 nums 中除 nums[i] 之外其余各元素的乘积 。 题目数据 保证 数组 nums之中任意元素的全部前缀元素和后缀的乘积都在 32 位 整数范围内。 请 不要使用除法,且在 O…...
