生信算法9 - 正则表达式匹配氨基酸序列、核型和字符串
建议在Jupyter实践。
1. 使用正则表达式匹配指定的氨基酸序列
import re# 氨基酸序列
seq = 'VSVLTMFRYAGWLDRLYMLVGTQLAAIIHGVALPLMMLI'# 正则表达式匹配
match = re.search(r'[A|G]W', seq)# 打印match及匹配到开始位置和结束位置
print(match)
# <re.Match object; span=(10, 12), match='GW'>
print(match.start())
print(match.end())if match:# 打印匹配到氨基酸print(match.group())# GW
else:print("no match!")
2. 使用正则表达式查找全部的氨基酸序列
import reseq = 'RQSAMGSNKSKPKDASQRRRSLEPAENVHGAGGGAFPASQRPSKP'# 匹配R开头、第二个氨基酸为任意、第三个氨基酸为S或T、第四个氨基酸不为P的连续4个氨基酸徐磊
matches = re.findall(r'R.[ST][^P]', seq)
print(matches)
# ['RQSA', 'RRSL', 'RPSK']# finditer 匹配对象迭代器
match_iter = re.finditer(r'R.[ST][^P]', seq)# 遍历
for match in match_iter:# 打印group和spanprint(match.group(), match.span())print(match.start(), match.end())# RQSA (0, 4)# 0 4# RRSL (18, 22)# 18 22# RPSK (40, 44)# 40 443. 使用正则表达式匹配多个特殊字符,分割字符串
import re# 匹配特殊字符|和;,并分割字符串
annotation = 'ATOM:CA|RES:ALA|CHAIN:B;NUMRES:166'
split_string = re.split(r'[|;]', annotation)print(split_string)
# ['ATOM:CA', 'RES:ALA', 'CHAIN:B', 'NUMRES:166']4. 正则表达式获取核型染色体数量,区带和CNV大小
karyotype1 = '46,XY; -11{p11.2-p13, 48.32Mb}'
karyotype2 = '47,XXX; +X{+3};-11{p11.2-p13.2, 48.32Mb}'#### 匹配染色体数量 ####
match = re.search(r'(\d+,\w+);', karyotype1)
print(match)
# <re.Match object; span=(0, 6), match='46,XY;'>chr = match.group(1)
print(chr)
# 46,XY#### 匹配染色体开始和结束区带和CNV大小 ####
match2 = re.search(r'([p|q|pter]\d+.?\d+)-([p|q|qter]\d+.?\d+), (\d+.?\d+)Mb', karyotype2)
print(match2)cyto_start = match2.group(1)
cyto_end = match2.group(2)
size = match2.group(3)print(cyto_start)
# p11.2
print(cyto_end)
# p13.2
print(size)
# 48.32
5. 正则表达式获取指定格式的字符串内容
# 结果变异VCF文件描述信息
string = """##ALT=<ID=DEL,Description="Deletion">##ALT=<ID=DUP,Description="Duplication">##ALT=<ID=INV,Description="Inversion">##ALT=<ID=INVDUP,Description="InvertedDUP with unknown boundaries">##ALT=<ID=TRA,Description="Translocation">##ALT=<ID=INS,Description="Insertion">##FILTER=<ID=UNRESOLVED,Description="An insertion that is longer than the read and thus we cannot predict the full size.">##INFO=<ID=CHR2,Number=1,Type=String,Description="Chromosome for END coordinate in case of a translocation">##INFO=<ID=END,Number=1,Type=Integer,Description="End position of the structural variant">##INFO=<ID=MAPQ,Number=1,Type=Integer,Description="Median mapping quality of paired-ends">##INFO=<ID=RE,Number=1,Type=Integer,Description="read support">##INFO=<ID=IMPRECISE,Number=0,Type=Flag,Description="Imprecise structural variation">##INFO=<ID=PRECISE,Number=0,Type=Flag,Description="Precise structural variation">##INFO=<ID=SVLEN,Number=1,Type=Integer,Description="Length of the SV">##INFO=<ID=SVMETHOD,Number=1,Type=String,Description="Type of approach used to detect SV">##INFO=<ID=SVTYPE,Number=1,Type=String,Description="Type of structural variant">##INFO=<ID=SEQ,Number=1,Type=String,Description="Extracted sequence from the best representative read.">##INFO=<ID=STRANDS2,Number=4,Type=Integer,Description="alt reads first + ,alt reads first -,alt reads second + ,alt reads second -.">##INFO=<ID=REF_strand,Number=.,Type=Integer,Description="plus strand ref, minus strand ref.">##INFO=<ID=Strandbias_pval,Number=A,Type=Float,Description="P-value for fisher exact test for strand bias.">##INFO=<ID=STD_quant_start,Number=A,Type=Float,Description="STD of the start breakpoints across the reads.">##INFO=<ID=STD_quant_stop,Number=A,Type=Float,Description="STD of the stop breakpoints across the reads.">##INFO=<ID=Kurtosis_quant_start,Number=A,Type=Float,Description="Kurtosis value of the start breakpoints across the reads.">##INFO=<ID=Kurtosis_quant_stop,Number=A,Type=Float,Description="Kurtosis value of the stop breakpoints across the reads.">##INFO=<ID=SUPTYPE,Number=.,Type=String,Description="Type by which the variant is supported.(SR,AL,NR)">##INFO=<ID=STRANDS,Number=A,Type=String,Description="Strand orientation of the adjacency in BEDPE format (DEL:+-, DUP:-+, INV:++/--)">##INFO=<ID=AF,Number=A,Type=Float,Description="Allele Frequency.">##INFO=<ID=ZMW,Number=A,Type=Integer,Description="Number of ZMWs (Pacbio) supporting SV.">##FORMAT=<ID=GT,Number=1,Type=String,Description="Genotype">##FORMAT=<ID=DR,Number=1,Type=Integer,Description="# high-quality reference reads">##FORMAT=<ID=DV,Number=1,Type=Integer,Description="# high-quality variant reads">"""import re# 创建空dataframe
df_output = pd.DataFrame()list_type = []
list_id = []
list_description = []# 遍历字符串内容,内容拷贝至结构变异VCF文件
for str in string.split('\n'):# 去除末尾\n和字符串内空格str = str.strip().replace(' ', '')# 内容为空或字符串为空则跳过if not str or str == '':continue# 正则表达式匹配##后的英文字符match = re.search(r'##(\w+)', str)type = match.group(1) if match else 'ERORR'# 匹配ID内容match = re.search(r'ID=(\w+)', str)id = match.group(1) if match else 'ERORR'# 匹配Description内容match = re.search(r'Description=\"(.*?)\"', str)description = match.group(1) if match else 'ERORR'# 加入列表list_type.append(type)list_id.append(id)list_description.append(description)print(list_description)
# 加入dataframe
df_output['Type'] = list_type
df_output['ID'] = list_id
df_output['Description'] = list_description# 保存至excel
df_output.to_excel('结构变异描述信息说明.xlsx', index=False)
生信算法文章推荐
生信算法1 - DNA测序算法实践之序列操作
生信算法2 - DNA测序算法实践之序列统计
生信算法3 - 基于k-mer算法获取序列比对索引
生信算法4 - 获取overlap序列索引和序列的算法
生信算法5 - 序列比对之全局比对算法
生信算法6 - 比对reads碱基数量统计及百分比统计
生信算法7 - 核酸序列Fasta和蛋白PDB文件读写与检索
生信算法8 - HGVS转换与氨基酸字母表
相关文章:

生信算法9 - 正则表达式匹配氨基酸序列、核型和字符串
建议在Jupyter实践。 1. 使用正则表达式匹配指定的氨基酸序列 import re# 氨基酸序列 seq VSVLTMFRYAGWLDRLYMLVGTQLAAIIHGVALPLMMLI# 正则表达式匹配 match re.search(r[A|G]W, seq)# 打印match及匹配到开始位置和结束位置 print(match) # <re.Match object; span(10, …...

linux ext2文件系统浅析
文章目录 前言ext2内容概述实验准备二进制对比分析1 super block2 group desc3 block bitmap4 inode bitmap5 inode_tableinode 1inode 2inode 11inode 12 6 dir entry7 data区8 间接块9 块组 前言 网上关于ext2文件系统的博客有很多,但看完之后还是有些云里雾里&a…...

「树莓派入门」树莓派进阶02-传感器应用与交通灯项目
传感器是树莓派实现智能化的关键。通过本教程,你可以开始尝试使用传感器来增强树莓派的功能。 一、传感器在树莓派中的作用 传感器是树莓派与外界环境交互的重要工具。它们可以检测各种物理量,如光、声音、温度等,并将这些物理量转换为电信号,供树莓派读取和处理。 二、数…...

pytorch 指定GPU设备
使用os.environ["CUDA_VISIBLE_DEVICES"] 这种方法是通过环境变量限制可见的CUDA设备,从而在多个GPU的机器上只让PyTorch看到并使用指定的GPU。这种方式的好处是所有后续的CUDA调用都会使用这个GPU,并且代码中不需要显式地指定设备索引。 im…...

华为od-C卷200分题目6 - 5G 网络建设
华为od-C卷200分题目6 - 5G 网络建设 题目描述 现需要在某城市进行 5G 网络建设,已经选取 N 个地点设置 5G 基站,编号固定为 1 到 N,接下来需要各个基站之间使用光纤进行连接以确保基站能互联互通,不同基站之间架设光纤的成本各不…...
步进电机(STM32+28BYJ-48)
一、简介 步进电动机(stepping motor)把电脉冲信号变换成角位移以控制转子转动的执行机构。在自动控制装置中作为执行器。每输入一个脉冲信号,步进电动机前进一步,故又称脉冲电动机。步进电动机多用于数字式计算机的外部设备&…...

Node.js介绍 , 安装与使用
1.Node.js 1 什么是Node.js 官网:https://nodejs.org/zh-cn/ 中文学习网:http://nodejs.cn/learn1.Node.js 是一个基于 Chrome V8 引擎的 JavaScript 运行环境。Node.js 使用了一个事件驱动、非阻塞式 I/O 的模型,使其轻量又高效。 2.前端的底层 html…...
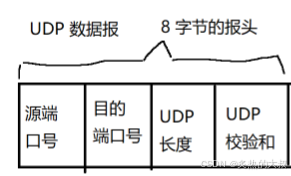
JavaEE初阶-网络原理1
文章目录 前言一、UDP报头二、UDP校验和2.1 CRC2.2 md5 前言 学习一个网络协议,最主要就是学习的报文格式,对于UDP来说,应用层数据到达UDP之后,会给应用层数据报前面加上UDP报头。 UDP数据报UDP包头载荷 一、UDP报头 如上图UDP的…...
)
leetcode秋招冲刺 (专题16--18)
专题16:分治 题目169:多数元素(YES) 解题思路:使用哈希表可以统计出现次数的性质,直接统计就行。 给定一个大小为 n 的数组 nums ,返回其中的多数元素。多数元素是指在数组中出现次数 大于 ⌊…...

学懂C#编程:实用方法——string字符串指定连接符拼接之 string.Join 的详细用法
在C#中,string.Join 方法用于将一个字符串数组或集合中的元素连接成一个单一的字符串,并在每个元素之间插入指定的分隔符。这个方法非常有用,特别是在需要将多个字符串合并成一个字符串时。以下是 string.Join 方法的详细用法: 方…...
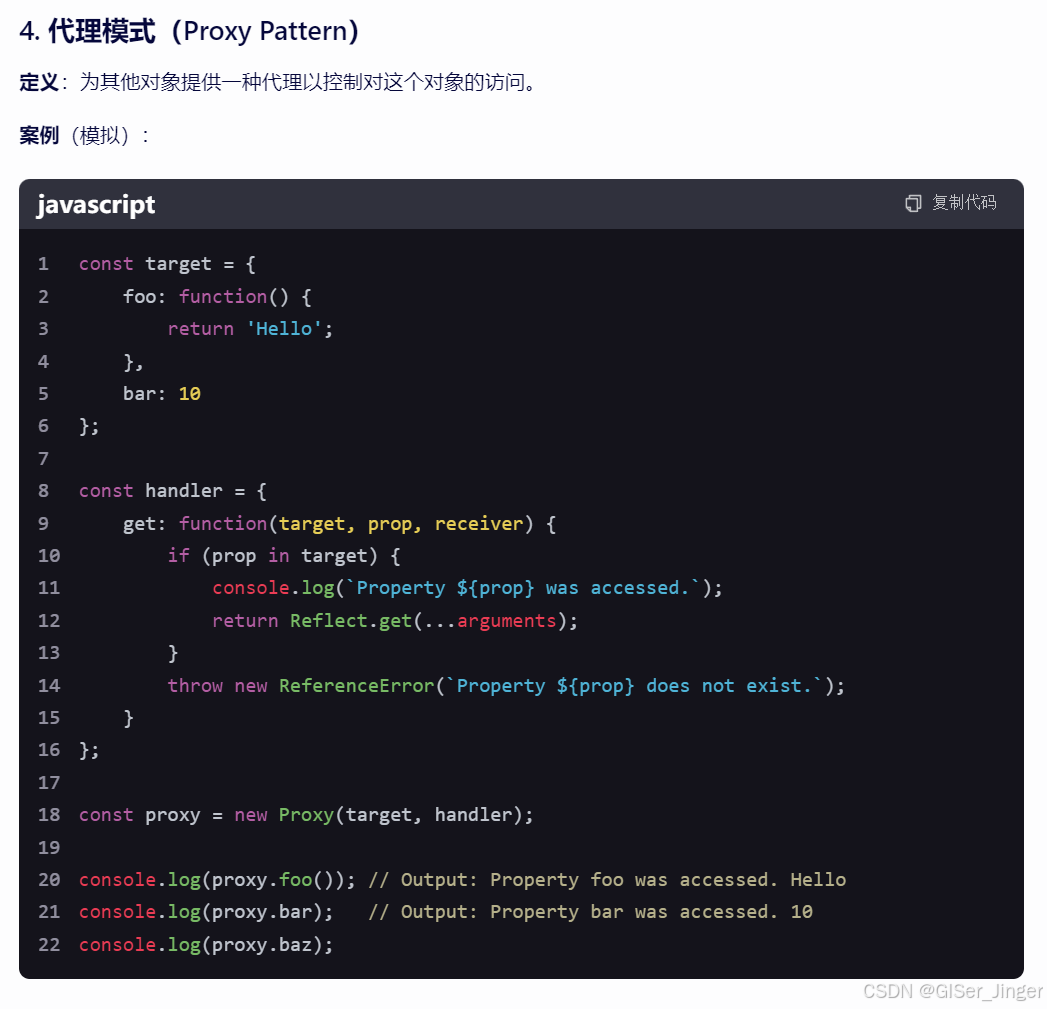
Javascript常见数据结构和设计模式
在JavaScript中,常见的数据结构包括两大类:原始数据类型(Primitive Types)和对象类型(Object Types)。对象类型又可以进一步细分为多种内置对象、数组、函数等。下面是一些JavaScript中常见的数据结构&…...

【ChatGPT】全面解析 ChatGPT:从起源到未来
ChatGPT 是由 OpenAI 开发的一个基于 GPT(Generative Pre-training Transformer)架构的聊天机器人。通过自然语言处理(NLP)技术,ChatGPT 能够理解和生成语言,与人类进行对话。本文将深入探讨其起源、发展、…...
html+css+js贪吃蛇游戏
贪吃蛇游戏🕹四个按钮控制方向🎮 源代码在图片后面 点赞❤️关注🙏收藏⭐️ 互粉必回🙏🙏😍😍😍 源代码📟 <!DOCTYPE html> <html lang"en"&…...
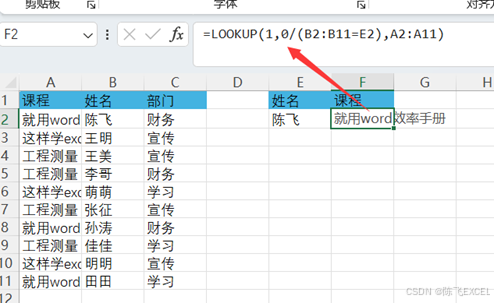
新手必学:掌握Excel中这些常用公式,轻松提升数据处理能力
各位同学好,今天和大家来分享几个常用函数公式的典型用法。 1、提取指定条件的不重复名单 如下图所示,某公司课程比赛,同一员工有多个比赛项目。希望从左侧的列表中,提取出财务部的参赛人员名单。F2单元格输入以下公式࿰…...
经济寒冬:竞品凶猛,你的产品如何求生?
那些年曾被竞品干掉的产品 1997年到2010年左右是国内互联网行业的快速发展和多元化发展的时期,这一时期涌现出来一大批优秀的产品,市场竞争越来越激烈。苹果 在20 世纪 80 年代,乔布斯的苹果电脑,在当时可是PC行业的老大…...

信号量——Linux并发之魂
欢迎来到 破晓的历程的 博客 引言 今天,我们继续学习Linux线程本分,在Linux条件变量中,我们对条件变量的做了详细的说明,今天我们要利用条件变量来引出我们的另一个话题——信号量内容的学习。 1.复习条件变量 在上一期博客中&…...
详解)
自动驾驶中的逆透视变换(Inverse Perspective Mapping,IPM)详解
前言 IPM(Inverse Perspective Mapping,逆透视变换)图的历史可以追溯到计算机视觉和图像处理领域的发展。逆透视变换是一种用于消除图像中透视效应的技术,使得原本由于透视产生的形变得以纠正,进而更准确地描述和理解图像中的场景。比如在行车中的车道线检测,泊车中的常见…...

Python地震波逆问题解构算法复杂信号分析
🎯要点 🎯时域、时频域以及时间和频率相关联偏振特性分析三种算法 | 🎯时域波参数估计算法 | 🎯机器学习模型波形指纹分析算法 | 🎯色散曲线和频率相关波分析算法 | 🎯动态倾斜校正算法 | 🎯声…...
C语言 -- 深入理解指针(二)
C语言 -- 深入理解指针(二) 1. 数组名的理解2. 使用指针访问数组3. 一维数组传参的本质4. 冒泡排序5. 二级指针6. 指针数组7. 指针数组模拟二维数组8. 字符指针变量9. 数组指针变量2.1数组指针变量是什么?2.2 数组指针变量怎么初始化 10. 二维…...

HTTP协议详解
HTTP协议详解 一、HTTP协议概述二、网络基础与HTTP2.1 TCP/IP协议2.2 发送HTTP请求过程2.3 HTTP请求的组成部分 三、HTTP报文HTTP请求报文HTTP响应报文 结语 一、HTTP协议概述 HTTP,即超文本传输协议(Hypertext Transfer Protocol)ÿ…...

利用最小二乘法找圆心和半径
#include <iostream> #include <vector> #include <cmath> #include <Eigen/Dense> // 需安装Eigen库用于矩阵运算 // 定义点结构 struct Point { double x, y; Point(double x_, double y_) : x(x_), y(y_) {} }; // 最小二乘法求圆心和半径 …...
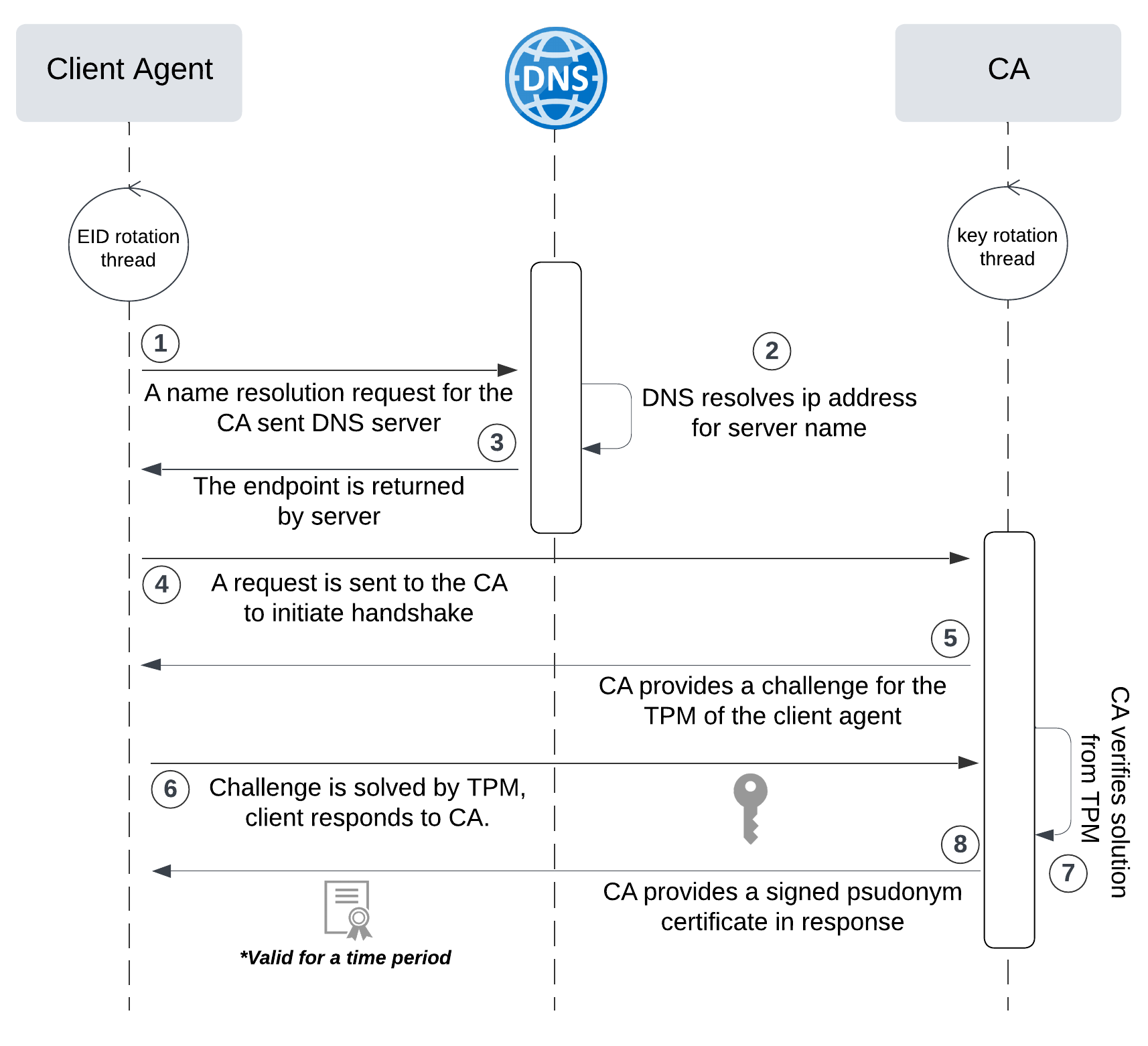
网络六边形受到攻击
大家读完觉得有帮助记得关注和点赞!!! 抽象 现代智能交通系统 (ITS) 的一个关键要求是能够以安全、可靠和匿名的方式从互联车辆和移动设备收集地理参考数据。Nexagon 协议建立在 IETF 定位器/ID 分离协议 (…...

OpenLayers 可视化之热力图
注:当前使用的是 ol 5.3.0 版本,天地图使用的key请到天地图官网申请,并替换为自己的key 热力图(Heatmap)又叫热点图,是一种通过特殊高亮显示事物密度分布、变化趋势的数据可视化技术。采用颜色的深浅来显示…...

基于大模型的 UI 自动化系统
基于大模型的 UI 自动化系统 下面是一个完整的 Python 系统,利用大模型实现智能 UI 自动化,结合计算机视觉和自然语言处理技术,实现"看屏操作"的能力。 系统架构设计 #mermaid-svg-2gn2GRvh5WCP2ktF {font-family:"trebuchet ms",verdana,arial,sans-…...
linux之kylin系统nginx的安装
一、nginx的作用 1.可做高性能的web服务器 直接处理静态资源(HTML/CSS/图片等),响应速度远超传统服务器类似apache支持高并发连接 2.反向代理服务器 隐藏后端服务器IP地址,提高安全性 3.负载均衡服务器 支持多种策略分发流量…...
)
React Native 导航系统实战(React Navigation)
导航系统实战(React Navigation) React Navigation 是 React Native 应用中最常用的导航库之一,它提供了多种导航模式,如堆栈导航(Stack Navigator)、标签导航(Tab Navigator)和抽屉…...
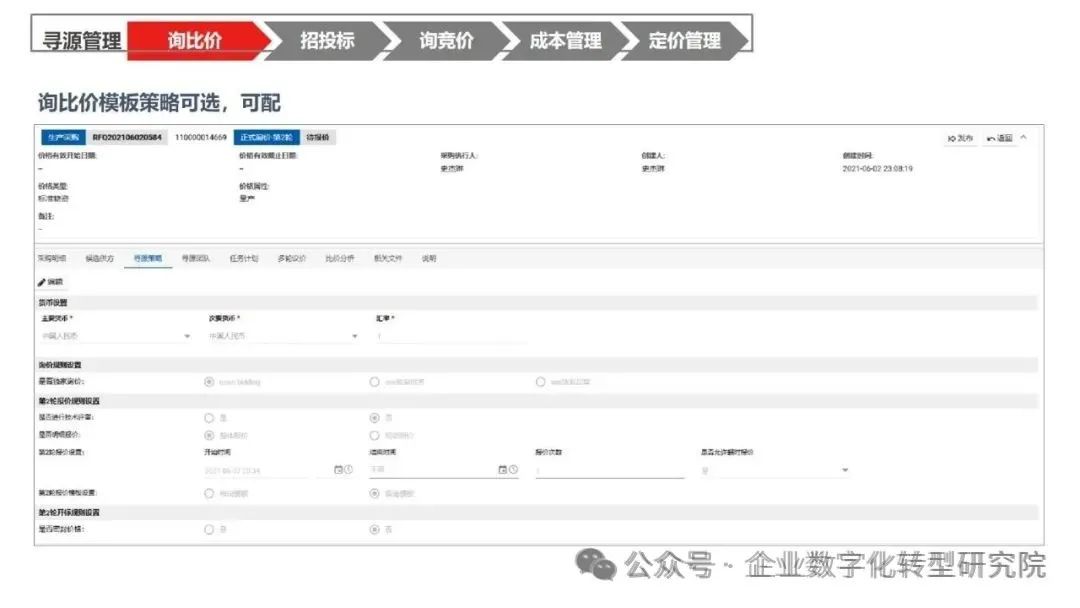
PPT|230页| 制造集团企业供应链端到端的数字化解决方案:从需求到结算的全链路业务闭环构建
制造业采购供应链管理是企业运营的核心环节,供应链协同管理在供应链上下游企业之间建立紧密的合作关系,通过信息共享、资源整合、业务协同等方式,实现供应链的全面管理和优化,提高供应链的效率和透明度,降低供应链的成…...
Mybatis逆向工程,动态创建实体类、条件扩展类、Mapper接口、Mapper.xml映射文件
今天呢,博主的学习进度也是步入了Java Mybatis 框架,目前正在逐步杨帆旗航。 那么接下来就给大家出一期有关 Mybatis 逆向工程的教学,希望能对大家有所帮助,也特别欢迎大家指点不足之处,小生很乐意接受正确的建议&…...
linux arm系统烧录
1、打开瑞芯微程序 2、按住linux arm 的 recover按键 插入电源 3、当瑞芯微检测到有设备 4、松开recover按键 5、选择升级固件 6、点击固件选择本地刷机的linux arm 镜像 7、点击升级 (忘了有没有这步了 估计有) 刷机程序 和 镜像 就不提供了。要刷的时…...
Python爬虫(一):爬虫伪装
一、网站防爬机制概述 在当今互联网环境中,具有一定规模或盈利性质的网站几乎都实施了各种防爬措施。这些措施主要分为两大类: 身份验证机制:直接将未经授权的爬虫阻挡在外反爬技术体系:通过各种技术手段增加爬虫获取数据的难度…...
