文献阅读:一种基于艾伦脑图谱的空间表达数据可视化、空间异质性描绘和单细胞配准工具
::: block-1
文献介绍

文献题目: AllenDigger,一种基于艾伦脑图谱的空间表达数据可视化、空间异质性描绘和单细胞配准的工具
研究团队: 王晓群(北京师范大学)
发表时间: 2023-03-16
发表期刊: The Journal of Physical Chemistry A
影响因子: 2.9(2023年)
DOI: 10.1021/acs.jpca.3c00145
:::
摘要
空间转录组学可用于捕获细胞空间组织,并促进对不同生物背景的新见解,包括发育生物学、癌症和神经科学。然而,其广泛应用仍因其技术挑战和不成熟的数据分析方法而受到阻碍。Allen Brain Atlas (ABA) 通过原位杂交图像数据为各个发育阶段的整个小鼠大脑的空间基因表达提供了重要的来源。据我们所知,为访问空间表达数据而开发的门户对生物学家来说并不是很有用。在这里,作者开发了一个工具包来收集和预处理来自 ABA 的表达数据,并允许更友好的查询来可视化感兴趣基因的空间分布,表征大脑的空间异质性,并注册从单细胞转录组数据到精细解剖学的细胞。通过机器学习方法高精度地识别大脑区域。AllenDigger 将在精确的空间基因表达查询方面对社区非常有帮助,并添加额外的空间信息,以经济有效的方式进一步解释 scRNA-seq 数据。
前言
组织结构和细胞空间组织对于维持和调节生物过程至关重要,并且不断受到细胞邻域和外部微环境的干扰。因此,解码空间背景对于充分理解生物复杂性至关重要。空间转录组学的快速发展促进了转录模式的全面表征,同时保留了组织空间组织,并已应用于研究空间基因表达异质性、细胞微环境和细胞间相互作用。例如,在发育生物学中,空间转录组学被用来构建空间基因表达景观,识别解剖学特异性表达基因并揭示细胞分化和迁移的空间背景。在神经病学中,空间转录组学已被用来描述功能失调的大脑的分子特征和细胞组织。阿尔茨海默病是一种进行性神经退行性疾病,会导致记忆力、思维能力和身体功能丧失。Chen et al. 通过分析阿尔茨海默病模型的空间转录组学,揭示了失调的基因和细胞网络。在病理学中,空间转录组学使我们能够精确探索肿瘤免疫环境以及肿瘤与免疫细胞之间的相互作用,这些相互作用负责细胞扩张和肿瘤进展。然而,对于不熟悉该领域的实验室来说,空间剖析技术和相关的数据分析方法在技术上仍然具有挑战性,而且成本高昂,需要付出更多努力来使其更加普遍、公众可访问和负担得起。
尽管当前的空间转录组学技术已经实现了基因表达捕获的细胞和亚细胞分辨率,但通过原位杂交 (ISH) 构建的基因表达图谱 Allen Brain Atlas 仍然是获取近 2100 全脑的全基因组空间基因表达谱数据的重要来源。在 Allen Brain Atlas 中,收集了小鼠大脑每个发育阶段的 ISH 结果,并将其转化为 3D 基因表达模型,并标注了其权威的大脑结构定义。在这个 3D 模型中,大脑结构被分为称为体素 (∼80-100 μm) 的网格立方体,其中每个体素都有其基因表达丰度、不同分辨率下的大脑区域注释等。因此,该模型可用于提供注释良好的空间基因表达景观,使得跨大脑区域的基因表达谱的探索成为可能。
为了方便访问 Allen Brain Atlas,Allen 研究所推出了 Brain Explorer 门户网站 (https://portal.brain-map.org/), 供社区查询和探索数据。该 Web 应用程序提供 2D 和 3D 空间中表达数据的交互式可视化,但不支持用于下游分析以进一步探索数据的高级模块。最近,Fleck et al. 构建了 Voxhunt,将类器官的 scRNA-seq 数据映射到 Allen Brain Atlas 中的全脑基因表达数据,从而使研究人员能够访问基因表达的空间分布,并通过计算相关系数将单个单细胞映射到体素上。尽管 Voxhunt 提供了将 scRNA-seq 的基因表达数据与 ABA 的空间基因表达数据整合的可能性,但其中采用的方法基于相关性分析,该分析仅捕获线性依赖性,并且可能受到用于测量细胞和体素之间相似性的基因的影响。因此,在不了解空间信息的情况下进行主观基因选择可能会导致分析出现偏差。
鉴于 ABA 提供了整个大脑的精确和严格的空间表达分析,以及神经科学领域绝大多数单细胞 RNA-seq 数据的公开可用性,作者在这里开发了一种名为 AllenDigger 的工具,它能够检索和可视化 ISH 以更具交互性和复杂性的方式获取图像数据,计算整个大脑的差异表达基因,并将细胞注册到大脑空间。在此工具中,作者实现了一个图自动编码器模型,该模型能够在对体素进行聚类时捕获空间信息,并且作者发现,与朴素自动编码器模型或使用纯表达数据的 Louvain 聚类结果相比,使用 ground truth 大脑区域标记可以获得更一致的聚类结果。更重要的是,该工具可用于通过实施基于机器学习的方法,将 scRNA-seq 数据中的单个细胞分配到具有高解剖分辨率的空间大脑区域(例如,皮质层、海马亚区域、下丘脑核),这可以为研究人员在分析 scRNA-seq 数据时添加空间信息,而无需额外成本。
研究方法
数据收集和格式化
Allen Brian Atlas 通过 API (http://help.brain-map.org/display/api/Allen%2BBrain%2BAtlas%2BAPI) 提供对其已发布数据和参考图谱的访问。为了方便使用和存储,小鼠大脑被分成 3D 网格立方体并转换为 3D 矩阵,其中每个体素与具有相同坐标的矩阵元素相匹配。分辨率从 80 到 200 μm 不等,具体取决于小鼠不同发育阶段的大脑大小。对于每个发展阶段,构建表达矩阵和结构参考矩阵。各个表达矩阵中的元素代表特定基因在特定体素处的表达能量,并计算为源自 ISH 图像的表达强度和表达密度的乘积。在参考矩阵中,每个元素代表一个精确解剖结构的 ID。通过重塑和连接每个发育阶段的所有 3D 基因表达阵列,基因阵列的 2D 体素被转换为 AnnData 格式,其中 x、y 和 z 坐标附加到匹配的体素(AnnData 是生物信息领域流行的数据格式,广泛用于多组学数据矩阵,例如转录组学和表观基因组学)。同时,参考矩阵被转换为分配给相应体素的多级注释结构的列表。所有这些注释和坐标都封装在 AnnData 对象的 obs 模块中。
输入图数据的构建
输入的图(graph)数据由邻接矩阵 A \mathbf{A} A 和特征矩阵 X \mathbf{X} X 组成。邻接矩阵是通过对点的空间坐标应用 K 近邻算法来构建的,并计算给定点与其 K 个邻居之间固定半径 r 内的欧氏距离。邻接矩阵 A \mathbf{A} A 用 1 和 0 填充。元素为 1 表示相应的一对由边连接的节点,而元素为 0 表示节点之间没有连接。此外,通过将对角线元素设置为 1 来考虑自循环。特征矩阵 X \mathbf{X} X 是通过从 Allen Brain Atlas 中提取每个点的基因表达信息而得到的。
图自编码器
图自编码器(graph autoencoder)是一种基于图数据进行学习的无监督方法。对于给定的无向图 G = ( V , E ) G = (V, E) G=(V,E),其中 N = ∣ V ∣ N = |V| N=∣V∣ 表示节点(spots), E E E 表示边(spots 之间的连接), A \mathbf{A} A 是 G G G 的邻接矩阵, D \mathbf{D} D 是其度矩阵。 X \mathbf{X} X 是包含节点特征的节点特征矩阵。
用于压缩图数据的编码器由两层图卷积网络(GCNs)组成,其定义如下

其中 G C N ( X , A ) = A ~ R e L U ( A ~ X W 0 ) W 1 GCN(\mathbf{X}, \mathbf{A}) = \tilde{A}ReLU(\tilde{A}\mathbf{X}W_0)W_1 GCN(X,A)=A~ReLU(A~XW0)W1, W i W_i Wi 表示权重矩阵。激活函数为 R e L U ( ⋅ ) = m a x ( 0 , ⋅ ) ReLU(·) = max (0, ·) ReLU(⋅)=max(0,⋅),对称归一化邻接矩阵为
A ~ = 𝐃 − 1 / 2 𝐀 𝐃 − 1 / 2 \tilde{A} =𝐃^{−1/2}𝐀𝐃^{−1/2} A~=D−1/2AD−1/2。潜在嵌入 Z Z Z 定义为编码器输出的函数,

解码器被定义为两个潜在嵌入之间的内积
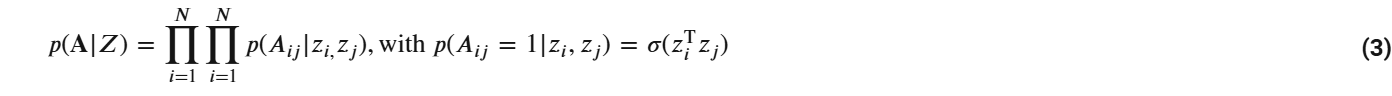
其中 A i j A_{ij} Aij 是 A \mathbf{A} A 的元素, σ ( ⋅ ) \sigma(·) σ(⋅) 是逻辑 sigmoid 激活函数。
损失函数 L L L 衡量重建的信息损失,定义如下:

其中 K L [ q ( ⋅ ) ∥ p ( ⋅ ) ] KL[q(·)∥p(·)] KL[q(⋅)∥p(⋅)] 是 q ( ⋅ ) q(·) q(⋅) 和 p ( ⋅ ) p(·) p(⋅) 之间的 Kullback-Leibler 散度
聚类分析
对于聚类分析,将 scikit-learn 的高斯混合模型应用于潜在空间中的特征,并考虑 Akaike 信息准则(AIC)和贝叶斯信息准则(BIC)手动选择分量数量。
空间差异表达基因的鉴定
为了计算空间差异表达的基因,作者首先使用 scanpy.tl.rank_genes_groups 函数和 "wilcoxon" 方法来计算基因分数。然后使用 scanpy.tl.filter_genes_groups 函数来选择空间差异表达的基因。这两个函数都来自 Python 中的 Scanpy 包。
特征选择
对于特征选择,应用了弹性网络。基本上,它结合了 L1 和 L2 惩罚,将不重要特征的系数缩小到接近零。简而言之,作者将 Python 中 sklearn 包中的线性模型 SGDClassifier 的弹性网络模式应用于 Allen ISH 数据,并以解剖空间注释作为类参考。因此,计算每组内基因的线性回归系数。接下来,根据基因的系数对基因进行排名,并选择排名靠前的基因作为相应解剖类别的签名特征,然后将其传递给随机森林分类器进行空间位置预测任务。
随机森林分类
分类器的训练将具有选定特征的 Allen ISH 数据作为输入数据,将解剖结构信息作为输入参考类标签。作者使用 Python 中 sklearn 包中的 GridSearchCV 模型选择函数来训练具有不同参数集的随机森林分类器,其中 max_depth 在 [2,3,5,10,20],min_samples_leaf 在 [5,10, 20,50,100,200],n_estimators 在 [10,25,30,50,100,200],并选择具有最高平均交叉验证分数的分类器进行空间位置推断。
研究结果
1. AllenDigger 概述
在 AllenDigger 中,9 个 ISH 和解剖数据集,包括来自 ABA 的 8 个发育中的小鼠大脑阶段 (E11.5∼P56) 和 1 个成年小鼠大脑被转换为 AnnData 格式,这是一个通用类广泛用于表达矩阵的操作(参见理论方法部分的“数据收集和格式化”)(Figure 1)。该工具提供了一个用于解剖和基因表达数据的 2D 和 3D 视觉表示的模块,以及 3 个分析模块,以促进揭示空间模式的高级分析功能(Figure 1)。在这 3 个分析模块中,通过构建图自编码器模型,将基因表达谱与空间信息相结合,实现空间表达模式的提取;根据 ABA 的解剖结构信息,可以识别大脑结构中的差异表达基因,并且基于具有表征个体解剖结构特征的特征基因的预测模型的构建,对来自 scRNA-seq 数据的细胞进行空间注册。
:::block-1
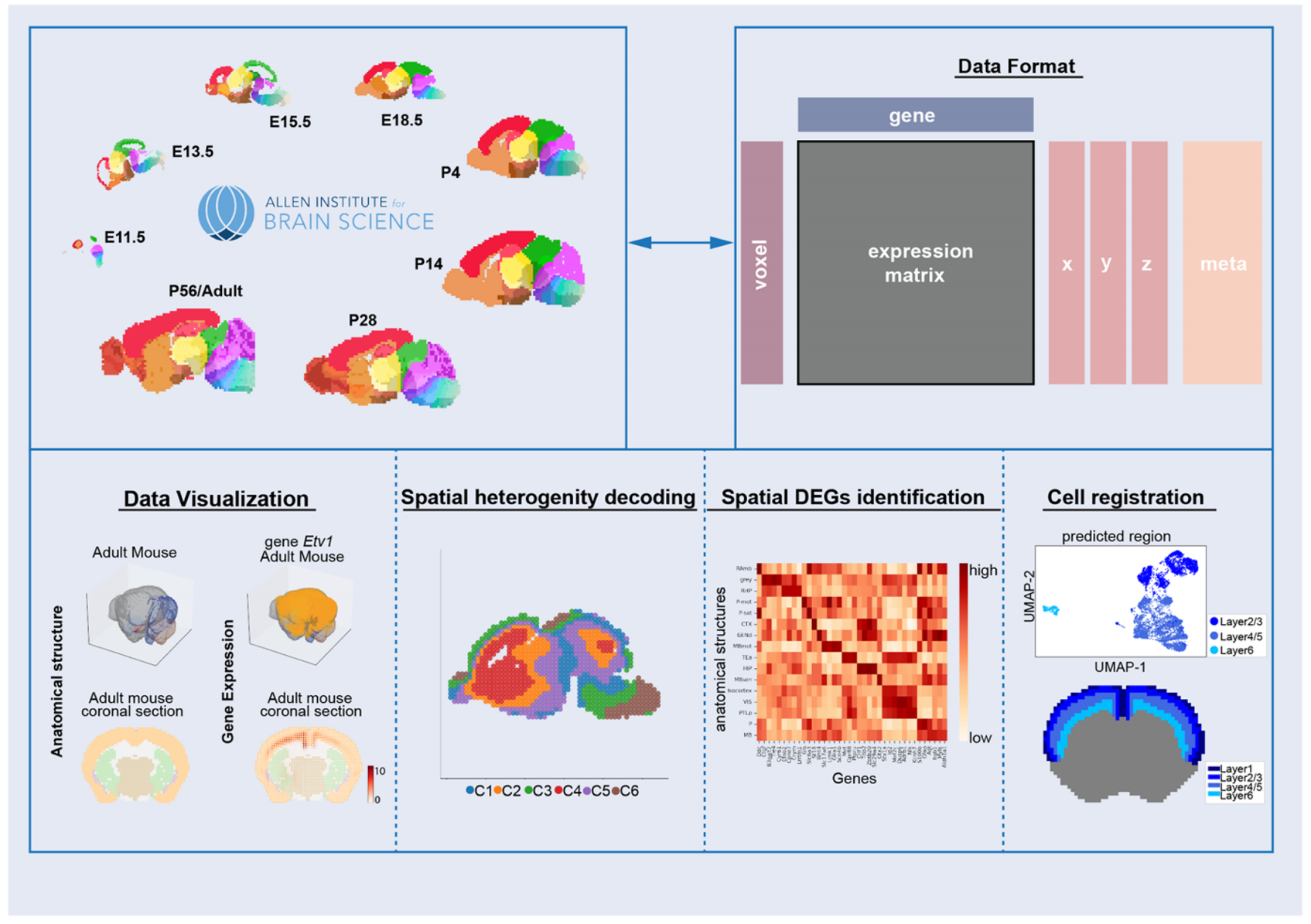
AllenDigger 涵盖了小鼠大脑 9 个阶段的 ISH 数据以及相应的结构注释。AllenDigger 目前的实现包括四个主要模块:数据可视化、空间异质性解码、空间 DEGs 识别和细胞注册。
:::
2. AllenDigger 实现全面、灵活的数据可视化
为了促进跨不同大脑结构的基因表达谱的探索,一个模块能够在不同注释级别描绘大脑结构的 3D 组织(在较低的注释级别实现粗略的大脑结构注释,而在较高的注释级别实现精细的结构注释),并使用原位点图表示将基因表达投影到 3D 大脑模型上。简而言之,每个点显示相应结构位置的基因表达水平,每个点的顺序渐变颜色随基因表达水平缩放(Figure 2a,b)。对于精细化大脑区域很重要的研究,重要的是在二维空间中以更高的注释级别可视化三个平面(冠状面、水平面和矢状面)中的大脑结构或大脑结构的特定子集(Figure 2a–c),这可以在 AllenDigger 中通过明确指定将使用哪些可视化平面、大脑区域和注释级别来实现。因此,AllenDigger 实现的可视化模块可以成为一个有用的查询工具,用于研究和可视化整个解剖大脑中感兴趣基因的表达谱。
:::block-1
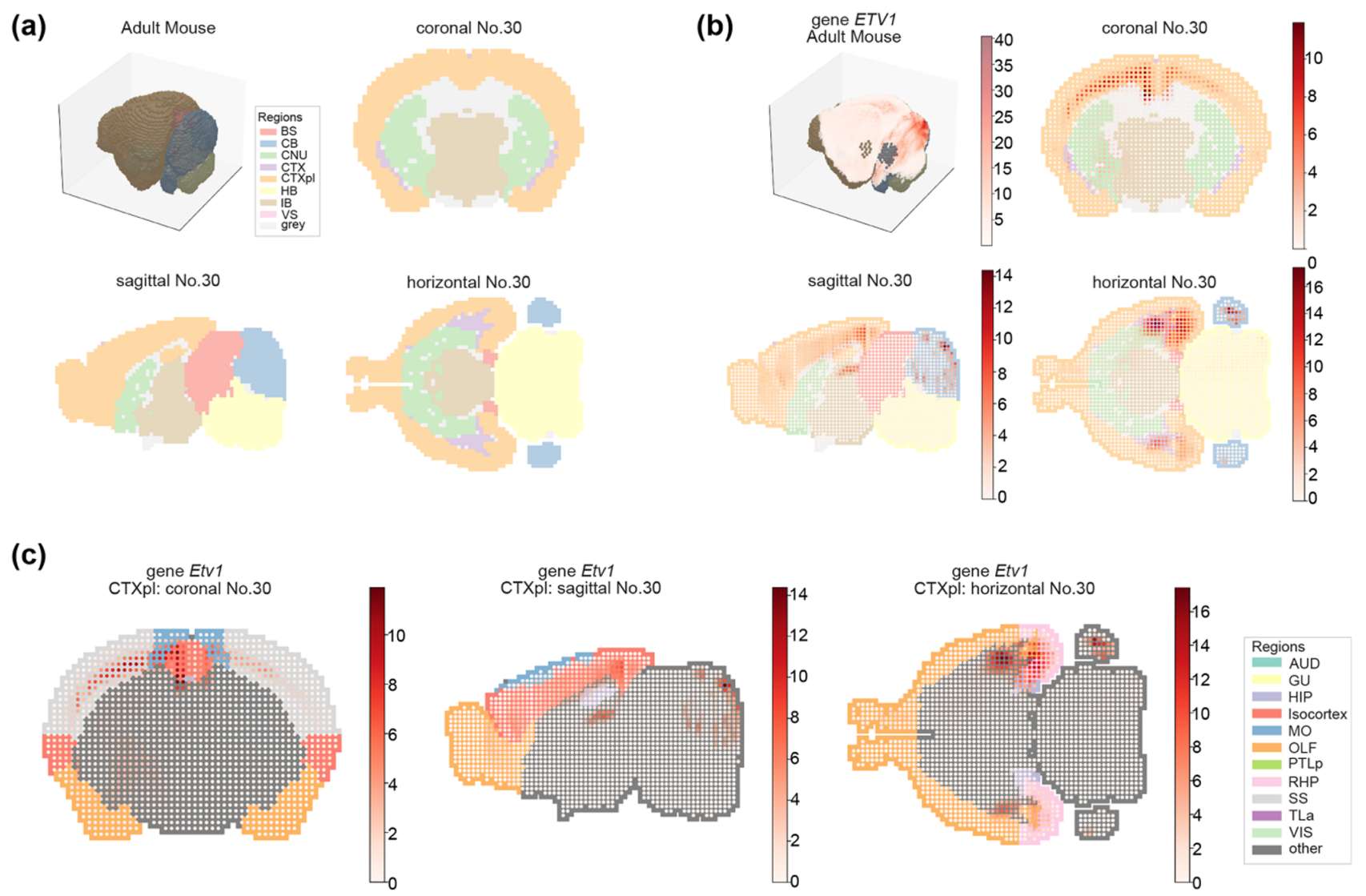
(a) 冠状面、矢状面、水平面的大脑解剖结构注释的可视化。
(b) 2D 空间中的冠状面、矢状面、水平面以及 3D 空间中的整个小鼠大脑中 Etv1 基因表达分布的可视化,并带有解剖结构注释。离散颜色代表不同的大脑结构,渐变颜色表示精确体素上的基因表达能量(白色,低表达;深红色,高表达)。
© 大脑皮层上的 Etv1 基因表达以及 level-6 解剖结构注释。离散颜色代表不同的大脑结构,梯度颜色根据精确体素上的基因表达能量进行缩放(白色,低表达;深红色,高表达)。灰色表示大脑皮层以外的大脑区域。
:::
3. AllenDigger 通过图自动编码器 (GAE) 模型揭示空间表达域
图自动编码器(GAE)是一种强大的基于图网络的方法,由编码器和解码器组成,能够将数据映射到具有高数据结构保真度的低维潜在空间,因此可用于降维和空间表达域的识别。在该模块中,图自动编码器采用图网络作为输入,该图网络是通过使用 K 近邻(KNN)算法计算体素之间的欧几里德距离而构建的,并使用基因表达数据来表示输入网络中每个节点的特征。编码器使用图卷积网络(GCN)将构造的图网络编码为低维嵌入表示,然后通过解码器重建该表示以重新生成原始输入图网络。GAE 模型抽象了图邻域结构,同时最小化了重建表示与输入图之间的相似性损失。然后使用高斯混合模型(GMM)将抽象嵌入聚类成子组,并使用 UMAP 进行可视化。
为了评估该模块的性能并使用 GAE 验证改进的输出,作者使用 AllenDigger 的 GAE 模型、STAGATE 中的自适应图注意自动编码器模型 (AdaGAE)、自动编码器模型 (AE),以及广泛使用的 SCANPY 中 的 scRNA-seq 聚类方法算法 (Louvain),对来自 Allen 小鼠大脑的 12 个代表性冠状切片(覆盖 28 个大脑区域)进行空间聚类分析。并采用调整后的 Rand 指数 (ARI)、同质性评分(HS)和归一化互信息(NMI)作为性能指标,根据 ABA 的解剖注释信息评估聚类准确性(Figure 3a)。总体而言,与 AdaGAE、AE 和 Louvain 算法相比,GAE 模型可以更准确地识别大脑解剖结构,其聚类一致性明显更高,并通过 ARI、HS 和 NMI 值进行量化(Figure 3b,c)。值得注意的是,将 GAE 模型应用于运动皮层数据证明了该模型即使在更高分辨率的情况下也能恢复结构复杂性(Figure 3d)。在 UMAP 上可以看到来自不同层的 spots 相对清晰的分离,并得到已知层标记的支持。例如,Lamp5、Etv1 和 Foxp2 分别被实验证实为第 2/3 层、第 5 层和第 6 层的标记基因,并在 GAE 模型识别的相应空间域上表现出较高的表达(Figure 3e)。此外,将该模块应用于 Allen Developing Mouse Brain Atlas 的冠状切片,实现了小鼠大脑在各个发育阶段的空间模式的解剖(Figures S1 and S2)。总的来说,在聚类分析中集成空间信息可以更准确地描绘空间结构,并且采用 AllenDigger 的 GAE 模型实现的模块在解读空间域方面表现出可靠的性能。
:::block-1
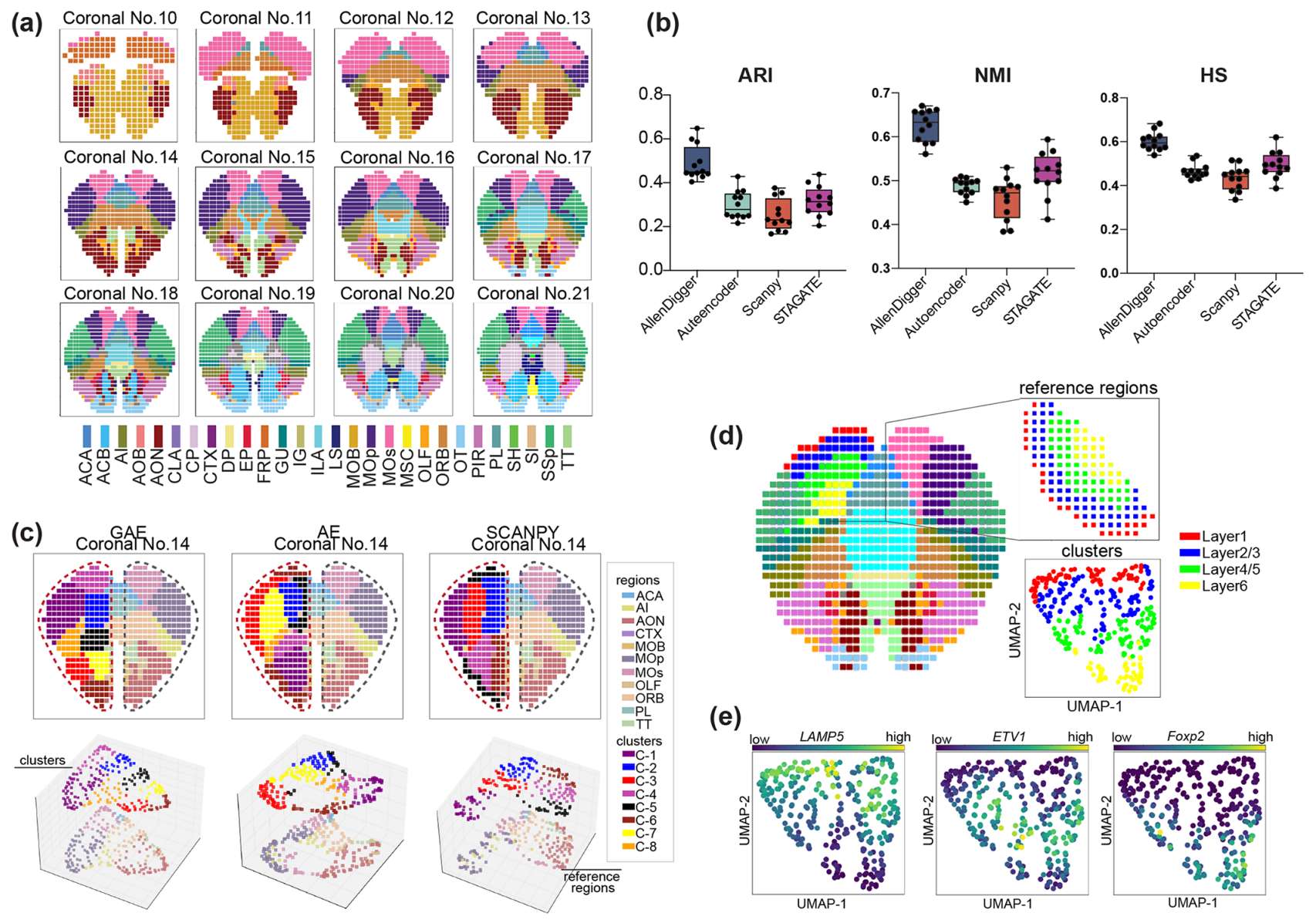
(a) Allen 小鼠大脑 12 个冠状切片的解剖结构信息可视化。
(b) 箱线图显示 4 种方法(AllenDigger、Autoencoder、Scanpy 和 STAGATE)12 个冠状切片的 ARI、HS 和 NMI 分数。在每个箱线图中,每个点代表一个单独的冠状切片,水平白线表示中值,下须和上须的边界分别代表最小值和最大值。
© GAE 模型、AE 模型和 SCANPY 在小鼠大脑模型(上)和 UMAP(下)的第 14 个冠状切片上进行的聚类结果的可视化。
(d) 大脑模型(左)和 UMAP(右)上运动皮层聚类结果的可视化。
(e) UMAP 上已知皮质层标记的表达。每个点代表单个 spot,并用表达水平着色(黄色,高表达;深紫色,低表达)。
:::
4. AllenDigger 可用于差异表达分析以识别结构的特征基因
由于 ISH 数据显示不同大脑区域甚至更精细注释结构的空间表达模式(Figure 3),因此识别每个区域的差异表达基因可以帮助理解空间组织背后的生物背景。为了完成差异表达分析,作者采用 Scanpy 中成熟的计算差异表达基因(DEG)的方法来计算不同结构之间的 DEGs,并保留 p 值小于 0.05 的基因。然后,按 log2Foldchange 值排序的基因被视为每个大脑区域的特征基因(Figures S3 and S4)。
5. AllenDigger 可以通过随机森林将细胞注册到详细的大脑解剖结构中
在过去的十年中,单细胞 RNA 测序技术已成为一种广泛使用的方法,通过将组织解剖成单个细胞来获得单细胞水平的基因表达谱,不幸的是,这导致了其原始空间信息的丢失。由于来自不同大脑区域的细胞的基因表达谱不同,并且在生物过程中发挥不同的作用,因此从 scRNA-seq 恢复细胞的空间位置对于复杂生物系统的综合研究至关重要。因此,在 AllenDigger 中,实现了一种基于模型的方法,旨在从 scRNA-seq 推断细胞的空间信息。简而言之,为了分配细胞解剖结构标签,首先使用来自 ABA 的 ISH 数据的基因表达数据以及结构注释信息,通过训练具有弹性网络模式的随机梯度下降 (SGD) 学习的线性模型来选择每个结构的代表性基因。然后,将各个区域中按线性回归系数排名靠前的基因作为一组特征基因,传递给后续的分类模型。使用精心设计的体素基因表达矩阵训练随机森林分类器,该矩阵仅包含特征集中的基因。最后,可以通过将预训练的分类器应用于 scRNA-seq 数据来实现细胞的空间配准。接下来,作者验证了细胞注册模块的实用性,首先将其应用于位于不同皮质层的细胞的 scRNA-seq 数据,这些数据根据标记基因的表达被注释为不同的皮质层(Figure 4c)。大脑皮层由 6 个细胞层组成,每个细胞层都有不同的转录谱(Figure 4a,b)。为了定量评估配准性能,将先前研究中的注释作为真实标签,通过将预测结果与真实标签进行比较来计算混淆矩阵。作者发现 scRNA-seq 中的大多数细胞都被正确分配到其空间位置,准确度相对较高,为 0.80(Figure 4d,e)。请注意,原始 scRNA-seq 数据中没有收集来自皮质层的细胞;因此,在作者的预测模型中,细胞很少被映射到皮质层 1。然后,为了定量评估该模块的预测精度,作者将 AllenDigger 得出的注册结果与三种现有的空间注册方法(Voxhunt、SingleR、和 Tangram)进行了比较。总体而言,AllenDigger 对空间位置的估计比其他方法更准确。事实上,与每个皮质层的真实空间标签相比,AllenDigger 产生的分配给错误空间标签的细胞比例要低得多。相比之下,其他方法在错误空间注释和真实空间注释之间产生明显较弱的对比度。总的来说,AllenDigger 的细胞注册模块准确地解决了空间变化,并为 scRNA-seq 中的细胞实现了更准确的空间注册(Figure S5)。
:::block-1
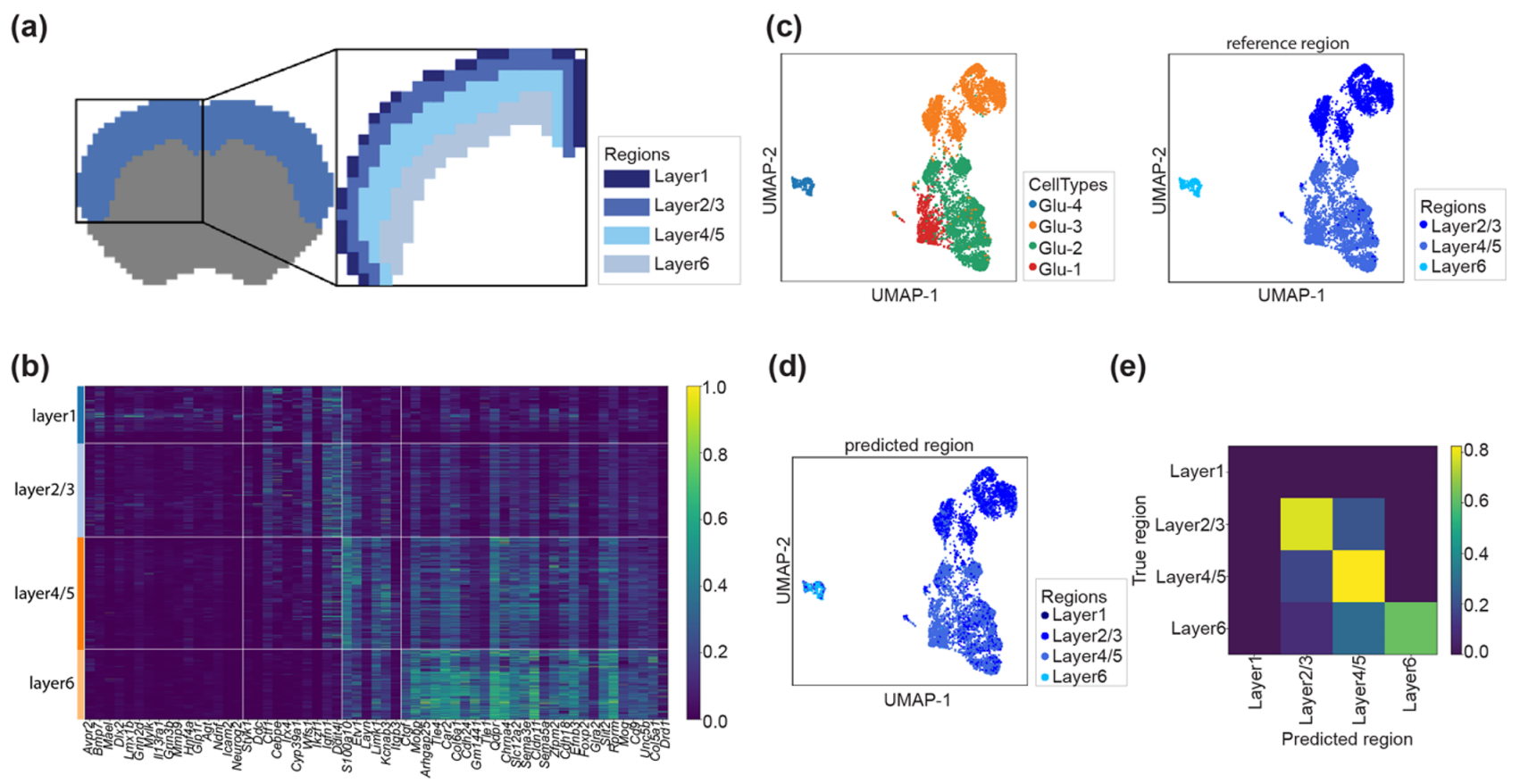
(a) 成年小鼠大脑参考第 25 号冠状切片上显示的小鼠大脑皮层的解剖层结构。
(b) 皮质层中已识别的空间差异表达基因的基因表达水平热图。
© UMAP 表示皮质单细胞 RNA 测序数据的层结构和细胞类型信息。
(d) 连接到每个细胞的预测层标签的 UMAP 可视化。
(e) 将皮质细胞注册到层结构的混淆矩阵结果。
:::
此外,该注册功能在推断来自海马体的功能分区(3 个 cornu ammonis 子区,CA1、CA2、CA3、DG)的 snRNA-seq 数据的空间位置方面表现出色,包括 1402 个具有相应空间信息的细胞。海马体的空间域通过空间 ISH 和 sc-RNA 测序数据得到了很好的表征(Figure 5a–c)。细胞位置的预测与其附加的标签显示出高度的一致性,具有相对较高的准确度 0.82(Figure 5d)。
:::block-1
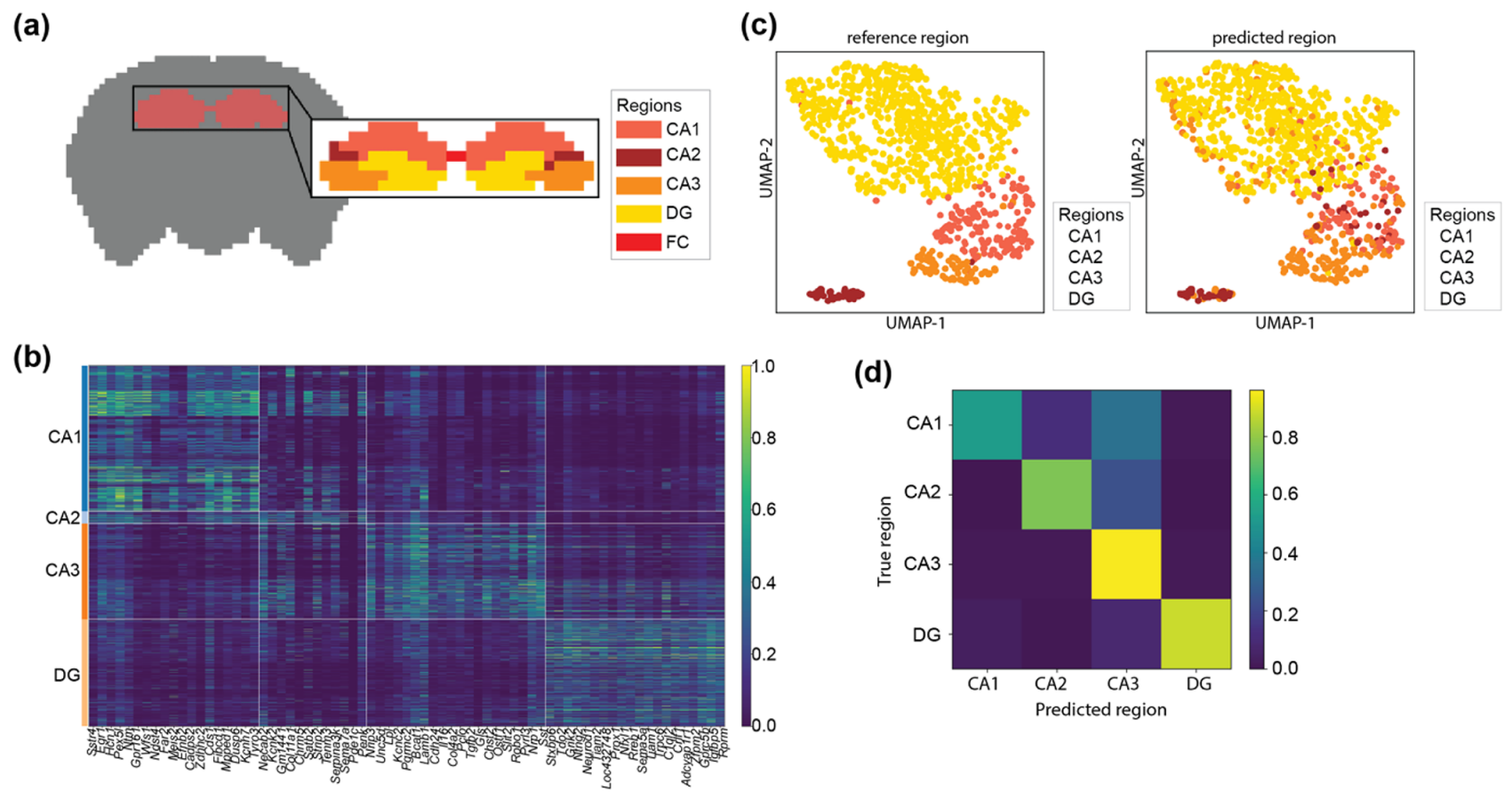
(a) 成年小鼠大脑参考第 35 号冠状切片上显示的小鼠海马体的解剖结构。
(b) 海马亚区域中确定的空间差异表达基因的基因表达水平热图。
© 海马 scRNA-seq 数据的海马亚区的 UMAP 呈现(左)和每个单细胞上预测的海马亚区标签(右)。
(d) 将海马细胞注册到海马亚区域的混淆矩阵分布。
:::
为了进一步评估将细胞注册到详细结构的能力,作者采用 AllenDigger 将下丘脑核的 scRNA-seq 数据中的细胞映射到空间位置。下丘脑是一个复杂的大脑结构,具有复杂的环路,由负责调节体内平衡的各种细胞群组成(Figure 6a,b)。因此,破译下丘脑细胞的空间位置对于展示其空间和环路组织很有价值。然而,空间复杂性使得将细胞分配到空间核上具有挑战性。在应用细胞注册模块进行空间位置预测后,作者计算了映射到下丘脑每个单独核上的 scRNA-seq 数据集中的细胞数量(Figure 6c)。大多数细胞分配给 mammillary (M) (16%)、posterior hypothalamus (PH) (41%)、lateral hypothalamic area (LHA) (11%) 和 preoptic nucleus (31%)。由于原始数据集中没有核注释,因此作者根据标记基因的表达作为这四个下丘脑核的代理来评估注册模块的性能(Figure 6d)。mammillary (M) 的基因标记 Foxb1 和 Lhx1、PH 的 Cck、LHA 的 Ss18l1、preoptic nucleus 的 Nts 和 Penk 的表达全部聚集在目标区域,验证了 AllenDigger 的细胞注册模块在破译复杂大脑结构方面的效用。
:::block-1
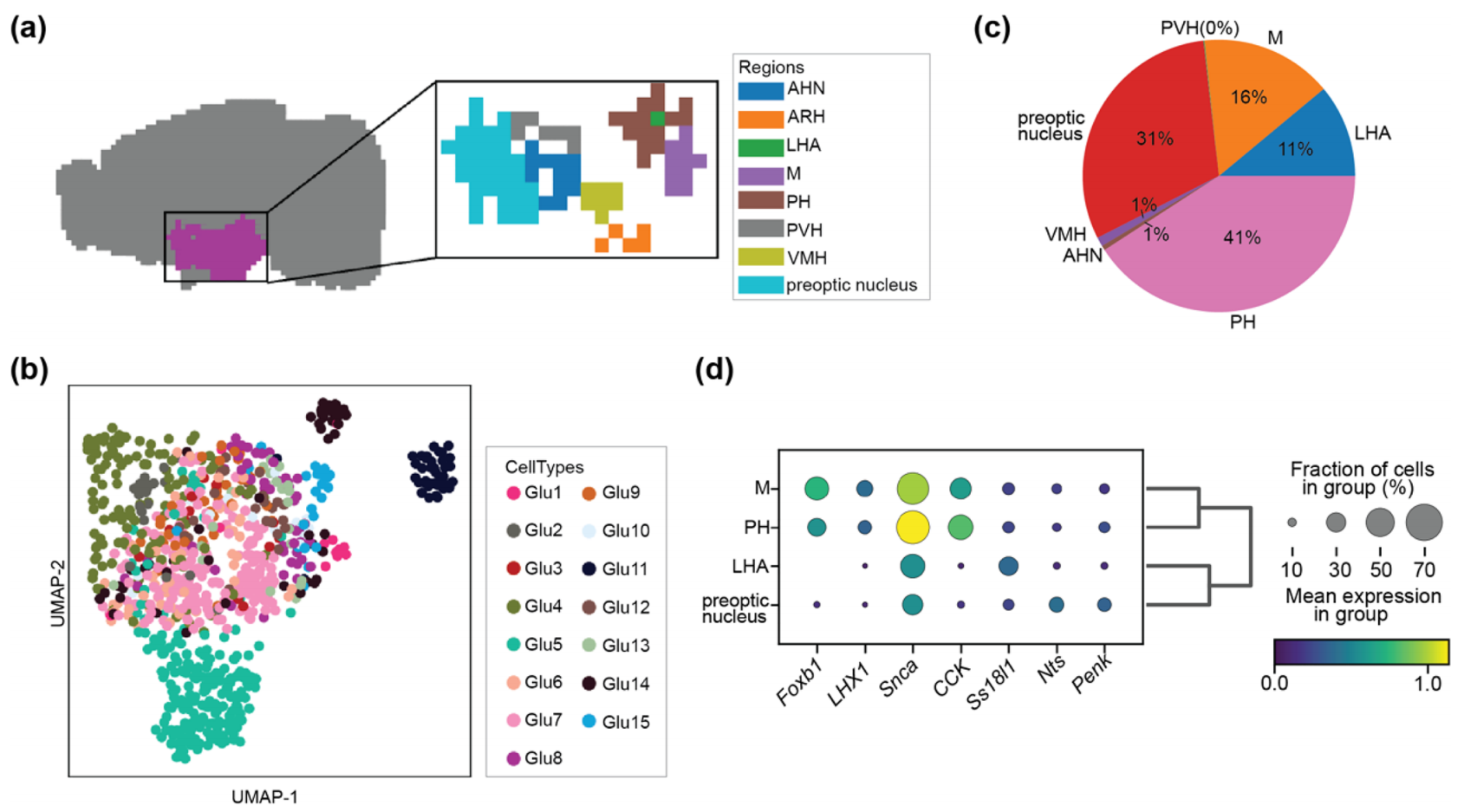
(a) 成年小鼠大脑参考第 30 矢状切面上显示的 8 个下丘脑核团的解剖位置。
(b) 下丘脑单细胞测序数据的细胞类型信息的 UMAP 可视化。
© 分配给下丘脑每个核的细胞分数。
(d) 预测核上标记基因表达分布的点图。
:::
总结
空间基因表达谱对于神经科学非常重要,因为大脑具有高度复杂的解剖功能空间结构并涉及各种细胞群。在这项工作中,作者构建了一个计算工具,提供多个模块来探索 Allen 大脑图谱中的大量数据,使研究人员能够访问和可视化各个发育阶段的大脑区域之间的基因表达谱。与简单的自动编码器、自适应图注意自动编码器或广泛使用的从纯表达数据导出的 Louvain 算法相比,作者将空间邻域信息融入到 GAE 模型中,并实现了更准确的大脑空间结构域描绘。此外,作者还实现了一个基于随机森林模型的模块,该模块能够高精度地将单细胞从 scRNA-Seq 数据映射到 ABA 定义的精细大脑区域。相信该工具将有利于神经科学研究,因为它可以灵活地查询 ABA 中感兴趣的基因的空间表达数据,并且能够高精度地向表达数据添加空间注释。
该工具有一些限制。首先,作者注意到某些大脑区域(例如下丘脑腹内侧区,VMH)没有足够的体素用于建模训练或相关性计算,当图谱数据进一步扩展时可以改善这一点。其次,为了增强图谱数据的空间分辨率,可以使用点反卷积算法(RCTD,cell2location)来有效解码一个大脑体素的细胞组成。此外,根据 Allen 研究所的基因表达能量计算方法和更精细的参考图谱,可以通过减少每个体素中包含的细胞数量来细化大脑的体素。第三,AllenDigger 目前有 9 个发育中和成年小鼠大脑数据集可供使用,而人类大脑进化出与其他哺乳动物截然不同的特征,备受人类疾病和行为研究的关注。因此,Allen Digger 数据库可以通过将人脑 ISH 图像从 ABA 转换为二元体素-基因矩阵来进一步扩展,以便探索人脑。
注:本文为个人学习笔记,仅供大家参考学习,不得用于任何商业目的。如有侵权,请联系作者删除。

相关文章:

文献阅读:一种基于艾伦脑图谱的空间表达数据可视化、空间异质性描绘和单细胞配准工具
::: block-1 文献介绍 文献题目: AllenDigger,一种基于艾伦脑图谱的空间表达数据可视化、空间异质性描绘和单细胞配准的工具 研究团队: 王晓群(北京师范大学) 发表时间: 2023-03-16 发表期刊:…...
Redis学习笔记(三)--Redis客户端
文章目录 一、命令行客户端二、图形界面客户端1、Redis Desktop Manager2、RedisPlus 三、java代码客户端 本文参考: Redis学习汇总(已完结) Redis超详细入门教程(基础篇) Redis视频从入门到高级,redis视频…...

面试知识梳理
一、vue篇章 1.vue2和vue3性能方面的提升最主要的原因是什么? 1、1响应式的系统优化: vue3使用了es6的proxy对象来实现响应式系统,取代了vue2中基于Object.defineProperty的方法。Proxy提供了更强大和灵活的拦截能力,可以更有效地…...

Unity3D ScrollView 滚动视图组件详解及代码实现
前言 在Unity3D中,ScrollView(滚动视图)是一种常用的UI组件,它允许用户通过滚动来查看超出当前视图范围的内容。ScrollView通常用于显示长列表、大量文本或图像等。本文将详细介绍Unity3D中的ScrollView组件,并提供代…...

13.java面向对象:封装
java面向对象:封装 我们程序设计要追求“高内聚,低耦合”。高内聚就是类的内部数据操作细节自己完成,不允许外部干涉;低耦合:仅暴露少量的方法给外部使用。 封装(数据的隐藏)通常应禁止直接访问一个对象中…...
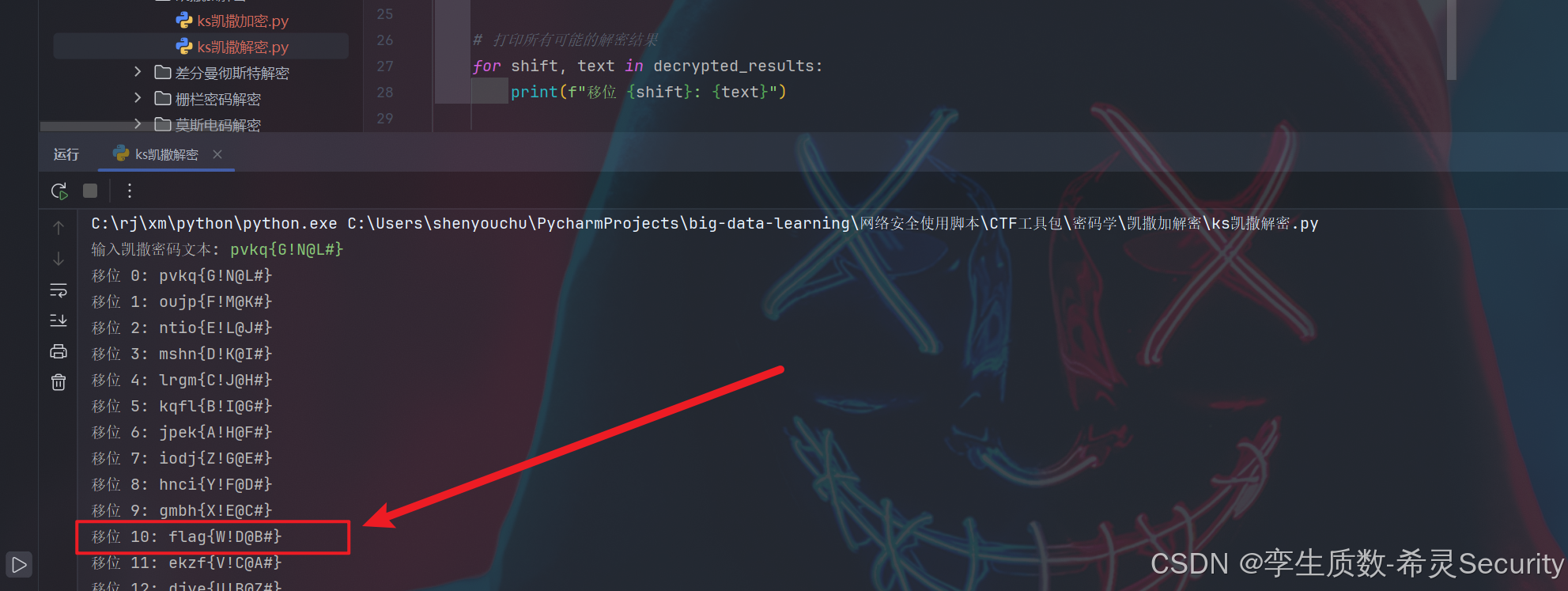
记录:网鼎杯2024赛前热身CRYPT01密码学
题目 下载并打开附件 判断为凯撒密码,尝试移位解密 在第10位发现flag字样 提交得分 解密脚本为个人自用,因比赛未结束故不开源...

GitHub加速
GitHub加速 终端命令行 支持终端命令行 git clone , wget , curl 等工具下载. 支持 raw.githubusercontent.com , gist.github.com , gist.githubusercontent.com 文件下载.注意:不支持 SSH Key 方式 git clone 下载. git clone git clone https://ghp.ci/https:…...

每天学习一个Linux命令:xrandr
xrandr 是一个用于在 X Window 系统中管理显示器的命令行工具。它可以用来设置显示器的分辨率、刷新率、旋转方向和连接状态等。下面是 xrandr 的详细用法和案例。 基本用法 xrandr [选项]常用选项 -q 或 --query: 查询当前显示器的状态。-s 或 --size: 设置显示器的分辨率。…...
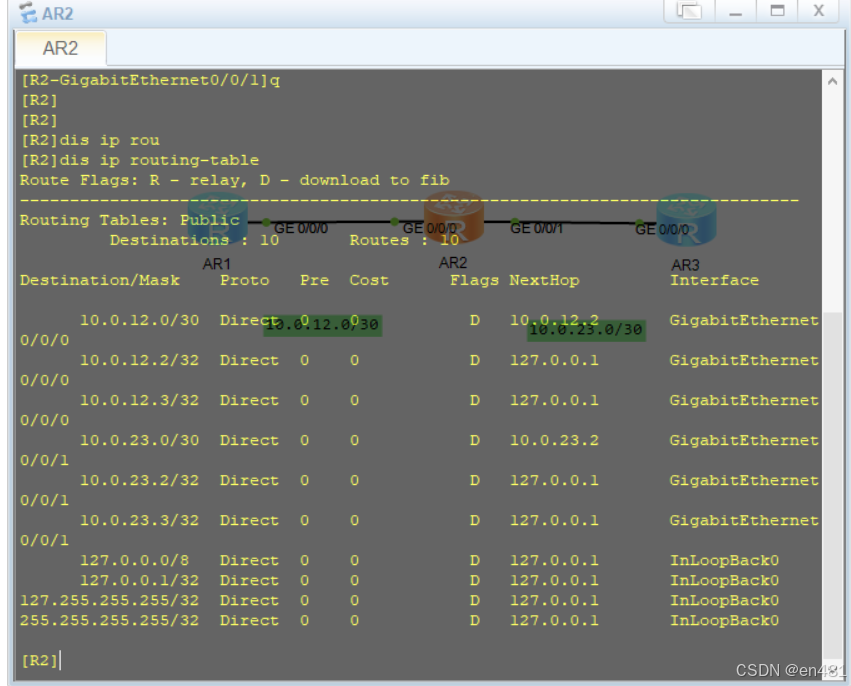
路由表来源(基于华为模拟器eNSP)
概叙 在交换网络中,若要实现不同网段之间的通信,需要依靠三层设备(路由器、三层交换机等),而路由器只知道其直连网段的路由条目,对于非直连的网段,在默认情况下,路由器是不可达的&a…...
)
并查集(Union-Find)
并查集(Disjoint Set,也称为Union-Find数据结构)是一种用于高效处理不相交集(即集合内元素互相独立,没有交集)的数据结构。它主要用于解决以下两种操作: 查找(Find)&…...

Linux上的AI框架都有哪些?哪些AI框架适合驱动EACO地球链自动发展完善?
Linux上的AI框架种类繁多,涵盖了深度学习、机器学习、自然语言处理等多个领域。以下是一些常用的AI框架: 深度学习框架 Deeplearning4j 简介:Deeplearning4j(Deep Learning For Java)是Java和Scala环境下的一个开源分…...

java的第一个游戏界面
看视频02_大鱼吃小鱼_添加背景图_尚学堂_哔哩哔哩_bilibili 学习方法: 就对的视频小代码,书籍没有,遇到不懂的问ai 今日成果, 界面代码 package new_gameobj;import java.awt.Graphics; import java.awt.Image; import java.…...

【AIGC】ChatGPT提示词Prompt高效编写模式:Self-ask Prompt、ReACT与Reflexion
博客主页: [小ᶻZ࿆] 本文专栏: AIGC | ChatGPT 文章目录 💯前言💯自我提问 (Self-ask Prompt)如何工作应用实例优势结论 💯协同思考和动作 (ReACT)如何工作应用实例优势结论 💯失败后自我反思 (Reflexion)如何工作…...

android studio无法下载依赖包问题
新建Flutter项目Android项目后,点击运行出现报错! error.png 这是镜像站点无法访问造成的!只需要修改为国内可访问的站点即可。 第一步:修改项目Android目录下的build.gradle buildscript { ext.kotlin_version 1.3.50 repositorie…...
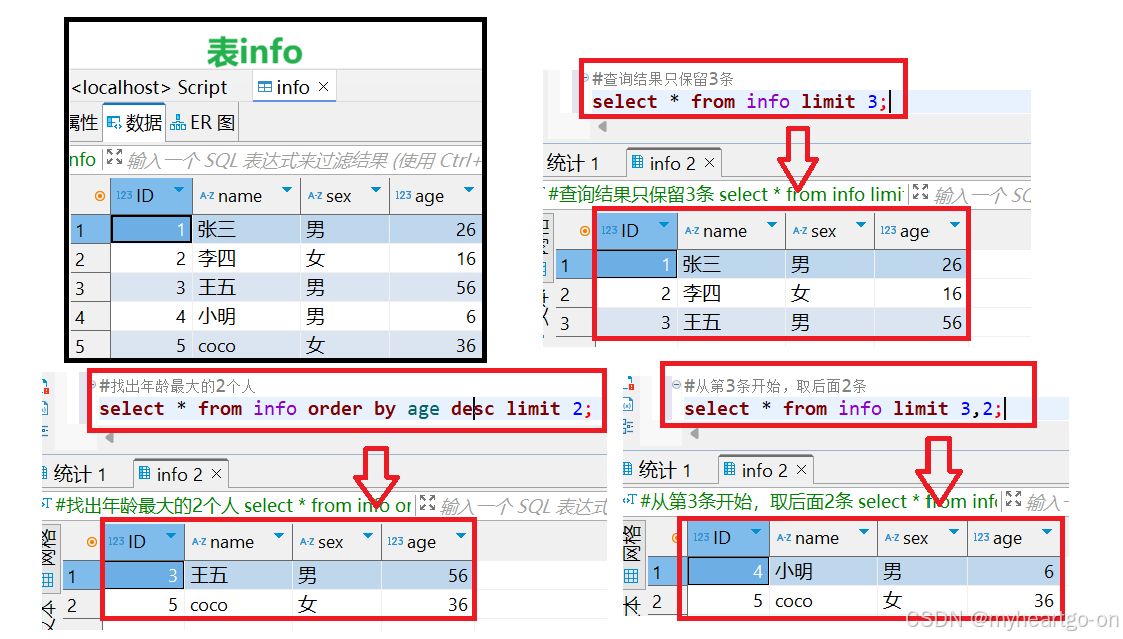
SQL入门
一、SQL 语言概述 数据库就是指数据存储的库,作用就是组织数据并存储数据,数据库如按照:库 -> 表 -> 数据三个层级进行数据组织,而 SQL 语言,就是一种对数据库、数据进行操作、管理、查询的工具,通过…...

Java中的Math类
关于Math类的介绍,这是一个在Java和其他许多编程语言中常见的内置库或模块,主要用于提供各种数学运算的方法。在Java中,Math类位于java.lang包下,它包含大量静态方法执行基本的数学函数,如三角函数、指数函数、对数函数…...

大厂常问iOS面试题–Runloop篇
大厂常问iOS面试题–Runloop篇 一.RunLoop概念 RunLoop顾名思义就是可以一直循环(loop)运行(run)的机制。这种机制通常称为“消息循环机制” NSRunLoop和CFRunLoopRef就是实现“消息循环机制”的对象。其实NSRunLoop本质是由CFRunLoopRef封装的,提供了面向对象的AP…...

【解决】mac报错“zsh: command not found: nvm”
问题描述: 安装nodejs时要先安装nvm,按照网上教程安装之后出现以下异常情况: 1.终端运行npm -v能查到版本,idea运行同样命令提示没找到,像是没安装一样 2.终端关闭重新打开之后,也像是没安装一样,需要重…...
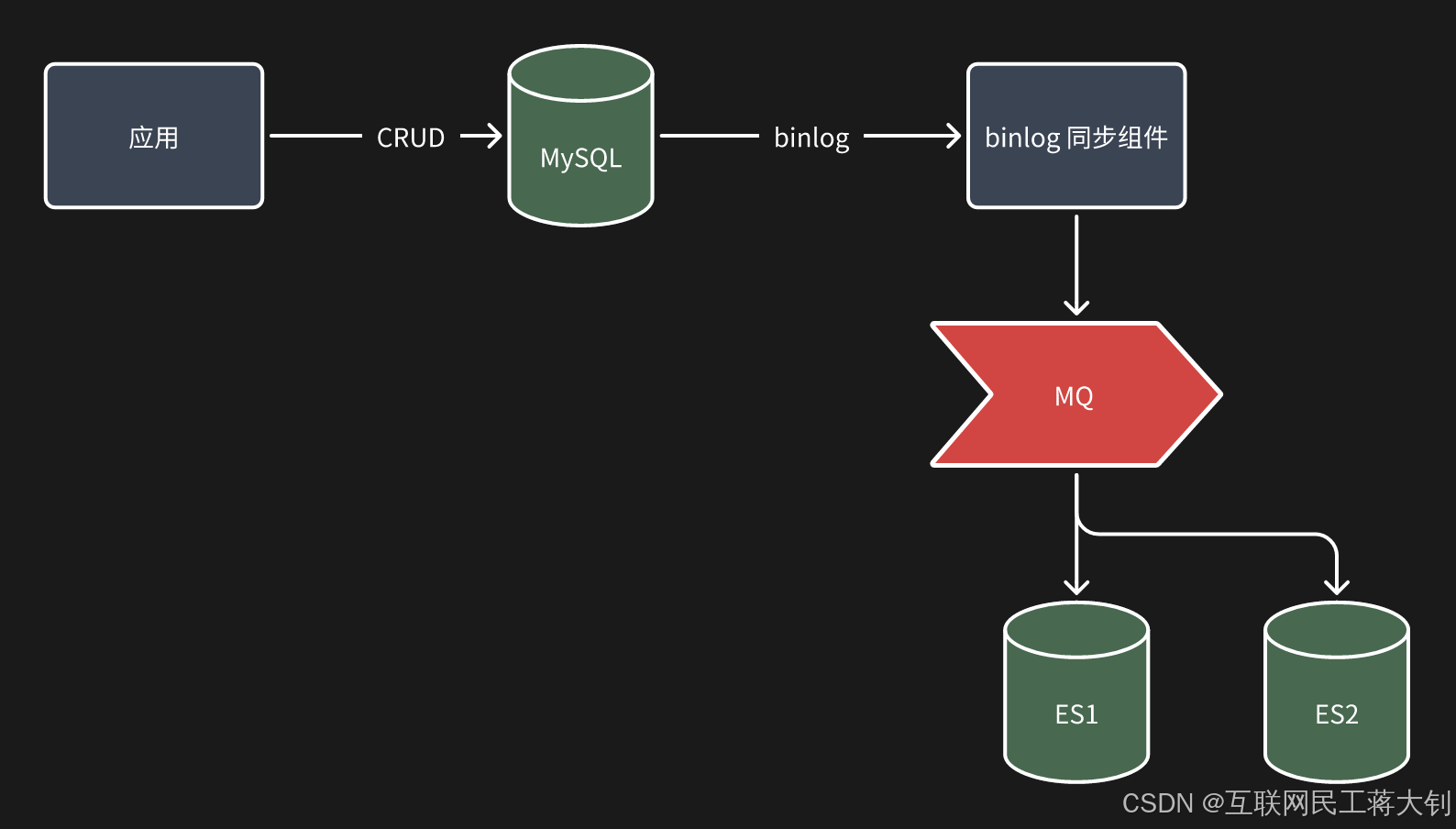
MySQL同步到ES的方案选型
文章目录 1. 同步双写优点缺点实现方式 2. 异步双写优点缺点实现方式 3. 另起应用 SQL 查询写入优点缺点实现方式 4. Binlog 实时同步优点缺点实现方式 5. 应用场景 本文参考: https://www.bilibili.com/video/BV13hvZeaErr/?vd_sourceb7e4d17fd13ffa91c4da6d37c08a6c7c 最近在…...

Transformer 与 CNN的对比
Transformer 相比于 CNN 的优点主要体现在以下几个方面: Transformer 相比 CNN 的优点: 全局依赖建模能力:Transformer 的核心机制是 自注意力机制,它可以直接建模输入序列中任意两个位置之间的依赖关系,无论它们之间的距离有多远。 相比之下,CNN 更擅长处理局部信息,它…...

多模态商品数据接口:融合图像、语音与文字的下一代商品详情体验
一、多模态商品数据接口的技术架构 (一)多模态数据融合引擎 跨模态语义对齐 通过Transformer架构实现图像、语音、文字的语义关联。例如,当用户上传一张“蓝色连衣裙”的图片时,接口可自动提取图像中的颜色(RGB值&…...

【论文笔记】若干矿井粉尘检测算法概述
总的来说,传统机器学习、传统机器学习与深度学习的结合、LSTM等算法所需要的数据集来源于矿井传感器测量的粉尘浓度,通过建立回归模型来预测未来矿井的粉尘浓度。传统机器学习算法性能易受数据中极端值的影响。YOLO等计算机视觉算法所需要的数据集来源于…...

sqlserver 根据指定字符 解析拼接字符串
DECLARE LotNo NVARCHAR(50)A,B,C DECLARE xml XML ( SELECT <x> REPLACE(LotNo, ,, </x><x>) </x> ) DECLARE ErrorCode NVARCHAR(50) -- 提取 XML 中的值 SELECT value x.value(., VARCHAR(MAX))…...

20个超级好用的 CSS 动画库
分享 20 个最佳 CSS 动画库。 它们中的大多数将生成纯 CSS 代码,而不需要任何外部库。 1.Animate.css 一个开箱即用型的跨浏览器动画库,可供你在项目中使用。 2.Magic Animations CSS3 一组简单的动画,可以包含在你的网页或应用项目中。 3.An…...

SQL慢可能是触发了ring buffer
简介 最近在进行 postgresql 性能排查的时候,发现 PG 在某一个时间并行执行的 SQL 变得特别慢。最后通过监控监观察到并行发起得时间 buffers_alloc 就急速上升,且低水位伴随在整个慢 SQL,一直是 buferIO 的等待事件,此时也没有其他会话的争抢。SQL 虽然不是高效 SQL ,但…...

python爬虫——气象数据爬取
一、导入库与全局配置 python 运行 import json import datetime import time import requests from sqlalchemy import create_engine import csv import pandas as pd作用: 引入数据解析、网络请求、时间处理、数据库操作等所需库。requests:发送 …...
通过 Ansible 在 Windows 2022 上安装 IIS Web 服务器
拓扑结构 这是一个用于通过 Ansible 部署 IIS Web 服务器的实验室拓扑。 前提条件: 在被管理的节点上安装WinRm 准备一张自签名的证书 开放防火墙入站tcp 5985 5986端口 准备自签名证书 PS C:\Users\azureuser> $cert New-SelfSignedCertificate -DnsName &…...
深入解析光敏传感技术:嵌入式仿真平台如何重塑电子工程教学
一、光敏传感技术的物理本质与系统级实现挑战 光敏电阻作为经典的光电传感器件,其工作原理根植于半导体材料的光电导效应。当入射光子能量超过材料带隙宽度时,价带电子受激发跃迁至导带,形成电子-空穴对,导致材料电导率显著提升。…...

Java多线程实现之Runnable接口深度解析
Java多线程实现之Runnable接口深度解析 一、Runnable接口概述1.1 接口定义1.2 与Thread类的关系1.3 使用Runnable接口的优势 二、Runnable接口的基本实现方式2.1 传统方式实现Runnable接口2.2 使用匿名内部类实现Runnable接口2.3 使用Lambda表达式实现Runnable接口 三、Runnabl…...

MLP实战二:MLP 实现图像数字多分类
任务 实战(二):MLP 实现图像多分类 基于 mnist 数据集,建立 mlp 模型,实现 0-9 数字的十分类 task: 1、实现 mnist 数据载入,可视化图形数字; 2、完成数据预处理:图像数据维度转换与…...
