PyMOL中常用的命令列表
PyMOL中常用的命令列表
- PyMOL中常用的命令列表,包括了加载文件、去除水分子、改变颜色、显示样式和图形优化等操作,可以帮助你完成全方位的分子展示设置。
基础命令流程
-
加载分子结构
load your_file.pdb # 加载PDB文件 -
去除水分子
remove solvent # 移除所有溶剂分子,包括水 # 或者 remove resn HOH # 仅移除水分子(PDB文件中水分子通常标记为 HOH) -
分子显示样式:选择分子结构的展示样式
show cartoon # 显示为卡通样式(适用于蛋白质) show sticks # 显示为棒状模型 show spheres # 显示为球状模型 hide everything # 隐藏所有显示 -
变换颜色:为分子各部分赋予五颜六色的效果
spectrum count, rainbow # 使用彩虹色渐变效果 color blue, chain A # 为链 A 着蓝色 color red, chain B # 为链 B 着红色 util.cbc # 随机为各链分配颜色 spectrum b, blue_white_red, minimum=10, maximum=50 # 使用温度因子渐变 -
分子选择:对分子结构的不同区域进行选择
select backbone, name CA+C+N+O # 选择骨架原子 select sidechain, not backbone # 选择侧链 -
调整分子显示:对选定的分子区域应用不同的显示样式
show sticks, sidechain # 侧链显示为棒状 show spheres, backbone # 骨架显示为球状 -
优化图像显示:改善图像质量和显示效果
set cartoon_fancy_helices, 1 # 优化卡通模式中的α螺旋显示 set ray_trace_mode, 1 # 启用光线追踪模式 set antialias, 2 # 抗锯齿处理,提高图像质量 -
保存图片或场景:将生成的图像导出为图片文件
ray 800, 600 # 生成高分辨率的图片(光线追踪渲染) png my_image.png, dpi=300 # 导出图片,设置为300dpi
示例:完整操作流程
以下是一个典型的完整操作流程示例,用于生成一个五颜六色、去除水分子的蛋白质分子结构图:
load your_file.pdb # 加载PDB文件
remove resn HOH # 去掉水分子
spectrum count, rainbow # 设置五颜六色的渐变
show cartoon # 选择卡通样式
util.cbc # 随机分配颜色
set cartoon_fancy_helices, 1 # 优化卡通模式中的螺旋显示
set ray_trace_mode, 1 # 启用光线追踪模式
set antialias, 2 # 开启抗锯齿
ray 1200, 800 # 渲染高分辨率图像
png my_image.png, dpi=300 # 保存图片,设置分辨率为300dpi
常用的其他命令
-
旋转和移动:手动旋转或移动分子
rotate x, 90 # 绕X轴旋转90度 translate [10, 0, 0] # 沿X轴移动10单位 -
测量距离:测量两个原子之间的距离
distance my_dist, name CA, resi 1 and name CB # 计算特定原子间的距离
在PyMOL中,可以将小分子和蛋白质的相互作用(例如氢键、疏水作用)清晰地展示出来,并且标记相关的名称和距离。以下是实现这一效果的完整命令流程:
基础流程
-
加载蛋白质和小分子结构文件:首先加载PDB文件。这里假设蛋白质和小分子分别存储在不同的PDB文件中。
load protein.pdb, protein # 加载蛋白质文件,并命名为“protein” load ligand.pdb, ligand # 加载小分子文件,并命名为“ligand” -
显示小分子和蛋白质的不同样式:可以用不同样式展示蛋白质和小分子,使其更易识别。
show cartoon, protein # 蛋白质显示为卡通模式 color cyan, protein # 为蛋白质设置颜色为青色 show sticks, ligand # 小分子显示为棒状模型 color yellow, ligand # 为小分子设置颜色为黄色 -
选择关键残基或与小分子相互作用的区域:选择小分子与蛋白质之间的相互作用区域(例如,5 Å内的原子)。
select binding_site, br. protein within 5 of ligand # 选择与小分子5 Å内的蛋白质原子 show sticks, binding_site # 绑定位点显示为棒状 color magenta, binding_site # 将绑定位点的颜色设置为紫色 -
添加相互作用连线:检测氢键和疏水作用,并自动添加连线。
distance hbonds, protein, ligand, mode=2 # 计算氢键,显示为连线 set dash_width, 3 # 设置连线宽度 set dash_color, red # 设置连线颜色为红色 -
标记小分子和残基名称:标记小分子名称和关键残基,以便更好地了解相互作用。
label ligand, resn # 为小分子标记名称(通常为其残基名) label binding_site, resn + resi # 为关键残基标记名称和序号 -
测量并显示距离:标记小分子和蛋白质之间的距离,以量化相互作用强度。
distance interaction_distances, binding_site, ligand set dash_gap, 0.5 # 调整连线间隔 set dash_color, green # 设置距离标记的连线颜色为绿色
完整操作流程
以下是一个完整的PyMOL命令序列示例,将小分子与蛋白质对接的相互作用展示出来,包括连线、颜色和名称标记:
load protein.pdb, protein # 加载蛋白质
load ligand.pdb, ligand # 加载小分子show cartoon, protein # 蛋白质为卡通模式
color cyan, protein # 蛋白质颜色设为青色
show sticks, ligand # 小分子为棒状模型
color yellow, ligand # 小分子颜色设为黄色select binding_site, br. protein within 5 of ligand # 选择绑定位点
show sticks, binding_site # 显示绑定位点为棒状
color magenta, binding_site # 绑定位点颜色设为紫色distance hbonds, protein, ligand, mode=2 # 添加氢键连线
set dash_width, 3 # 设置连线宽度
set dash_color, red # 氢键连线颜色设为红色label ligand, resn # 标记小分子名称
label binding_site, resn + resi # 标记绑定位点的残基名称和序号distance interaction_distances, binding_site, ligand # 显示距离
set dash_gap, 0.5 # 调整连线间隔
set dash_color, green # 距离连线颜色设为绿色
提示
-
手动添加相互作用连线:如果自动计算连线不够准确,可以手动添加。选择两个原子并计算它们的距离,如:
distance manual_dist, (ligand and name C1), (protein and resi 50 and name O)
以下是一个完整的PyMOL操作流程,包含加载文件、去除水分子、设置颜色、展示小分子和蛋白质的相互作用、标记名称、添加透明背景以及保存超清4K图像的所有步骤。所有操作步骤将以命令行的方式写出,方便直接复制到PyMOL中执行。
完整PyMOL操作命令
# 1. 加载文件
load protein.pdb, protein # 加载蛋白质文件
load ligand.pdb, ligand # 加载小分子文件# 2. 移除水分子
remove solvent # 移除所有溶剂分子,包括水
# 或者仅移除水分子
remove resn HOH# 3. 设置显示样式和颜色
show cartoon, protein # 将蛋白质显示为卡通模式
color cyan, protein # 蛋白质颜色设置为青色
show sticks, ligand # 将小分子显示为棒状
color yellow, ligand # 小分子颜色设置为黄色# 4. 选择与小分子有相互作用的区域
select binding_site, br. protein within 5 of ligand # 选择与小分子5 Å内的蛋白质原子
show sticks, binding_site # 将绑定位点显示为棒状
color magenta, binding_site # 设置绑定位点的颜色为紫色# 5. 添加相互作用连线
distance hbonds, protein, ligand, mode=2 # 自动添加氢键连线
set dash_width, 3 # 设置连线宽度
set dash_color, red # 氢键连线颜色设置为红色# 6. 标记小分子和关键残基的名称
label ligand, resn # 标记小分子名称
label binding_site, resn + resi # 标记关键残基的名称和序号# 7. 显示并标记距离
distance interaction_distances, binding_site, ligand # 计算并显示距离
set dash_gap, 0.5 # 调整距离连线间隔
set dash_color, green # 距离连线颜色设置为绿色# 8. 设置透明背景
bg_color white # 设置背景颜色为白色
set ray_opaque_background, 0 # 设置背景为透明# 9. 优化图像质量
set cartoon_fancy_helices, 1 # 优化卡通模式中的α螺旋显示
set ray_trace_mode, 1 # 启用光线追踪模式
set antialias, 2 # 开启抗锯齿处理,提升图像质量# 10. 保存超清4K图像
ray 3840, 2160 # 渲染4K分辨率(3840x2160)的图像
png output_image.png, dpi=300 # 保存渲染结果为PNG格式,分辨率为300dpi
步骤解析
- 加载文件:使用
load命令分别加载蛋白质和小分子文件,并命名为protein和ligand。 - 去除水分子:使用
remove solvent或remove resn HOH以去除水分子。 - 设置显示样式和颜色:设置蛋白质为卡通样式,小分子为棒状模型,并为两者选择不同的颜色。
- 选择相互作用位点:选择与小分子距离5 Å内的蛋白质原子区域,显示为棒状并设为紫色。
- 添加相互作用连线:添加氢键连线,使用红色表示,设置连线宽度为3。
- 标记名称:标记小分子和蛋白质关键残基的名称,以便查看相互作用的具体信息。
- 显示距离:计算小分子与蛋白质间的距离,并用绿色标记距离连线。
- 设置透明背景:将背景设置为白色并设为透明,以便导出图像适应各种背景。
- 优化图像质量:启用光线追踪模式、抗锯齿和高级卡通样式,使图像更美观。
- 保存4K图像:渲染4K分辨率图像,并导出为PNG格式,设置为300dpi,确保图像清晰度适合打印或展示。
相关文章:

PyMOL中常用的命令列表
PyMOL中常用的命令列表 PyMOL中常用的命令列表,包括了加载文件、去除水分子、改变颜色、显示样式和图形优化等操作,可以帮助你完成全方位的分子展示设置。 基础命令流程 加载分子结构 load your_file.pdb # 加载PDB文件去除水分子 remove solvent …...
)
坏块处理 ORA-01578: ORACLE data block corrupted (file # 3, block # 152588)
帮客户检查环境时,发现sysaux表空间的数据文件有坏块,8月25日发生的,备份保留3个月,直接恢复处理。 rman备份log报错如下 RMAN-00571: RMAN-00569: ERROR MESSAGE STACK FOLLOWS RMAN-00571: RMAN-03009: failure of backu…...

像`npm i`作为`npm install`的简写一样,使用`pdm i`作为`pdm install`的简写
只需安装插件pdm-plugin-i即可: pdm plugin add pdm-plugin-i 然后就可以愉快地pdm i了,例如: git clone https://github.com/waketzheng/fast-dev-cli cd fast-dev-cli python -m pip install --user pipx pipx install pdm pdm plugin a…...

DNS域名解析服务器--RHCE
1.DNS简介 DNS ( Domain Name System )是互联网上的一项服务,它作为将域名和 IP 地址相互映射的一个分布式 数据库,能够使人更方便的访问互联网 DNS 系统使用的是网络的查询,那么自然需要有监听的 port 。 DNS 使用的是…...

数据库物化视图的工作原理与Java实现
引言 物化视图(Materialized View)是数据库中一种特殊的对象,它存储了查询结果的物理副本,使得复杂查询的结果可以快速地被访问。本文将详细介绍物化视图的工作原理、技术策略,并提供Java代码示例。 1. 物化视图的基…...

炫酷的登录框!(附源码)
大家想看什么前端效果请留言 预览效果 源码 <!DOCTYPE html> <html lang"zh"> <head><meta charset"UTF-8"><meta name"viewport" content"widthdevice-width, initial-scale1.0"><title>登录页…...

使用Python实现智能生态系统监测与保护的深度学习模型
随着人类活动的增加,生态系统受到的威胁也在不断加剧。为了更好地保护我们的生态环境,智能生态系统监测与保护成为了一项重要的任务。通过深度学习技术,我们可以实现生态系统的自动化监测与管理,从而及时发现和应对环境变化。本文将详细介绍如何使用Python构建一个深度学习…...

Rust 力扣 - 54. 螺旋矩阵
文章目录 题目描述题解思路题解代码题目链接 题目描述 题解思路 我们只需要一圈一圈的从外向内遍历矩阵,每一圈遍历顺序为上边、右边、下边、左边 我们需要注意的是如果上边与下边重合或者是右边与左边重合,我们只需要遍历上边、右边即可 题解代码 i…...
)
Flutter 简述(1)
Flutter 简述 简述 Flutter是Google开源的应用框架,只要一套代码兼顾Android、iOS、Web、Windows、macOS和Linux六个平台,它的设计思路可以说更加先进,不像ReactNative每个组件都需要有对应的原生组件实现,而是通过skia或者其他…...
BGP实验--BGP路由反射器
AR1、AR2上的Loopback 1接口分别为10.1.1.1/24、10.2.2.2/24,用于模拟用户网段 所有设备均使用Loopback 0地址为BGP Router ID,AR1与AR2、AR2与AR3、AR3与AR4、AR4与AR2之间基于直连接口建立IBGP对等体关系,其中AR1为AR2的路由反射器客户端&a…...
域渗透-域环境部署
01-域渗透部署 一、工作组和域 1、为什么需要域 在早期Windows主机都是属于工作组网络,单独的个体,在企业环境中,针对于工作组网络的计算机要达到统一管理相当麻烦,为实现将一个企业中所有的用户和计算机进行集中管理ÿ…...

【Oracle】空格单字符通配符查询匹配失败
问题 在进行模糊查询的时候,通过全局任意字符串匹配出含有两个字刘姓的人,但是通过刘_不能匹配出结果。 解决 检查后发现,姓名中包含空格 SELECT * FROM student WHERE TRIM(sname) LIKE 刘_;第一种解决方案就是查询的时候进行去空格处理&a…...

uniapp实现中间平滑凸起tabbar
uniapp实现中间平滑凸起tabbar 背景实现思路代码实现尾巴 背景 在移动端开发中,tabar是一个使用频率很高的组件,几乎是每个APP都会用到。今天给大家分享一个中间平滑凸起的tabbar组件,有需要的可以做下参考。先上图镇楼: 实现思…...

【视频】OpenCV:识别颜色、绘制轮廓
1、安装OpenCV库 sudo apt install libopencv-dev2、链接库 将 OpenCV 头文件路径和库添加到CMake中,在 CMakeLists.txt 中添加 1)查找库 find_package(OpenCV REQUIRED) 或者 find_package(OpenCV REQUIRED core imgproc highgui) 2)添加头文件路径 include_directories…...

C++_STL_xx_番外01_关于STL的总结(常见容器的总结;关联式容器分类及特点;二叉树、二叉搜索树、AVL树(平衡二叉搜索树)、B树、红黑树)
文章目录 1. 常用容器总结2. 关联式容器分类3. 二叉树、二叉搜索树、AVL树、B树、红黑树 1. 常用容器总结 针对常用容器的一些总结: 2. 关联式容器分类 关联式容器分为两大类: 基于红黑树的set和map;基于hash表的unorder_set和unorder_ma…...

xlrd.biffh.XLRDError: Excel xlsx file; not supported
文章目录 一、问题报错二、报错原因三、解决思路四、解决方法 一、问题报错 在处理Excel文件时,特别是当我们使用Python的xlrd库来读取.xlsx格式的文件,偶尔会遇到这样一个错误:“xlrd.biffh.XLRDError: Excel xlsx file; not supported”。…...

ENNSP中ACL的实验配置
ACL:访问控制列表 1访问控制----在路由器的入或者出的接口上,匹配流量,之后产生动作---允许或拒绝 2.定义感兴趣流量-----帮助其他软件抓流量 访问控制的匹配规则: 拓扑图如下 基础配置 基础配置弄好后,随便p…...

数字后端零基础入门系列 | Innovus零基础LAB学习Day8
###LAB15 Detail Routing for Signal Integrity, Timing, Power and Design for Yield 这个章节虽然标题有点长,但不要被它吓到,其实这个章节就是Innovus工具的绕线Routing。只不过这个阶段做Route不是仅仅是把所有的逻辑连接,用实际的金属层…...

【AI开源项目】FastGPT- 快速部署FastGPT以及使用知识库的两种方式!
文章目录 一、FastGPT大模型介绍1. 开发团队2. 发展史3. 基本概念 二、FastGPT与其他大模型的对比三、使用 Docker Compose 快速部署 FastGPT1、安装 Docker 和 Docker Compose(1). 安装 Docker(2). 安装 Docker Composeÿ…...

信息学科平台系统开发:Spring Boot实用指南
3系统分析 3.1可行性分析 通过对本基于保密信息学科平台系统实行的目的初步调查和分析,提出可行性方案并对其一一进行论证。我们在这里主要从技术可行性、经济可行性、操作可行性等方面进行分析。 3.1.1技术可行性 本基于保密信息学科平台系统采用Spring Boot框架&a…...

iOS 26 携众系统重磅更新,但“苹果智能”仍与国行无缘
美国西海岸的夏天,再次被苹果点燃。一年一度的全球开发者大会 WWDC25 如期而至,这不仅是开发者的盛宴,更是全球数亿苹果用户翘首以盼的科技春晚。今年,苹果依旧为我们带来了全家桶式的系统更新,包括 iOS 26、iPadOS 26…...

ubuntu搭建nfs服务centos挂载访问
在Ubuntu上设置NFS服务器 在Ubuntu上,你可以使用apt包管理器来安装NFS服务器。打开终端并运行: sudo apt update sudo apt install nfs-kernel-server创建共享目录 创建一个目录用于共享,例如/shared: sudo mkdir /shared sud…...
vscode(仍待补充)
写于2025 6.9 主包将加入vscode这个更权威的圈子 vscode的基本使用 侧边栏 vscode还能连接ssh? debug时使用的launch文件 1.task.json {"tasks": [{"type": "cppbuild","label": "C/C: gcc.exe 生成活动文件"…...

使用van-uploader 的UI组件,结合vue2如何实现图片上传组件的封装
以下是基于 vant-ui(适配 Vue2 版本 )实现截图中照片上传预览、删除功能,并封装成可复用组件的完整代码,包含样式和逻辑实现,可直接在 Vue2 项目中使用: 1. 封装的图片上传组件 ImageUploader.vue <te…...

论文浅尝 | 基于判别指令微调生成式大语言模型的知识图谱补全方法(ISWC2024)
笔记整理:刘治强,浙江大学硕士生,研究方向为知识图谱表示学习,大语言模型 论文链接:http://arxiv.org/abs/2407.16127 发表会议:ISWC 2024 1. 动机 传统的知识图谱补全(KGC)模型通过…...

拉力测试cuda pytorch 把 4070显卡拉满
import torch import timedef stress_test_gpu(matrix_size16384, duration300):"""对GPU进行压力测试,通过持续的矩阵乘法来最大化GPU利用率参数:matrix_size: 矩阵维度大小,增大可提高计算复杂度duration: 测试持续时间(秒&…...
12.找到字符串中所有字母异位词
🧠 题目解析 题目描述: 给定两个字符串 s 和 p,找出 s 中所有 p 的字母异位词的起始索引。 返回的答案以数组形式表示。 字母异位词定义: 若两个字符串包含的字符种类和出现次数完全相同,顺序无所谓,则互为…...

什么?连接服务器也能可视化显示界面?:基于X11 Forwarding + CentOS + MobaXterm实战指南
文章目录 什么是X11?环境准备实战步骤1️⃣ 服务器端配置(CentOS)2️⃣ 客户端配置(MobaXterm)3️⃣ 验证X11 Forwarding4️⃣ 运行自定义GUI程序(Python示例)5️⃣ 成功效果
聊一聊接口测试的意义有哪些?
目录 一、隔离性 & 早期测试 二、保障系统集成质量 三、验证业务逻辑的核心层 四、提升测试效率与覆盖度 五、系统稳定性的守护者 六、驱动团队协作与契约管理 七、性能与扩展性的前置评估 八、持续交付的核心支撑 接口测试的意义可以从四个维度展开,首…...
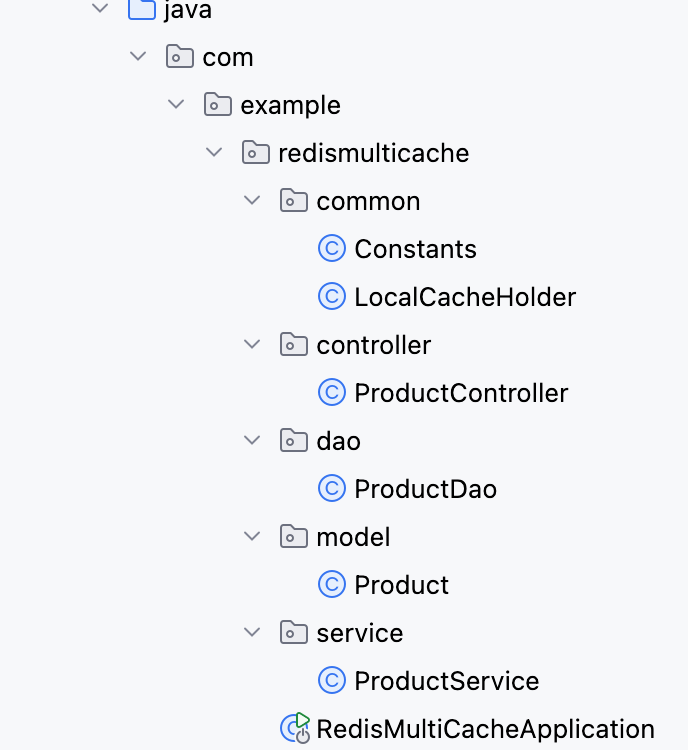
【Redis】笔记|第8节|大厂高并发缓存架构实战与优化
缓存架构 代码结构 代码详情 功能点: 多级缓存,先查本地缓存,再查Redis,最后才查数据库热点数据重建逻辑使用分布式锁,二次查询更新缓存采用读写锁提升性能采用Redis的发布订阅机制通知所有实例更新本地缓存适用读多…...
