基于qiime2的16S数据分析全流程:从导入数据到下游分析一条龙
目录
创建metadata
把数据导入qiime2
去除引物序列
双端合并 (dada2不需要)
质控 (dada2不需要)
使用deblur获得特征序列
使用dada2生成代表序列与特征表
物种鉴定
可视化物种鉴定结果
构建进化树(ITS一般不构建进化树)
生成多样性分析所需数据
α多样性分析
beta多样性分析
绘制稀释曲线
差异物种分析
如何把.qza格式文件导出?比如把特征表导出
演示单端数据导入qiime2


创建metadata


把数据导入qiime2
去除引物序列
双端合并 (dada2不需要)
质控 (dada2不需要)
使用deblur获得特征序列

使用dada2生成代表序列与特征表
物种鉴定
可视化物种鉴定结果


构建进化树(ITS一般不构建进化树)


生成多样性分析所需数据


α多样性分析

beta多样性分析

绘制稀释曲线

差异物种分析
如何把.qza格式文件导出?比如把特征表导出
演示单端数据导入qiime2

相关文章:

基于qiime2的16S数据分析全流程:从导入数据到下游分析一条龙
目录 创建metadata 把数据导入qiime2 去除引物序列 双端合并 (dada2不需要) 质控 (dada2不需要) 使用deblur获得特征序列 使用dada2生成代表序列与特征表 物种鉴定 可视化物种鉴定结果 构建进化树(ITS一般不构建进化树…...
)
【软件测试开发】:软件测试常用函数1.0(C++)
1. 元素的定位 web⾃动化测试的操作核⼼是能够找到⻚⾯对应的元素,然后才能对元素进⾏具体的操作。 常⻅的元素定位⽅式⾮常多,如id,classname,tagname,xpath,cssSelector 常⽤的主要由cssSelector和xpath…...

vue2项目修改浏览器显示的网页图标
1.准备一个新的图标文件,通常是. ico格式,也可以是. Png、. Svg等格式 2.将新的图标文件(例如:faviconAt.png)放入项目的public文件夹中。如下图 public文件夹中的所有文件都会在构建时原样复制到最终的输出目录(通常是dist) 3. 修改vue项目…...

开源、创新与人才发展:机器人产业的战略布局与稚晖君成功案例解析
目录 引言 一、开源:机器人产业的战略布局 促进技术进步和生态建设 吸引人才和合作伙伴 建立标准和网络效应 降低研发风险与成本 二、稚晖君:华为"天才少年计划"的成功典范 深厚的技术积累与动手能力 强烈的探索和创新意识 持续公开…...

线程相关作业
1.创建两个线程,分支线程1拷贝文件的前一部分,分支线程2拷贝文件的后一部分 #include "head.h"#define BUFFER_SIZE 1024// 线程参数结构体,包含文件名和文件偏移量 typedef struct {FILE *src_file;FILE *dest_file;long start_o…...

通义万相2.1开源版本地化部署攻略,生成视频再填利器
2025 年 2 月 25 日晚上 11:00 通义万相 2.1 开源发布,前两周太忙没空搞它,这个周末,也来本地化部署一个,体验生成效果如何,总的来说,它在国内文生视频、图生视频的行列处于领先位置,…...

【模拟CMOS集成电路设计】带隙基准(Bandgap)设计与仿真(基于运放的电流模BGR)
【模拟CMOS集成电路设计】带隙基准(Bandgap)设计与仿真 前言工程文件&部分参数计算过程,私聊~ 一、 设计指标指标分析: 二、 电路分析三、 仿真3.1仿真电路图3.2仿真结果(1)运放增益(2)基准温度系数仿真(3)瞬态启动仿真(4)静态…...

如何选择国产串口屏?
目录 1、迪文 2、淘晶驰 3、广州大彩 4、金玺智控 5、欣瑞达 6、富莱新 7、冠显 8、有彩 串口屏,顾名思义,就是通过串口通信接口(如RS232、RS485、TTL UART等)与主控设备进行通信的显示屏。其核心功能是显示信息和接收输入…...

Solana中的程序派生地址(PDAs):是什么,为什么,以及如何?
程序派生地址 (PDA) 在 Solana 中的应用:什么、为什么和如何? 在学习 Solana 时,你会经常听到关于 程序派生地址 (PDAs) 的讨论。它们就像这样 —— 强大、多功能,而且最重要的是,稍微被误解。如果你是一个开发者&…...

利用FatJar彻底解决Jar包冲突(一)
利用FatJar彻底解决Jar包冲突 序FatJar的加载与隔离⼀、 FatJar概念⼆、FatJar的加载三、FatJar的隔离四、隔离机制验证五、 FatJar的定位六、 打包注意点 序 今天整理旧电脑里的资料,偶然翻到大概10年前实习时写的笔记,之前经常遇到Java依赖冲突的问题…...

Spring MVC笔记
01 什么是Spring MVC Spring MVC 是 Spring 框架中的一个核心模块,专门用于构建 Web 应用程序。它基于经典的 MVC 设计模式(Model-View-Controller),但通过 Spring 的特性(如依赖注入、注解驱动)大幅简化了…...

BurpSuite插件jsEncrypter使用教程
一、前言 在当今Web应用安全测试中,前端加密已成为开发者保护敏感数据的常用手段。然而,这也给安全测试人员带来了挑战,传统的抓包方式难以获取明文数据,测试效率大打折扣。BurpSuite作为一款强大的Web安全测试工具,其…...

【C#实现手写Ollama服务交互,实现本地模型对话】
前言 C#手写Ollama服务交互,实现本地模型对话 最近使用C#调用OllamaSharpe库实现Ollama本地对话,然后思考着能否自己实现这个功能。经过一番查找,和查看OllamaSharpe源码发现确实可以。其实就是开启Ollama服务后,发送HTTP请求&a…...

Android Glide 框架线程管理模块原理的源码级别深入分析
一、引言 在现代的 Android 应用开发中,图片加载是一个常见且重要的功能。Glide 作为一款广泛使用的图片加载框架,以其高效、灵活和易用的特点受到了开发者的青睐。其中,线程管理模块是 Glide 框架中至关重要的一部分,它负责协调…...

每天记录一道Java面试题---day32
MySQL索引的数据结构、各自优劣 回答重点 B树:是一个平衡的多叉树,从根节点到每个叶子节点的高度差不超过1,而且同层级的节点间有指针相互连接。在B树上的常规检索,从根节点到叶子节点的搜索效率基本相当,不会出现大…...

Vue3 Pinia 符合直觉的Vue.js状态管理库
Pinia 符合直觉的Vue.js状态管理库 什么时候使用Pinia 当两个关系非常远的组件,要传递参数时使用Pinia组件的公共参数使用Pinia...

深度学习与大模型基础-向量
大家好!今天我们来聊聊向量(Vector)。别被这个词吓到,其实向量在我们的生活中无处不在,只是我们没注意罢了。 1. 向量是什么? 简单来说,向量就是有大小和方向的量。比如你从家走到学校&#x…...

【网络编程】完成端口 IOCP
10.11 完成端口 10.11.1 基本概念 完成端口的全称是I/O 完成端口,英文为IOCP(I/O Completion Port) 。IOCP是一个异 步I/O 的 API, 可以高效地将I/O 事件通知给应用程序。与使用select() 或是其他异步方法不同 的是,一个套接字与一个完成端口关联了起来…...

《苍穹外卖》SpringBoot后端开发项目重点知识整理(DAY1 to DAY3)
目录 一、在本地部署并启动Nginx服务1. 解压Nginx压缩包2. 启动Nginx服务3. 验证Nginx是否启动成功: 二、导入接口文档1. 黑马程序员提供的YApi平台2. YApi Pro平台3. 推荐工具:Apifox 三、Swagger1. 常用注解1.1 Api与ApiModel1.2 ApiModelProperty与Ap…...

管理网络安全
防火墙在 Linux 系统安全中有哪些重要的作用? 防火墙作为网络安全的第一道防线,能够根据预设的规则,对进出系统的网络流量进行严格筛选。它可以阻止未经授权的外部访问,只允许符合规则的流量进入系统,从而保护系统免受…...

【大模型RAG】拍照搜题技术架构速览:三层管道、两级检索、兜底大模型
摘要 拍照搜题系统采用“三层管道(多模态 OCR → 语义检索 → 答案渲染)、两级检索(倒排 BM25 向量 HNSW)并以大语言模型兜底”的整体框架: 多模态 OCR 层 将题目图片经过超分、去噪、倾斜校正后,分别用…...
)
Java入门学习详细版(一)
大家好,Java 学习是一个系统学习的过程,核心原则就是“理论 实践 坚持”,并且需循序渐进,不可过于着急,本篇文章推出的这份详细入门学习资料将带大家从零基础开始,逐步掌握 Java 的核心概念和编程技能。 …...
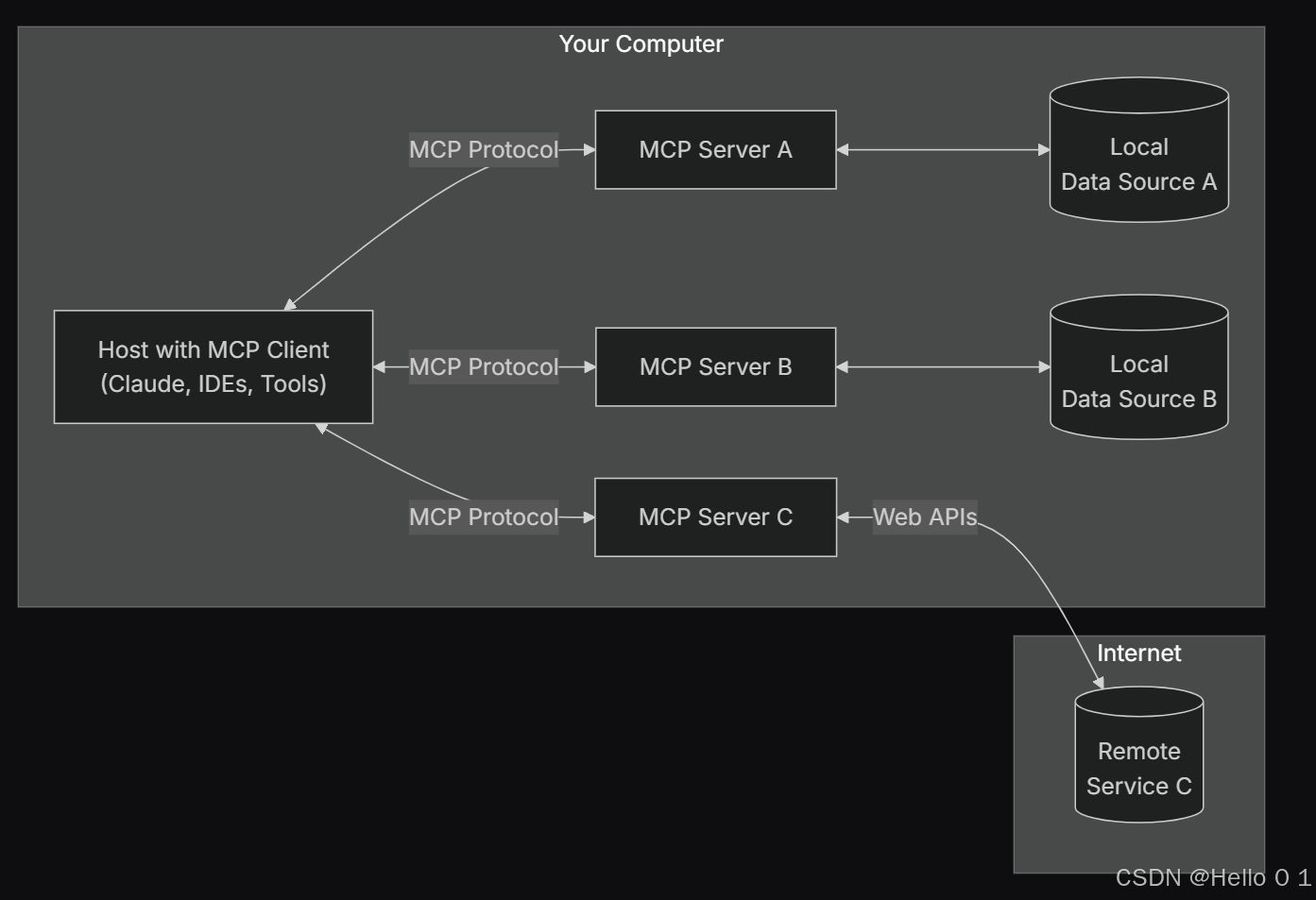
让AI看见世界:MCP协议与服务器的工作原理
让AI看见世界:MCP协议与服务器的工作原理 MCP(Model Context Protocol)是一种创新的通信协议,旨在让大型语言模型能够安全、高效地与外部资源进行交互。在AI技术快速发展的今天,MCP正成为连接AI与现实世界的重要桥梁。…...

PAN/FPN
import torch import torch.nn as nn import torch.nn.functional as F import mathclass LowResQueryHighResKVAttention(nn.Module):"""方案 1: 低分辨率特征 (Query) 查询高分辨率特征 (Key, Value).输出分辨率与低分辨率输入相同。"""def __…...
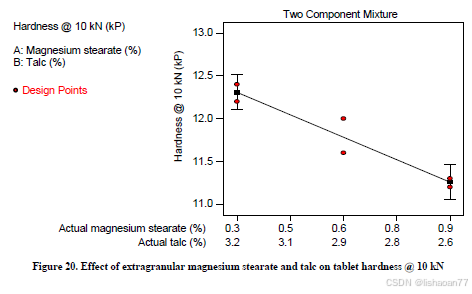
R语言速释制剂QBD解决方案之三
本文是《Quality by Design for ANDAs: An Example for Immediate-Release Dosage Forms》第一个处方的R语言解决方案。 第一个处方研究评估原料药粒径分布、MCC/Lactose比例、崩解剂用量对制剂CQAs的影响。 第二处方研究用于理解颗粒外加硬脂酸镁和滑石粉对片剂质量和可生产…...
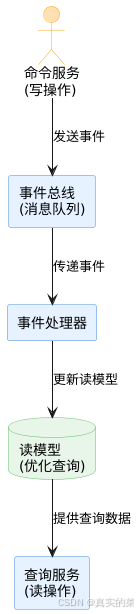
消息队列系统设计与实践全解析
文章目录 🚀 消息队列系统设计与实践全解析🔍 一、消息队列选型1.1 业务场景匹配矩阵1.2 吞吐量/延迟/可靠性权衡💡 权衡决策框架 1.3 运维复杂度评估🔧 运维成本降低策略 🏗️ 二、典型架构设计2.1 分布式事务最终一致…...
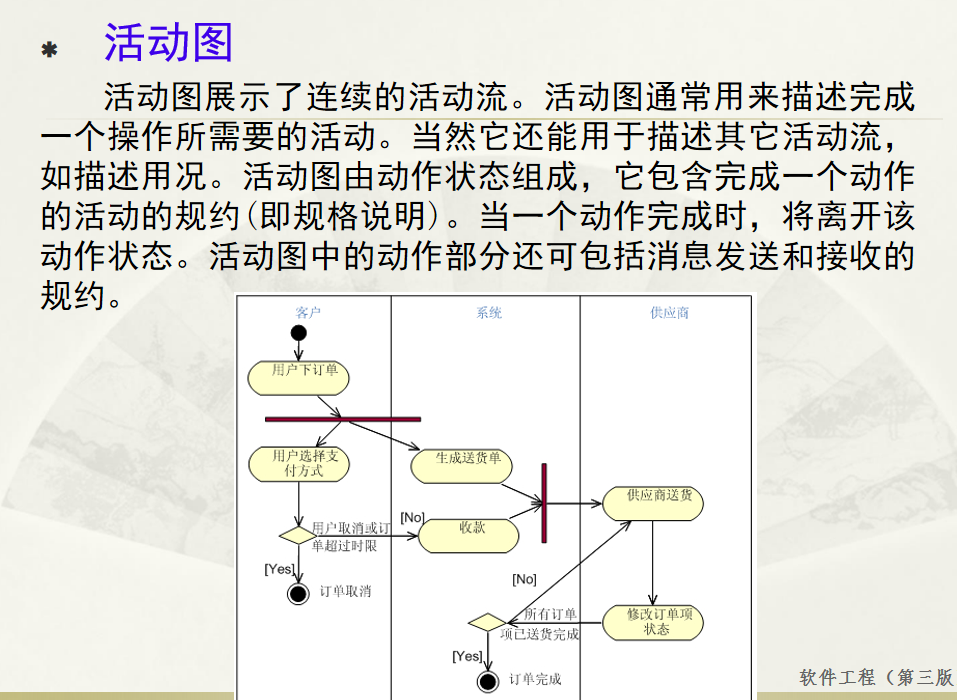
软件工程 期末复习
瀑布模型:计划 螺旋模型:风险低 原型模型: 用户反馈 喷泉模型:代码复用 高内聚 低耦合:模块内部功能紧密 模块之间依赖程度小 高内聚:指的是一个模块内部的功能应该紧密相关。换句话说,一个模块应当只实现单一的功能…...

Neko虚拟浏览器远程协作方案:Docker+内网穿透技术部署实践
前言:本文将向开发者介绍一款创新性协作工具——Neko虚拟浏览器。在数字化协作场景中,跨地域的团队常需面对实时共享屏幕、协同编辑文档等需求。通过本指南,你将掌握在Ubuntu系统中使用容器化技术部署该工具的具体方案,并结合内网…...

DAY 26 函数专题1
函数定义与参数知识点回顾:1. 函数的定义2. 变量作用域:局部变量和全局变量3. 函数的参数类型:位置参数、默认参数、不定参数4. 传递参数的手段:关键词参数5 题目1:计算圆的面积 任务: 编写一…...
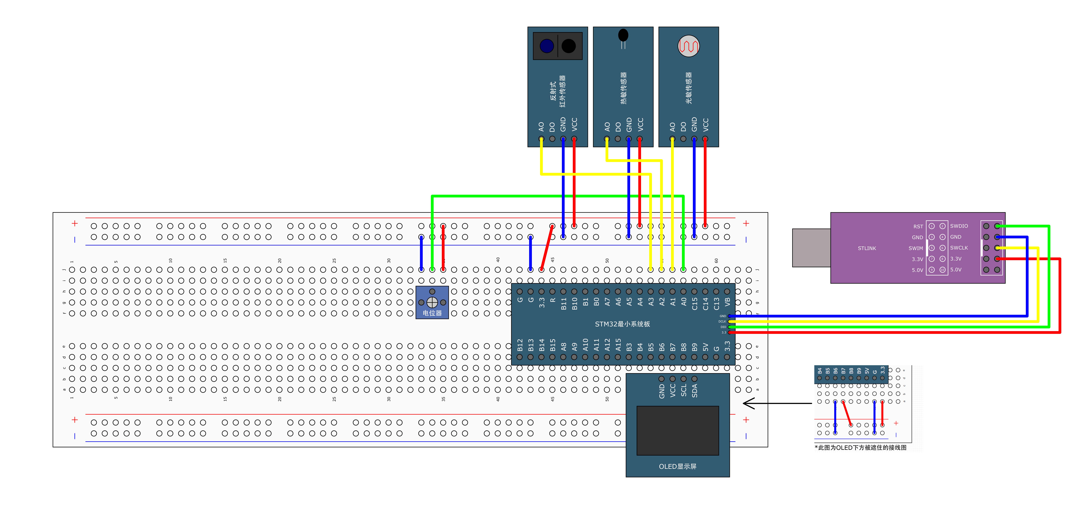
STM32标准库-ADC数模转换器
文章目录 一、ADC1.1简介1. 2逐次逼近型ADC1.3ADC框图1.4ADC基本结构1.4.1 信号 “上车点”:输入模块(GPIO、温度、V_REFINT)1.4.2 信号 “调度站”:多路开关1.4.3 信号 “加工厂”:ADC 转换器(规则组 注入…...
