【Python机器学习实战】 | 基于PCA主成分分析技术读入空气质量监测数据进行数据预处理并计算空气质量综合评测结果

🎩 欢迎来到技术探索的奇幻世界👨💻
📜 个人主页:@一伦明悦-CSDN博客
✍🏻 作者简介: C++软件开发、Python机器学习爱好者
🗣️ 互动与支持:💬评论 👍🏻点赞 📂收藏 👀关注+
如果文章有所帮助,欢迎留下您宝贵的评论,
点赞加收藏支持我,点击关注,一起进步!
引言
主成分分析(Principal Component Analysis, PCA)是一种常用的机器学习和数据分析技术,用于降低数据维度、识别数据中的模式、发现变量之间的关系等。以下是PCA技术的详细介绍:
基本概念:
- PCA旨在通过线性变换将原始数据投影到一个新的坐标系中,新坐标系中的坐标轴称为主成分。这些主成分按照方差大小递减的顺序排列,第一主成分具有最大的方差,第二主成分具有第二大的方差,依此类推。
数学原理:
- PCA通过计算数据的协方差矩阵或相关矩阵,然后对其进行特征值分解(或奇异值分解),以获得主成分和对应的特征向量。这些特征向量定义了新的坐标系,而数据投影到这些特征向量上形成的新坐标即为主成分。
应用步骤:
- 数据标准化:对原始数据进行标准化处理,使得不同维度的数据具有相同的尺度。
- 计算协方差矩阵:计算标准化后的数据的协方差矩阵。
- 特征值分解:对协方差矩阵进行特征值分解或奇异值分解,得到特征值和特征向量。
- 选择主成分:按照特征值的大小选择前k个主成分,这些主成分对应的特征向量构成了新的特征空间。
降维和数据压缩:
- PCA通过保留最重要的主成分(即方差较大的主成分)来实现数据的降维,从而减少数据的复杂度和存储空间,同时保留数据中的关键信息。
PCA的应用领域:
- 数据可视化:通过降维将高维数据可视化到二维或三维空间,帮助分析人员理解数据的结构。
- 特征提取:在机器学习中,PCA可以用来提取数据中最重要的特征,以便于后续的分类、回归或聚类任务。
- 噪声过滤:PCA可以过滤掉数据中的噪声成分,提高数据的质量和后续分析的准确性。
正文
01-对数据集进行主成分分析并计算各主成分的方差贡献率
这段代码主要进行了以下操作:
导入所需的模块:
- 导入了numpy(np)、pandas(pd)和matplotlib.pyplot(plt)等常用的数据处理和可视化模块。
- 导入了Axes3D和cm模块用于创建3D图形和设置颜色映射。
- 使用warnings模块过滤警告信息,避免在程序运行过程中输出警告。
- 设置matplotlib绘图显示中文字符的字体和解决负号显示问题。
- 导入了make_regression、make_circles、make_s_curve等数据集生成函数,以及train_test_split等模型选择相关函数。
- 导入FactorAnalyzer用于因子分析。
创建子图及设置图形参数:
- 使用plt.subplots创建1行2列的子图,设置子图的大小为15x6。
生成数据并进行可视化:
- 对于noise取30和10两种情况,分别生成包含50个样本观测点的数据集。
- 使用make_regression生成具有线性关系的数据集,其中包括一个特征和一个目标变量,同时受到指定程度的噪声干扰。
- 将生成的数据集绘制为散点图分布在两个子图中。
- 设置子图标题、横纵坐标标签、显示网格线,并在图中添加标注显示各特征的离散程度。
保存图形:
- 使用plt.savefig将绘制的图形保存为文件,文件名为"…/4.png",分辨率为500dpi。
总体来说,这段代码通过生成具有线性关系的带有噪声的数据集,并通过散点图可视化展示了各个特征的分布情况,同时对数据的离散程度进行了说明。并最终将绘制的图形保存为文件。
#本章需导入的模块
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from pylab import *
import matplotlib.cm as cm
import warnings
warnings.filterwarnings(action = 'ignore')
%matplotlib inline
plt.rcParams['font.sans-serif']=['SimHei'] #解决中文显示乱码问题
plt.rcParams['axes.unicode_minus']=False
from sklearn.datasets import make_regression,make_circles,make_s_curve
from sklearn.model_selection import train_test_split
from scipy.stats import multivariate_normal
from sklearn import decomposition
from factor_analyzer import FactorAnalyzerfig,axes=plt.subplots(nrows=1,ncols=2,figsize=(15,6))
N=50
for i,noise in enumerate([30,10]): X,Y=make_regression(n_samples=N,n_features=1,random_state=123,noise=noise,bias=0)X=np.hstack((X,Y.reshape(len(X),1)))axes[i].scatter(X[:,0],X[:,1],marker="o",s=50)axes[i].set_title("%d个样本观测点的分布"%N)axes[i].set_xlabel("X1")axes[i].set_ylabel("X2")axes[i].grid(True,linestyle='-.')axes[i].text(-3,75,"离散程度:X1=%.2f;\n X2=%.2f;"%(np.std(X[:,0])/np.mean(X[:,0]),np.std(X[:,1])/np.mean(X[:,1])),fontdict={'size':'12','color':'b'}) plt.savefig("../4.png", dpi=500) 运行结果如下图所示:

这段代码的作用是使用主成分分析(PCA)对生成的带有噪声的数据集进行降维,并可视化降维后的结果。
具体解释如下:
创建子图及设置图形参数:
fig, axes = plt.subplots(nrows=1, ncols=2, figsize=(15, 6)):创建包含1行2列的子图,每个子图的大小为15x6英寸。PCA模型的应用:
pca = decomposition.PCA(n_components=2, random_state=1):初始化PCA对象,指定降维后保留的主成分数量为2,并设置随机种子以确保结果的可重复性。生成数据并进行降维:
for i, noise in enumerate([30, 10])::循环两次,分别处理噪声为30和10的情况。X, Y = make_regression(n_samples=N, n_features=1, random_state=123, noise=noise, bias=0):生成包含50个样本观测点的数据集,其中特征数为1,同时受指定程度的噪声影响。X = np.hstack((X, Y.reshape(len(X), 1))):将生成的特征X和目标变量Y合并成一个数组。pca.fit(X):对数据集X进行PCA拟合,计算主成分和其它相关参数。获取PCA结果并绘图:
pca.singular_values_:获取PCA模型中的奇异值,即主成分的特征值。pca.components_:获取PCA模型的主成分(特征向量)。y = pca.transform(X):将原始数据集X转换到主成分空间,得到降维后的数据y。axes[i].scatter(y[:, 0], y[:, 1], marker="o", s=50):在第i个子图中绘制降维后的数据y的散点图,x轴为第一个主成分,y轴为第二个主成分。axes[i].set_title("%d个样本观测点的分布(方差贡献率:y1=%.3f,y2=%.3f)\n系数:%s"%(N, p1, p2, a)):设置子图标题,显示样本数量、每个主成分的方差贡献率、以及主成分系数。axes[i].set_xlabel("y1")和axes[i].set_ylabel("y2"):设置子图的x轴和y轴标签。axes[i].grid(True, linestyle='-.'):显示子图的网格线。保存图形:
plt.savefig("../4.png", dpi=500):将绘制的子图保存为文件,文件名为"…/4.png",分辨率为500dpi。总体来说,这段代码通过PCA将高维数据降至二维,并展示降维后的数据分布情况及主成分的贡献率和系数,最后将结果保存为图片文件。
fig,axes=plt.subplots(nrows=1,ncols=2,figsize=(15,6))
pca=decomposition.PCA(n_components=2,random_state=1)
for i,noise in enumerate([30,10]): X,Y=make_regression(n_samples=N,n_features=1,random_state=123,noise=noise,bias=0)X=np.hstack((X,Y.reshape(len(X),1)))pca.fit(X) p1=pca.singular_values_[0]/sum(pca.singular_values_) #p1=pca.explained_variance_ratio_[0]p2=pca.singular_values_[1]/sum(pca.singular_values_) #p2=pca.explained_variance_ratio_[1] a=pca.components_y=pca.transform(X)axes[i].scatter(y[:,0],y[:,1],marker="o",s=50)axes[i].set_title("%d个样本观测点的分布(方差贡献率:y1=%.3f,y2=%.3f)\n系数:%s"%(N,p1,p2,a))axes[i].set_xlabel("y1")axes[i].set_ylabel("y2")axes[i].grid(True,linestyle='-.')
plt.savefig("../4.png", dpi=500) 运行结果如下图所示:

02-核主成分分析
这段代码的作用是生成并可视化两个不同维度的数据集,包括二维平面和三维空间的样本分布。
具体解释如下:
导入必要的库:
import numpy as np: 导入数值计算库numpy。import pandas as pd: 导入数据处理库pandas(未在代码中使用)。import matplotlib.pyplot as plt: 导入绘图库matplotlib。from mpl_toolkits.mplot3d import Axes3D: 导入用于绘制3D图形的模块。from pylab import *: 导入pylab模块中的所有内容(未在代码中使用)。import matplotlib.cm as cm: 导入颜色映射模块。import warnings: 导入警告处理模块。warnings.filterwarnings(action='ignore'): 设置忽略警告。%matplotlib inline: 在Jupyter Notebook中使用matplotlib绘图时需要的魔法命令,用于在输出单元格中显示图形。plt.rcParams['font.sans-serif']=['SimHei']和plt.rcParams['axes.unicode_minus']=False:设置matplotlib以显示中文和解决负号显示问题。from sklearn.datasets import make_regression, make_circles, make_s_curve: 导入生成数据集的模块。from sklearn.model_selection import train_test_split: 导入数据集划分模块。from scipy.stats import multivariate_normal: 导入多元正态分布模块。from sklearn import decomposition: 导入降维算法PCA的模块。from factor_analyzer import FactorAnalyzer: 导入因子分析模块(未在代码中使用)。设置常量和生成二维数据:
N = 100: 设置生成数据集的样本数量为100。X, Y = make_circles(n_samples=N, noise=0.2, factor=0.5, random_state=123): 生成包含100个样本观测点的圆环形数据集,噪声为0.2,因子为0.5。绘制二维数据分布:
fig = plt.figure(figsize=(18, 6)): 创建图形对象,设置图形大小为18x6英寸。markers = ['^', 'o']: 定义两种不同的标记样式。ax = fig.add_subplot(121): 创建第一个子图,将其添加到图形中。- 循环绘制两个类别的数据点,根据类别使用不同的标记样式。
ax.set_title("100个样本观测点在二维空间中的分布"): 设置子图标题。ax.set_xlabel("y1")和ax.set_ylabel("y2"): 设置x轴和y轴标签。ax.grid(True, linestyle='-.'): 显示子图的网格线。生成三维数据并绘制分布:
ax = fig.add_subplot(122, projection='3d'): 创建第二个子图,使用3D投影。var = multivariate_normal(mean=[0,0], cov=[[1,0],[0,1]]): 定义二维正态分布的参数。- 将二维数据与正态分布的概率密度函数值结合,形成一个包含三个特征的数据集。
- 使用循环绘制两个类别的数据点,根据类别使用不同的标记样式。
ax.set_xlabel('X1'),ax.set_ylabel('X2'),ax.set_zlabel('X3'): 设置三维坐标轴的标签。ax.set_title('三维空间下100个样本观测点的分布'): 设置子图标题。保存图形:
plt.savefig("../4.png", dpi=500): 将绘制的整个图形保存为文件,文件名为"…/4.png",分辨率为500dpi。总体来说,这段代码通过使用make_circles生成二维数据和multivariate_normal生成三维数据,展示了不同维度空间下数据分布的可视化效果,并保存为图片文件。
#本章需导入的模块
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from pylab import *
import matplotlib.cm as cm
import warnings
warnings.filterwarnings(action = 'ignore')
%matplotlib inline
plt.rcParams['font.sans-serif']=['SimHei'] #解决中文显示乱码问题
plt.rcParams['axes.unicode_minus']=False
from sklearn.datasets import make_regression,make_circles,make_s_curve
from sklearn.model_selection import train_test_split
from scipy.stats import multivariate_normal
from sklearn import decomposition
from factor_analyzer import FactorAnalyzerN=100
X,Y=make_circles(n_samples=N,noise=0.2,factor=0.5,random_state=123)
fig = plt.figure(figsize=(18,6))
markers=['^','o']
ax = fig.add_subplot(121)
for k,m in zip([1,0],markers):ax.scatter(X[Y==k,0],X[Y==k,1],marker=m,s=50)
ax.set_title("100个样本观测点在二维空间中的分布")
ax.set_xlabel("y1")
ax.set_ylabel("y2")
ax.grid(True,linestyle='-.')
ax = fig.add_subplot(122, projection='3d')
var = multivariate_normal(mean=[0,0], cov=[[1,0],[0,1]])Z=np.zeros((len(X),))
for i,x in enumerate(X):Z[i]=var.pdf(x)
X=np.hstack((X,Z.reshape(len(X),1)))
for k,m in zip([1,0],markers):ax.scatter(X[Y==k,0],X[Y==k,1],X[Y==k,2],marker=m,s=40)
ax.set_xlabel('X1')
ax.set_ylabel('X2')
ax.set_zlabel('X3')
ax.set_title('三维空间下100个样本观测点的分布')
plt.savefig("../4.png", dpi=500) 运行结果如下图所示:

这段代码实现了核主成分分析(Kernel PCA),并将结果可视化。下面是详细解释:
设置图形大小:
plt.figure(figsize=(15, 6)): 创建一个新的图形对象,并设置图形大小为15x6英寸。定义核函数类型:
kernels = ['linear', 'rbf']: 定义了两种核函数类型,线性核和径向基函数(RBF)核。进行核主成分分析和可视化:
for i, kernel in enumerate(kernels):: 循环处理每种核函数类型。
kpca = decomposition.KernelPCA(n_components=2, kernel=kernel): 创建一个核主成分分析对象,指定将数据映射到2维空间,并选择当前循环的核函数类型。kpca.fit(X): 对原始数据X进行核主成分分析拟合。y = kpca.transform(X): 将原始数据映射到降维后的2维空间。输出方差贡献率:
print('方差贡献率(%s):%s'%(kernel,kpca.lambdas_/sum(kpca.lambdas_))): 输出每种核函数类型下,每个主成分的方差贡献率。绘制子图:
plt.subplot(1, 2, i+1): 在1x2的子图中,选择第i+1个子图进行绘制。for k, m in zip([1, 0], markers):: 循环绘制两个类别的数据点。
plt.scatter(y[Y==k, 0], y[Y==k, 1], marker=m, s=50): 根据降维后的数据y和类别Y,绘制散点图,使用不同的标记样式m表示不同的类别。plt.grid(True, linestyle='-.'): 显示子图的网格线。plt.title('核主成分分析结果(核函数:%s)' % kernel): 设置子图的标题,指明当前使用的核函数类型。保存图形:
plt.savefig("../4.png", dpi=500): 将绘制的整个图形保存为文件,文件名为"…/4.png",分辨率为500dpi。这段代码通过核主成分分析对原始数据
X进行降维,并利用不同核函数类型(线性和RBF)展示了降维后的数据分布情况。每个子图显示了相应核函数下的数据点分布,同时保存整个图形为文件。
plt.figure(figsize=(15,6))
kernels=['linear','rbf']
for i,kernel in enumerate(kernels):kpca=decomposition.KernelPCA(n_components=2,kernel=kernel)kpca.fit(X)y=kpca.transform(X)print('方差贡献率(%s):%s'%(kernel,kpca.lambdas_/sum(kpca.lambdas_)))plt.subplot(1,2,i+1)for k,m in zip([1,0],markers):plt.scatter(y[Y==k,0],y[Y==k,1],marker=m,s=50)plt.grid(True,linestyle='-.')plt.title('核主成分分析结果(核函数:%s)'%kernel)
plt.savefig("../4.png", dpi=500) 运行结果如下图所示:

这段代码用于生成一个三维 S 形曲面数据集,并将其可视化为三维散点图,并保存为图片。
生成 S 形曲面数据集:
X, t = make_s_curve(n_samples=8000, noise=0, random_state=123): 使用make_s_curve函数生成一个包含8000个样本的 S 形曲面数据集。X是特征数据,t是对应的颜色标签(在此处用来绘制不同颜色的数据点)。创建图形对象:
fig = plt.figure(figsize=(12, 6)): 创建一个新的图形对象,设置图形大小为12x6英寸。添加三维子图:
ax = fig.add_subplot(111, projection='3d'): 在图形对象中添加一个三维子图,使用投影类型为3D。绘制三维散点图:
color = np.array(t): 将颜色标签t转换为numpy数组,用于散点的颜色映射。ax.scatter(X[:, 0], X[:, 1], X[:, 2], s=8, color=plt.cm.Spectral(color)): 绘制三维散点图。X[:, 0],X[:, 1],X[:, 2]分别表示数据集中的三个特征维度,s=8指定散点的大小,color=plt.cm.Spectral(color)根据颜色标签t使用彩色映射来绘制不同颜色的数据点。设置坐标轴标签:
ax.set_xlabel("X1"),ax.set_ylabel("X2"),ax.set_zlabel("X3"): 设置三维子图的 x、y、z 轴标签为 “X1”、“X2”、“X3”。保存图形:
plt.savefig("../4.png", dpi=500): 将绘制的三维散点图保存为文件,文件名为"…/4.png",分辨率为500dpi。这段代码通过使用
make_s_curve函数生成的 S 形曲面数据集,利用三维散点图展示了数据点的分布情况,并将图形保存为文件。
X,t=make_s_curve(n_samples=8000, noise=0, random_state=123)
fig = plt.figure(figsize=(12,6))
#ax = Axes3D(fig)
ax = fig.add_subplot(111, projection='3d')
color=np.array(t)
ax.scatter(X[:,0],X[:,1],X[:,2],s=8,color=plt.cm.Spectral(color))
ax.set_xlabel("X1")
ax.set_ylabel("X2")
ax.set_zlabel("X3")
plt.savefig("../4.png", dpi=500)运行结果如下图所示:

这段代码创建了一个包含三个子图的图形,并在每个子图中绘制了二维散点图,展示了数据集在不同维度的投影关系。最终将整个图形保存为文件。
创建包含三个子图的图形:
fig, axes = plt.subplots(nrows=1, ncols=3, figsize=(20, 6)): 创建一个具有一行三列的子图布局,总共包含三个子图,设置图形大小为20x6英寸。fig是整个图形对象,axes是包含三个子图的数组。在第一个子图绘制 X1-X2 的散点图:
axes[0].scatter(X[:, 0], X[:, 1], s=8, color=plt.cm.Spectral(color)): 在第一个子图中绘制 X1-X2 的二维散点图。数据集的第一列作为X轴,第二列作为Y轴,s=8指定散点的大小,color=plt.cm.Spectral(color)根据颜色标签t使用彩色映射来绘制不同颜色的数据点。axes[0].set_xlabel("X1"): 设置第一个子图的 x 轴标签为 “X1”。axes[0].set_ylabel("X2"): 设置第一个子图的 y 轴标签为 “X2”。在第二个子图绘制 X1-X3 的散点图:
axes[1].scatter(X[:, 0], X[:, 2], s=8, color=plt.cm.Spectral(color)): 在第二个子图中绘制 X1-X3 的二维散点图。数据集的第一列作为X轴,第三列作为Y轴,s=8指定散点的大小,color=plt.cm.Spectral(color)使用彩色映射来绘制不同颜色的数据点。axes[1].set_xlabel("X1"): 设置第二个子图的 x 轴标签为 “X1”。axes[1].set_ylabel("X3"): 设置第二个子图的 y 轴标签为 “X3”。在第三个子图绘制 X2-X3 的散点图:
axes[2].scatter(X[:, 1], X[:, 2], s=8, color=plt.cm.Spectral(color)): 在第三个子图中绘制 X2-X3 的二维散点图。数据集的第二列作为X轴,第三列作为Y轴,s=8指定散点的大小,color=plt.cm.Spectral(color)使用彩色映射来绘制不同颜色的数据点。axes[2].set_xlabel("X2"): 设置第三个子图的 x 轴标签为 “X2”。axes[2].set_ylabel("X3"): 设置第三个子图的 y 轴标签为 “X3”。保存图形:
plt.savefig("../4.png", dpi=500): 将包含三个子图的图形保存为文件,文件名为"…/4.png",分辨率为500dpi。这段代码将数据集在不同维度的投影关系通过三个二维散点图展示了出来,并将整个图形保存为文件。
fig,axes=plt.subplots(nrows=1,ncols=3,figsize=(20,6))
axes[0].scatter(X[:,0],X[:,1],s=8,color=plt.cm.Spectral(color))
axes[0].set_xlabel("X1")
axes[0].set_ylabel("X2")
axes[1].scatter(X[:,0],X[:,2],s=8,color=plt.cm.Spectral(color))
axes[1].set_xlabel("X1")
axes[1].set_ylabel("X3")
axes[2].scatter(X[:,1],X[:,2],s=8,color=plt.cm.Spectral(color))
axes[2].set_xlabel("X2")
axes[2].set_ylabel("X3")
plt.savefig("../4.png", dpi=500) 运行结果如下图所示 :

这段代码执行了核主成分分析(Kernel PCA),并将结果可视化在三个不同的子图中,每个子图使用不同的核函数进行分析。
数据准备:
tmp = X[:, [0, 2]]: 从原始数据集X中选择第1列和第3列特征作为新的数据集tmp,用于后续的主成分分析。定义核函数列表:
kernels = ['linear', 'rbf', 'poly']: 定义了三种核函数类型,分别是线性核(linear)、高斯径向基函数核(rbf)、多项式核(poly)。创建图形对象:
plt.figure(figsize=(15, 4)): 创建一个图形对象,设置图形大小为15x4英寸。循环执行主成分分析和可视化:
for i, kernel in enumerate(kernels)::遍历三种核函数类型。核主成分分析:
kpca = decomposition.KernelPCA(n_components=2, kernel=kernel): 使用当前循环的核函数类型kernel创建一个 KernelPCA 对象,指定将数据降至二维。kpca.fit(tmp): 对数据集tmp进行核主成分分析,拟合模型。y = kpca.transform(tmp): 将数据集tmp转换到降维后的二维空间,结果存储在y中。绘制子图:
plt.subplot(1, 3, i+1): 在图形对象中创建一个一行三列的子图布局,并定位到当前循环的位置。plt.scatter(y[:, 0], y[:, 1], s=8, color=plt.cm.Spectral(color)): 在当前子图中绘制主成分分析后的二维散点图。y[:, 0]和y[:, 1]分别表示降维后的第一和第二主成分,s=8指定散点的大小,color=plt.cm.Spectral(color)根据颜色标签t使用彩色映射来绘制不同颜色的数据点。plt.title("核主成分分析(核函数=%s)" % kernel): 设置当前子图的标题,显示当前使用的核函数类型。plt.xlabel("第1主成分"): 设置当前子图的 x 轴标签。plt.ylabel("第2主成分"): 设置当前子图的 y 轴标签。打印方差贡献率:
print('方差贡献率(%s):%s' % (kernel, kpca.lambdas_ / sum(kpca.lambdas_))):计算并打印出当前核函数下每个主成分的方差贡献率,kpca.lambdas_包含了每个主成分的方差。这段代码的目的是比较不同核函数在核主成分分析下的效果,通过降维后的二维散点图展示数据在主成分空间的分布情况,并输出每个主成分的方差贡献率。
tmp=X[:,[0,2]]
kernels=['linear','rbf','poly']
plt.figure(figsize=(15,4))
for i,kernel in enumerate(kernels):kpca=decomposition.KernelPCA(n_components=2,kernel=kernel)kpca.fit(tmp)y=kpca.transform(tmp)plt.subplot(1,3,i+1)plt.scatter(y[:,0],y[:,1],s=8,color=plt.cm.Spectral(color))plt.title("核主成分分析(核函数=%s)"%kernel)plt.xlabel("第1主成分")plt.ylabel("第2主成分") print('方差贡献率(%s):%s'%(kernel,kpca.lambdas_/sum(kpca.lambdas_)))运行结果如下图所示 :

这段代码是进行不同核函数下的核主成分分析(Kernel PCA),并将结果可视化在一个包含四个子图的图形中。
设置图形大小:
plt.figure(figsize=(12, 8)): 创建一个图形对象,设置图形大小为12x8英寸。定义核函数列表:
kernels = ['linear', 'rbf', 'poly', 'sigmoid']: 定义了四种核函数类型,包括线性核(linear)、高斯径向基函数核(rbf)、多项式核(poly)和sigmoid核。循环执行主成分分析和可视化:
for i, kernel in enumerate(kernels)::遍历四种核函数类型。核主成分分析:
kpca = decomposition.KernelPCA(n_components=2, kernel=kernel): 使用当前循环的核函数类型kernel创建一个 KernelPCA 对象,指定将数据降至二维。kpca.fit(X): 对完整的原始数据集X进行核主成分分析,拟合模型。y = kpca.transform(X): 将整个数据集X转换到降维后的二维空间,结果存储在y中。绘制子图:
plt.subplot(2, 2, i+1): 在图形对象中创建一个两行两列的子图布局,并定位到当前循环的位置。plt.scatter(y[:, 0], y[:, 1], s=8, color=plt.cm.Spectral(color)): 在当前子图中绘制主成分分析后的二维散点图。y[:, 0]和y[:, 1]分别表示降维后的第一和第二主成分,s=8指定散点的大小,color=plt.cm.Spectral(color)根据颜色标签t使用彩色映射来绘制不同颜色的数据点。plt.title("不同核函数下的主成分分析(核函数:%s)\n方差贡献率%s:" % (kernel, kpca.lambdas_ / sum(kpca.lambdas_))): 设置当前子图的标题,显示当前使用的核函数类型以及每个主成分的方差贡献率。plt.subplots_adjust(hspace=0.5): 调整子图之间的垂直间距,增加可读性。通过这段代码,可以直观地比较不同核函数在核主成分分析下的效果,了解数据在主成分空间中的分布情况以及各主成分的贡献率。
plt.figure(figsize=(12,8))
kernels=['linear','rbf','poly','sigmoid']
for i,kernel in enumerate(kernels):kpca=decomposition.KernelPCA(n_components=2,kernel=kernel)kpca.fit(X)y=kpca.transform(X)plt.subplot(2,2,i+1)plt.scatter(y[:,0],y[:,1],s=8,color=plt.cm.Spectral(color))plt.title("不同核函数下的主成分分析(核函数:%s)\n方差贡献率%s:"%(kernel,kpca.lambdas_/sum(kpca.lambdas_)))plt.subplots_adjust(hspace=0.5)运行结果如下图所示 :

03-基于主成分分析的因子分析
这段代码执行了基于主成分分析的因子分析,并进行了可视化。让我来详细解释每个步骤:
导入必要的模块:
- 导入了用于数学计算和数据操作的模块,如NumPy和Pandas。
- 导入了用于绘图的Matplotlib模块和其子模块,以及设置中文显示的相关配置。
- 导入了生成数据和模型选择所需的模块,如
make_s_curve用于生成S形曲线样本数据。- 导入了用于因子分析的FactorAnalyzer类。
生成数据集:
X, t = make_s_curve(n_samples=8000, noise=0, random_state=123): 生成了包含8000个样本的S形曲线数据集,t是每个样本的颜色标签。数据预处理:
color = np.array(t): 将颜色标签转换为NumPy数组。X = pd.DataFrame(X): 将数据集转换为DataFrame格式(可选)。R = X.corr(): 计算数据集的样本相关性矩阵R。计算因子载荷矩阵:
eig_value, eigvector = np.linalg.eig(R): 计算样本相关性矩阵R的特征值和特征向量。- 对特征值进行排序和处理,得到因子载荷矩阵
factorM,它是特征向量与特征值开平方的乘积。计算因子值系数:
score = np.linalg.inv(R) * factorM: 计算因子值系数,即特征值的逆乘以因子载荷矩阵。因子分析:
fa = FactorAnalyzer(method='principal', n_factors=2, rotation=None): 使用主成分法进行因子分析,设定因子数量为2。fa.fit(X): 拟合因子分析模型,获取因子载荷矩阵和相关统计信息。- 输出因子载荷矩阵、变量共同度以及因子的方差贡献率等。
可视化:
y = fa.transform(X): 将原始数据集X转换到由两个主成分构成的因子空间中。plt.scatter(y.T[0], y.T[1], s=8, color=plt.cm.Spectral(color)): 绘制因子空间中的散点图,其中每个点的颜色根据color数组确定。- 添加标题和轴标签,并保存图像到文件中。
这段代码的主要目的是展示如何通过主成分分析进行因子分析,分析数据集中变量之间的关系,并将结果可视化以便观察数据在因子空间中的分布情况。
#本章需导入的模块
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from pylab import *
import matplotlib.cm as cm
import warnings
warnings.filterwarnings(action = 'ignore')
%matplotlib inline
plt.rcParams['font.sans-serif']=['SimHei'] #解决中文显示乱码问题
plt.rcParams['axes.unicode_minus']=False
from sklearn.datasets import make_regression,make_circles,make_s_curve
from sklearn.model_selection import train_test_split
from scipy.stats import multivariate_normal
from sklearn import decomposition
from factor_analyzer import FactorAnalyzerX,t=make_s_curve(n_samples=8000, noise=0, random_state=123)
color=np.array(t)
X=pd.DataFrame(X)
R=X.corr() #样本相关性矩阵
eig_value, eigvector = np.linalg.eig(R)#求矩阵R的全部特征值,特征向量。
sortkey,eig=list(eig_value.argsort()),list(eig_value) #按升序排序
eig.sort()
eig.reverse()
sortkey.reverse()A = np.zeros((eigvector.shape[1],eigvector.shape[1]))
for i,e in enumerate(eig):A[i,:]=np.sqrt(e)*eigvector[:,sortkey[i]]
factorM=A.T
print("因子载荷矩阵:\n{0}".format(factorM)) score=np.linalg.inv(R)*factorM
print("因子值系数:\n{0}".format(score))fa = FactorAnalyzer(method='principal',n_factors=2,rotation=None)
fa.fit(X)
print("因子载荷矩阵:\n", fa.loadings_)
print("\n变量共同度:\n", fa.get_communalities())
tmp=fa.get_factor_variance()
print("因子的方差贡献:{0}".format(tmp[0]))
print("因子的方差贡献率:{0}".format(tmp[1]))
print("因子的累计方差贡献率:{0}".format(tmp[2]))
y=fa.transform(X)
plt.scatter(y.T[0],y.T[1],s=8,color=plt.cm.Spectral(color))
plt.title("基于主成分分析的因子分析")
plt.xlabel("因子f1")
plt.ylabel("因子f2")
plt.savefig("../4.png", dpi=500)
plt.show()运行结果如下图所示:

这段代码是在之前基于主成分分析的因子分析基础上,进行了旋转后的因子分析,并对结果进行了可视化。让我来详细解释每个步骤:
旋转后的因子分析设置:
fa = FactorAnalyzer(method='principal', n_factors=2, rotation='varimax'): 创建一个因子分析对象fa,使用主成分法进行因子分析,设定因子数量为2,并选择了Varimax旋转方法 (rotation='varimax'),用于旋转因子载荷矩阵以提高解释性。拟合模型:
fa.fit(X): 将数据集X应用于因子分析模型,拟合模型以获取旋转后的因子载荷矩阵和相关统计信息。输出结果:
print("\n变量共同度:\n", fa.get_communalities()): 打印出变量的共同度,即每个变量与所有因子的相关性的平方和。print("旋转后的因子载荷矩阵:\n", fa.loadings_): 打印旋转后的因子载荷矩阵,显示了每个变量与每个因子的载荷(相关性)。因子方差贡献分析:
tmp = fa.get_factor_variance(): 计算因子的方差贡献率相关统计信息。print("因子的方差贡献:{0}".format(tmp[0])): 打印因子的方差贡献。print("因子的方差贡献率:{0}".format(tmp[1])): 打印因子的方差贡献率,即每个因子解释的总方差的百分比。print("因子的累计方差贡献率:{0}".format(tmp[2])): 打印因子的累计方差贡献率,即所有因子累计解释的总方差的百分比。数据转换和可视化:
y = fa.transform(X): 将原始数据集X转换为因子空间中的坐标,即每个样本在新的因子空间中的位置。plt.scatter(y.T[0], y.T[1], s=8, color=plt.cm.Spectral(color)): 绘制因子空间中的散点图,其中每个点的颜色由color数组确定。plt.title("基于主成分分析的因子分析(旋转)"): 设置图像标题。plt.xlabel("因子f1"): 设置X轴标签。plt.ylabel("因子f2"): 设置Y轴标签。plt.savefig("../4.png", dpi=500): 将图像保存为文件,分辨率为500dpi。plt.show(): 显示图像。这段代码的主要目的是展示在应用Varimax旋转后的因子分析中,如何解释数据集中变量之间的结构,并通过可视化观察数据在旋转后因子空间中的分布情况。
fa = FactorAnalyzer(method='principal',n_factors=2,rotation='varimax')
fa.fit(X)
print("\n变量共同度:\n", fa.get_communalities())
print("旋转后的因子载荷矩阵:\n", fa.loadings_)
tmp=fa.get_factor_variance()
print("因子的方差贡献:{0}".format(tmp[0]))
print("因子的方差贡献率:{0}".format(tmp[1]))
print("因子的累计方差贡献率:{0}".format(tmp[2]))
y=fa.transform(X)
plt.scatter(y.T[0],y.T[1],s=8,color=plt.cm.Spectral(color))
plt.title("基于主成分分析的因子分析(旋转)")
plt.xlabel("因子f1")
plt.ylabel("因子f2")
plt.savefig("../4.png", dpi=500)
plt.show()运行结果如下图所示:

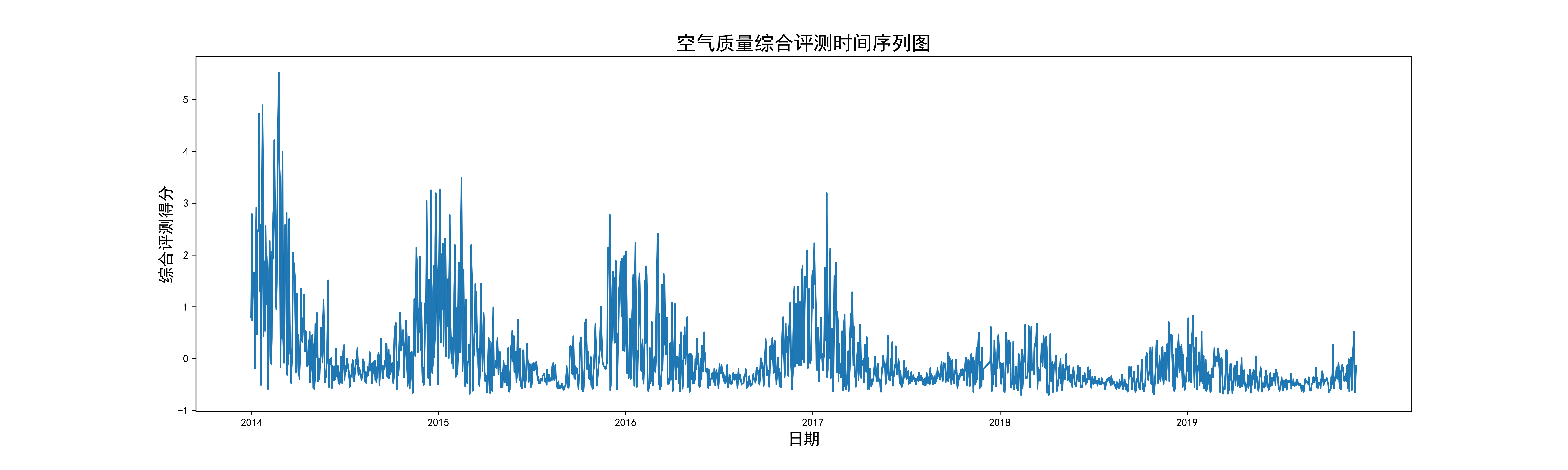
04-基于主成分分析对空气质量监测数据进行分析
这段代码的作用是进行因子分析(Factor Analysis)并输出相关统计结果。下面是详细解释:
数据导入和预处理:
import语句导入所需的库和模块,包括数据处理、可视化和警告管理的工具。- 从Excel文件中读取数据,并进行一些预处理步骤,如将0值替换为NaN,然后删除包含NaN的行,确保数据的完整性和质量。
数据准备和因子分析:
X = data.iloc[:, 3:-1]:选择数据中的特征变量列,通常排除了非预测变量和目标变量。fa = FactorAnalyzer(method='principal', n_factors=2, rotation='varimax'):创建一个因子分析对象,指定使用主成分方法、2个因子,并应用Varimax旋转以促进因子的解释性。因子分析模型拟合和结果输出:
fa.fit(X):对选择的数据进行因子分析拟合。- 打印输出以下统计量:
- 因子载荷矩阵 (
fa.loadings_):显示每个变量与每个因子之间的相关性强度。- 变量共同度 (
fa.get_communalities()):给出每个变量的共同度,即它们可以通过公共因子解释的方差比例。- 因子的方差贡献 (
tmp[0]):描述因子解释的总方差。- 因子的方差贡献率 (
tmp[1]):各个因子解释的方差比例。- 因子的累计方差贡献率 (
tmp[2]):前几个因子累计解释的总方差比例,用于评估模型解释数据变异程度的能力。综上所述,这段代码的目的是利用因子分析技术来探索数据集中潜在的因子结构,并提供关于每个因子及其解释能力的详细统计信息。
#本章需导入的模块
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from pylab import *
import matplotlib.cm as cm
import warnings
warnings.filterwarnings(action = 'ignore')
%matplotlib inline
plt.rcParams['font.sans-serif']=['SimHei'] #解决中文显示乱码问题
plt.rcParams['axes.unicode_minus']=False
from sklearn.datasets import make_regression,make_circles,make_s_curve
from sklearn.model_selection import train_test_split
from scipy.stats import multivariate_normal
from sklearn import decomposition
from factor_analyzer import FactorAnalyzerdata=pd.read_excel('北京市空气质量数据.xlsx')
data=data.replace(0,np.NaN)
data=data.dropna()
X=data.iloc[:,3:-1]
print(X.columns)
fa = FactorAnalyzer(method='principal',n_factors=2,rotation='varimax')
fa.fit(X)
print("因子载荷矩阵\n",fa.loadings_)
print("变量共同度:\n", fa.get_communalities())
tmp=fa.get_factor_variance()
print("因子的方差贡献:{0}".format(tmp[0]))
print("因子的方差贡献率:{0}".format(tmp[1]))
print("因子的累计方差贡献率:{0}".format(tmp[2]))运行结果如下图所示:
总结
PCA作为一种无监督学习方法,广泛应用于数据预处理、特征选择、模式识别和数据压缩等领域,是理解和分析高维数据的重要工具之一。
相关文章:

【Python机器学习实战】 | 基于PCA主成分分析技术读入空气质量监测数据进行数据预处理并计算空气质量综合评测结果
🎩 欢迎来到技术探索的奇幻世界👨💻 📜 个人主页:一伦明悦-CSDN博客 ✍🏻 作者简介: C软件开发、Python机器学习爱好者 🗣️ 互动与支持:💬评论 &…...

学习java第一百零八天
Spring的AOP理解: OOP面向对象,允许开发者定义纵向的关系,但并不适用于定义横向的关系,会导致大量代码的重复,而不利于各个模块的重用。 AOP,一般称为面向切面,作为面向对象的一种补充ÿ…...

Linux通配符总结
Linux通配符总结 大家好,我是免费搭建查券返利机器人省钱赚佣金就用微赚淘客系统3.0的小编,也是冬天不穿秋裤,天冷也要风度的程序猿! 在Linux系统中,通配符是一种用于匹配文件名或路径名的特殊字符。通过使用通配符&a…...
ffmpeg的安装教程
1.官网下载ffmpeg 进入Download FFmpeg网址,点击下载windows版ffmpeg(点击左下第一个绿色的行) 在release builds第一个绿框里面选择一个版本下载。 2.配置 下载完成后解压该压缩包单击进入ffmpeg\bin,会出现如下界面࿱…...
禅道身份认证绕过漏洞(QVD-2024-15263)复现
禅道项目管理系统在开源版、企业版、旗舰版的部分版本中都存在此安全漏洞。攻击者可利用该漏洞创建任意账号实现未授权登录。 1.漏洞级别 高危 2.漏洞搜索 fofa: title"禅道"3.影响范围 v16.x < 禅道 < v18.12 (开源版) v6.x <…...

深入分析 Android BroadcastReceiver (六)
文章目录 深入分析 Android BroadcastReceiver (六)1. 广播机制的高级优化策略1.1 使用 Sticky Broadcast(粘性广播)示例:粘性广播(过时,不推荐) 1.2 使用 LiveData 和 ViewModel 进行组件通信示例…...

mysql 查询的一般思路
能用单表优先用单表,即便是需要用group by、order by、limit等,效率一般也比多表高 不能用单表时优先用连接,连接是SQL中非常强大的用法,小表驱动大表建立合适索引合理运用连接条件,基本上连接可以解决绝大部分问题。…...
【Web APIs】DOM 文档对象模型 ⑤ ( 获取特殊元素 | 获取 html 元素 | 获取 body 元素 )
文章目录 一、获取特殊元素1、获取 html 元素2、获取 body 元素3、完整代码示例 本博客相关参考文档 : WebAPIs 参考文档 : https://developer.mozilla.org/zh-CN/docs/Web/APIgetElementById 函数参考文档 : https://developer.mozilla.org/zh-CN/docs/Web/API/Document/getE…...

Android11 以Window的视角来看FallbackHome的启动
在WMS中,使用WindowState代表着一个Window并维护着一个Window的"层级树",每个Window需要按照"层级"的规则进行排列。对于FallbackHome,其Window是挂载在home task上,而home task挂载在DefaultTaskDisplayArea…...

9 RestClient客户端操作文档
1. match_all GetMapping("matchAll")public void matchAll() throws IOException {//1. 准备requestSearchRequest request new SearchRequest("hotel");//2. 组织DSL参数request.source().query(QueryBuilders.matchAllQuery());SearchResponse respon…...

『Z-Weekly Feed 08』加密资产观 | FHE应用前景 | OPAL协议
一位机构投资者的加密资产观 作者:Hongbo 01 💡TL;DR 在加密投资领域如何找到真正的“价值”:Crypto 作为一种新兴资产,应该找到一种区别于传统公司股票资产的估值方法,本文重点阐述了加密货币作为新的资产类型与传统资…...

酒店预定系统
酒店预定系统本身设计过程中会遇到售卖系统两个常见问题,第一个同一个房间同一日期被多个订单预定,或者预定和库存数据不一致,这些都会涉及到金钱,需要在系统涉及是被重点考虑。 问题1:同一个房间同一个日期被多个订单预定 酒店…...
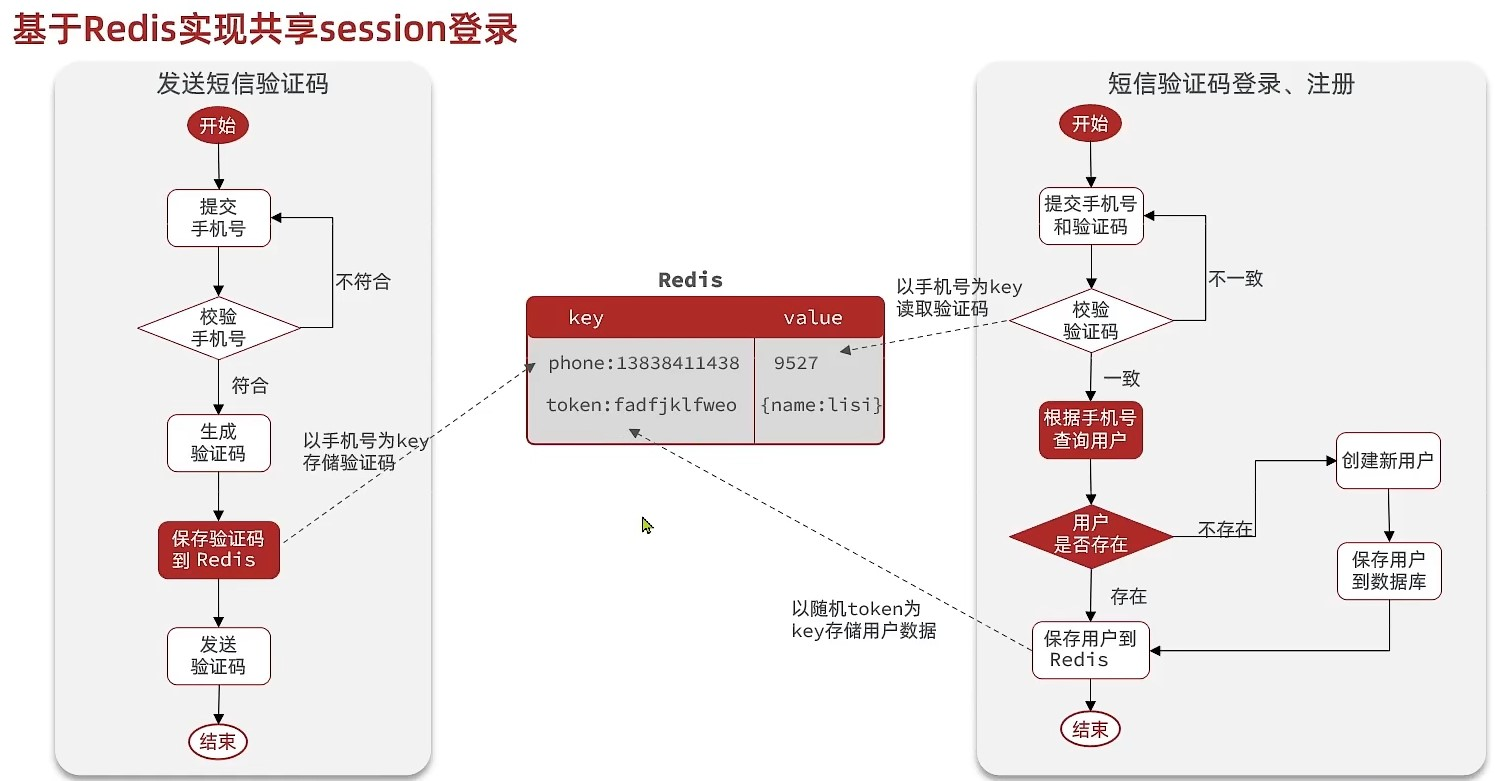
Redis的实战常用一、验证码登录(解决session共享问题)(思路、意识)
一、基于session实现登录功能 第一步:发送验证码: 用户在提交手机号后,会校验手机号是否合法: 如果不合法,则要求用户重新输入手机号如果手机号合法,后台此时生成对应的验证码,同时将验证码进行…...
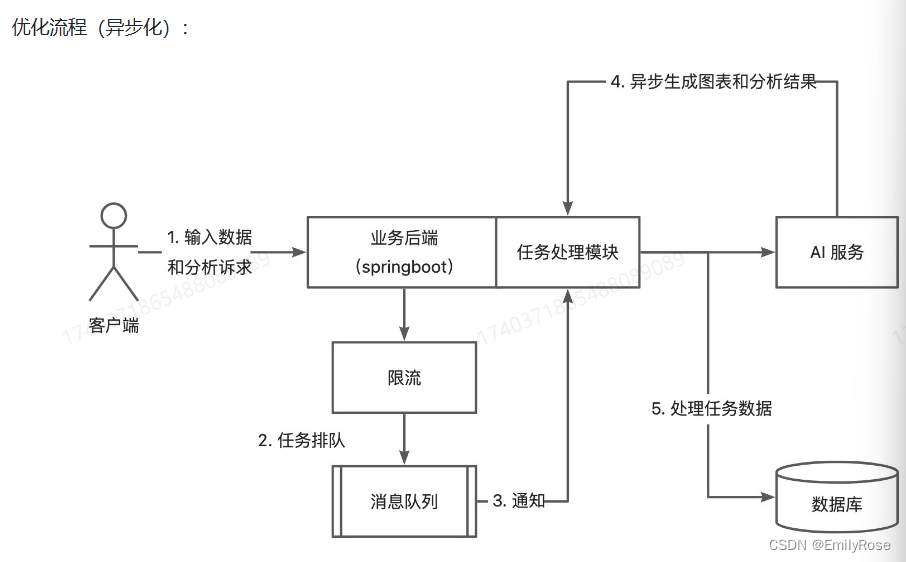
基于Spring Boot的智能分析平台
项目介绍: 智能分析平台实现了用户导入需要分析的原始数据集后,利用AI自动生成可视化图表和分析结论,改善了传统BI系统需要用户具备相关数据分析技能的问题。该项目使用到的技术是SSMSpring Boot、redis、rabbitMq、mysql等。在项目中&#…...
——显示模式)
HTML(13)——显示模式
目录 显示模式 块级元素 行内元素 行内块元素 转换显示模式 显示模式:标签的显示方式 作用:布局网页时,根据标签的显示模式选择合适的标签摆放内容 显示模式 块级元素 独占一行宽度默认为父级的100%添加宽高属性生效 行内元素 …...

【Spring】Spring Boot 快速入门
📚博客主页:爱敲代码的小杨. ✨专栏:《Java SE语法》 | 《数据结构与算法》 | 《C生万物》 |《MySQL探索之旅》 |《Web世界探险家》 ❤️感谢大家点赞👍🏻收藏⭐评论✍🏻,您的三连就是我持续更…...

Go自定义数据的序列化流程
💝💝💝欢迎莅临我的博客,很高兴能够在这里和您见面!希望您在这里可以感受到一份轻松愉快的氛围,不仅可以获得有趣的内容和知识,也可以畅所欲言、分享您的想法和见解。 推荐:「stormsha的主页」…...
)
贪心算法练习题(2024/6/18)
什么是贪心 贪心的本质是选择每一阶段的局部最优,从而达到全局最优。 贪心算法一般分为如下四步: 将问题分解为若干个子问题找出适合的贪心策略求解每一个子问题的最优解将局部最优解堆叠成全局最优解 1分发饼干 假设你是一位很棒的家长,…...

4.1 四个子空间的正交性
一、四个子空间的正交性 如果两个向量的点积为零,则两个向量正交: v ⋅ w v T w 0 \boldsymbol v\cdot\boldsymbol w\boldsymbol v^T\boldsymbol w0 v⋅wvTw0。本章着眼于正交子空间、正交基和正交矩阵。两个子空间的中的向量,一组基中的向…...
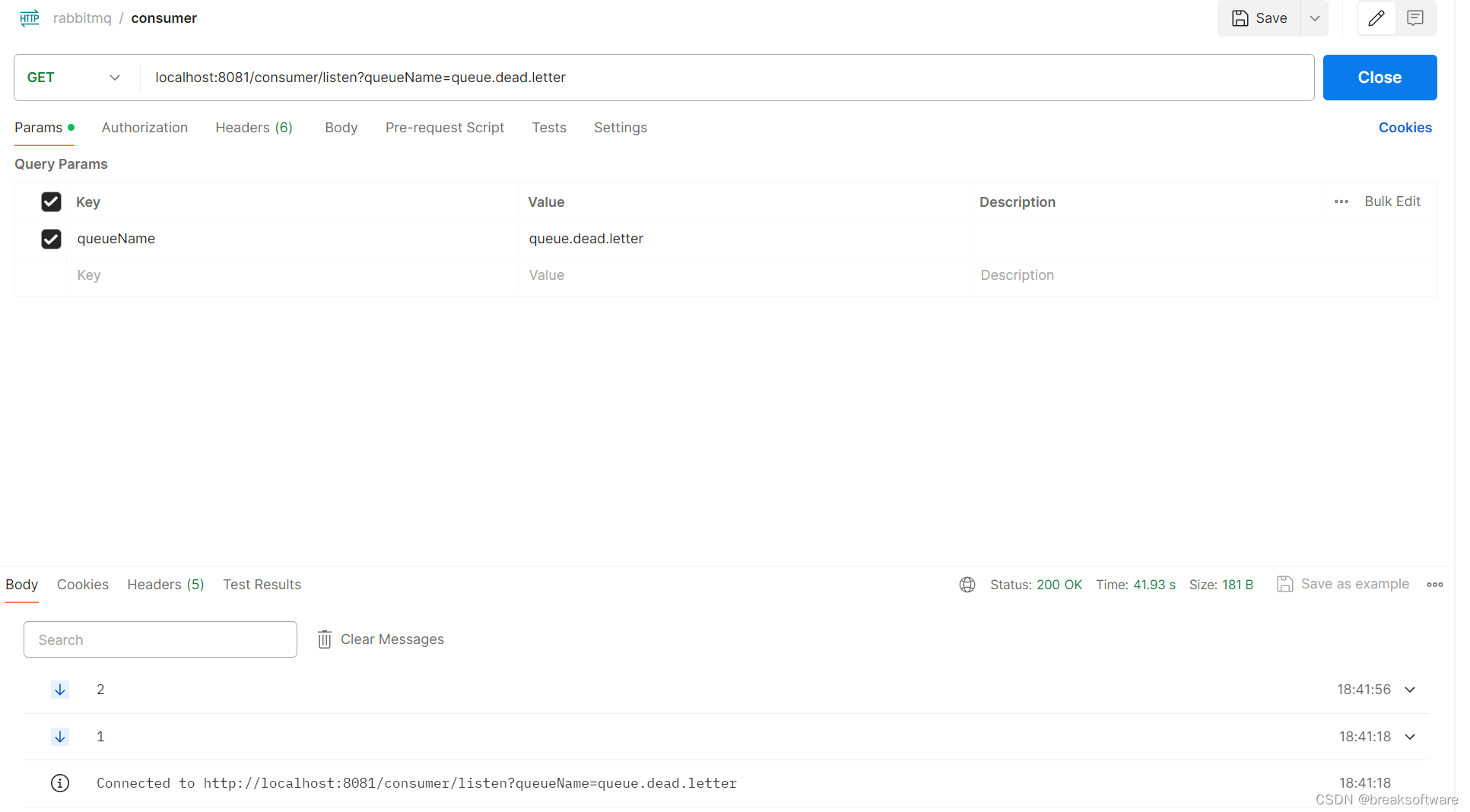
RabbitMQ实践——使用WebFlux响应式方式实时返回队列中消息
大纲 Pom.xml监听队列实时返回消息测试完整代码工程代码 在之前的案例中,我们在管理后台收发消息都是通过短连接的形式。本文我们将探索对队列中消息的实时读取,并通过流式数据返回给客户端。 webflux是反应式Web框架,客户端可以通过一个长连…...
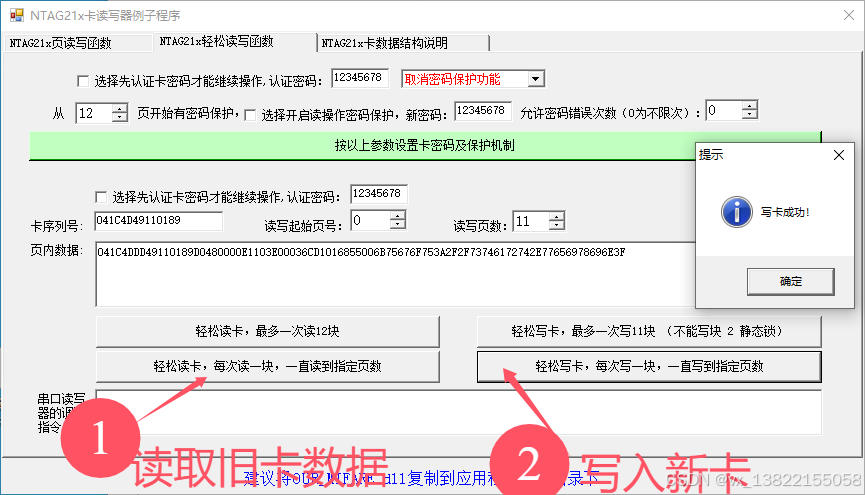
VB.net复制Ntag213卡写入UID
本示例使用的发卡器:https://item.taobao.com/item.htm?ftt&id615391857885 一、读取旧Ntag卡的UID和数据 Private Sub Button15_Click(sender As Object, e As EventArgs) Handles Button15.Click轻松读卡技术支持:网站:Dim i, j As IntegerDim cardidhex, …...

多场景 OkHttpClient 管理器 - Android 网络通信解决方案
下面是一个完整的 Android 实现,展示如何创建和管理多个 OkHttpClient 实例,分别用于长连接、普通 HTTP 请求和文件下载场景。 <?xml version"1.0" encoding"utf-8"?> <LinearLayout xmlns:android"http://schemas…...

376. Wiggle Subsequence
376. Wiggle Subsequence 代码 class Solution { public:int wiggleMaxLength(vector<int>& nums) {int n nums.size();int res 1;int prediff 0;int curdiff 0;for(int i 0;i < n-1;i){curdiff nums[i1] - nums[i];if( (prediff > 0 && curdif…...

汇编常见指令
汇编常见指令 一、数据传送指令 指令功能示例说明MOV数据传送MOV EAX, 10将立即数 10 送入 EAXMOV [EBX], EAX将 EAX 值存入 EBX 指向的内存LEA加载有效地址LEA EAX, [EBX4]将 EBX4 的地址存入 EAX(不访问内存)XCHG交换数据XCHG EAX, EBX交换 EAX 和 EB…...
均衡后的SNRSINR
本文主要摘自参考文献中的前两篇,相关文献中经常会出现MIMO检测后的SINR不过一直没有找到相关数学推到过程,其中文献[1]中给出了相关原理在此仅做记录。 1. 系统模型 复信道模型 n t n_t nt 根发送天线, n r n_r nr 根接收天线的 MIMO 系…...

力扣-35.搜索插入位置
题目描述 给定一个排序数组和一个目标值,在数组中找到目标值,并返回其索引。如果目标值不存在于数组中,返回它将会被按顺序插入的位置。 请必须使用时间复杂度为 O(log n) 的算法。 class Solution {public int searchInsert(int[] nums, …...
多光源(Multiple Lights))
C++.OpenGL (14/64)多光源(Multiple Lights)
多光源(Multiple Lights) 多光源渲染技术概览 #mermaid-svg-3L5e5gGn76TNh7Lq {font-family:"trebuchet ms",verdana,arial,sans-serif;font-size:16px;fill:#333;}#mermaid-svg-3L5e5gGn76TNh7Lq .error-icon{fill:#552222;}#mermaid-svg-3L5e5gGn76TNh7Lq .erro…...

django blank 与 null的区别
1.blank blank控制表单验证时是否允许字段为空 2.null null控制数据库层面是否为空 但是,要注意以下几点: Django的表单验证与null无关:null参数控制的是数据库层面字段是否可以为NULL,而blank参数控制的是Django表单验证时字…...
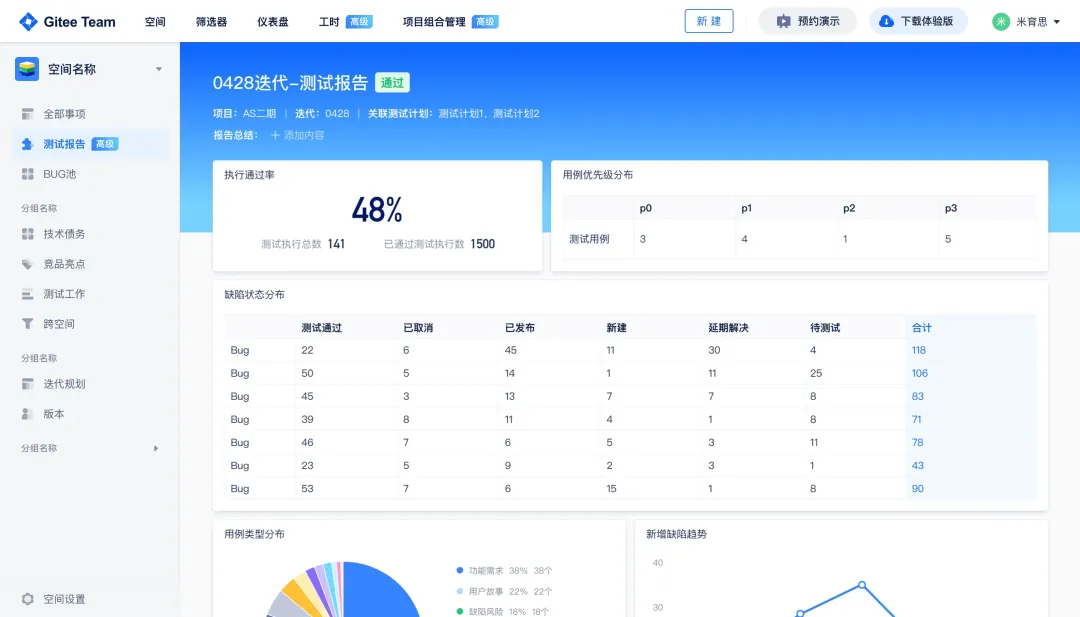
从“安全密码”到测试体系:Gitee Test 赋能关键领域软件质量保障
关键领域软件测试的"安全密码":Gitee Test如何破解行业痛点 在数字化浪潮席卷全球的今天,软件系统已成为国家关键领域的"神经中枢"。从国防军工到能源电力,从金融交易到交通管控,这些关乎国计民生的关键领域…...
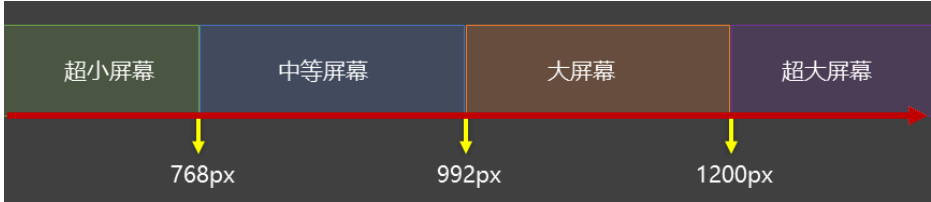
CSS3相关知识点
CSS3相关知识点 CSS3私有前缀私有前缀私有前缀存在的意义常见浏览器的私有前缀 CSS3基本语法CSS3 新增长度单位CSS3 新增颜色设置方式CSS3 新增选择器CSS3 新增盒模型相关属性box-sizing 怪异盒模型resize调整盒子大小box-shadow 盒子阴影opacity 不透明度 CSS3 新增背景属性ba…...
