202310-宏基组学物种分析工具-MetaPhlAn4安装和使用方法-Anaconda3- centos9 stream
MetaPhlAn 4是一种基于DNA序列的微生物组分析工具,它能够从宏基因组测序数据中识别和分离微生物的组成。以下是安装和使用MetaPhlAn 4的步骤:
安装MetaPhlAn 4:
裸机环境,手动安装
1. 安装依赖项:
MetaPhlAn 4需要Python 3.7以上的版本(建议使用Anaconda环境),同时还需要安装Biopython、pandas和numpy等包。可以使用pip命令进行安装,例如:
pip install biopython pandas numpy
2. 下载MetaPhlAn 4程序:
从MetaPhlAn 4的官方网站(https://github.com/biobakery/MetaPhlAn)下载最新的MetaPhlAn 4程序,并解压缩到指定目录中。
数据库地址(建议手动下载,地址及文件见下方)
要配置MetaPhlAn4最新数据库,您可以按照以下步骤进行操作:
1. 下载最新的MetaPhlAn4数据库文件,可以在MetaPhlAn4官方网站上下载。
2. 解压缩下载的MetaPhlAn4数据库文件,得到一个包含多个文件的目录。
3. 打开MetaPhlAn4配置文件,通常为"metaphlan_database.cfg",可以在MetaPhlAn4的安装目录中找到。
4. 在配置文件中,找到"bowtie2db"和"mpa_pkl"这两个参数,并将它们的值分别改为MetaPhlAn4数据库目录下的"bowtie2"和"mpa"文件的路径。
5. 保存配置文件。
使用conda环境安装(推荐)
本示例使用anaconda3,
以下是在anaconda3中安装MetaPhlAn4的步骤:
步骤1:安装conda
如果您尚未安装conda,请使用以下命令在终端中安装:
到这里去找安装包吧,什么版本都有,这里下载linux64最新版
https://repo.anaconda.com/archive
###下载安装包
wget https://repo.anaconda.com/archive/Anaconda3-2023.09-0-Linux-x86_64.sh
###安装
sh Anaconda3-2023.09-0-Linux-x86_64.sh###先进去后会要让同意许可协议,注意太快可能错过跳出,到后面慢一点向下翻
#到最后询问是否同意时输入yes
##遇到按键太快跳过的话重新执行安装命令再次进入#在配置安装目录时要注意输入自己想要安装的目录
步骤2:创建conda环境
打开终端并输入以下命令,创建一个名为“metaphlan4”的conda环境:
#创建metaphlan4的conda环境
conda create -n metaphlan4 python=3.7步骤3:激活conda环境
输入以下命令,激活“metaphlan4”环境:
#激活指定环境
conda activate metaphlan4步骤4:安装MetaPhlAn4
使用以下命令安装MetaPhlAn4:
#在激活环境中安装metaphlan
conda install -c bioconda -c conda-forge metaphlan#需要一点时间,等待完成步骤5:测试MetaPhlAn4
输入以下命令测试安装是否成功:
metaphlan --version
如果可以正确显示版本信息,则安装成功。
步骤6:配置MetaPhlAn数据库
默认情况下可以使用metaphlan --install命令可以安装数据库,但有时候下载速度慢,可能引起失败,建议手动下载最新数据库:
地址在这里:http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/
下载这几个文件:
http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/bowtie2_indexes/mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.md5
http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/bowtie2_indexes/mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/mpa_vOct22_CHOCOPhlAnSGB_202212.md5
http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/mpa_vOct22_CHOCOPhlAnSGB_202212.tar
http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/mpa_vOct22_CHOCOPhlAnSGB_202212_marker_info.txt.bz2
http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/mpa_vOct22_CHOCOPhlAnSGB_202212_species.txt.bz2
http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/mpa_latest
下载完成后将所有下载文件放入下面目录(根据自己安装目录找ananconda3的位置,后面路径都一样):
anaconda3/envs/mpa/lib/python3.7/site-packages/metaphlan/metaphlan_databases
后面第一次去运行metaphlan去注释你的序列的时候会自动建库
使用MetaPhlAn 4:
1. 准备输入文件:MetaPhlAn 4需要输入FASTQ或FASTA格式的文件。如果是PE读取,需要将两个序列文件合并为一个文件。
2. 运行MetaPhlAn 4:在终端中进入MetaPhlAn 4的安装目录,并运行以下命令:
python3 metaphlan --input_file example.fastq --output_file output.txt其中,--input_file参数指定输入文件的路径和名称,--output_file参数指定输出文件的路径和名称。
3. 查看结果:MetaPhlAn 4的输出文件包含了每个微生物的相对丰度和物种注释信息。可以使用文本编辑器或者Excel等软件打开输出文件进行查看和分析。
conda环境下使用MetaPhlAn4
##激活conda环境
source activate metaphlan4#直接使用压缩包文件运行,建议使用nohup运行,因为运行时间比较长
#nohup
nohup metaphlan f1.fastq.gz,r2.fastq.gz --bowtie2out f1r2.bowtie2.bz2 --nproc 60 --input_type fastq >f1r2_mtphlan.txt 2>&1 &
#直接运行
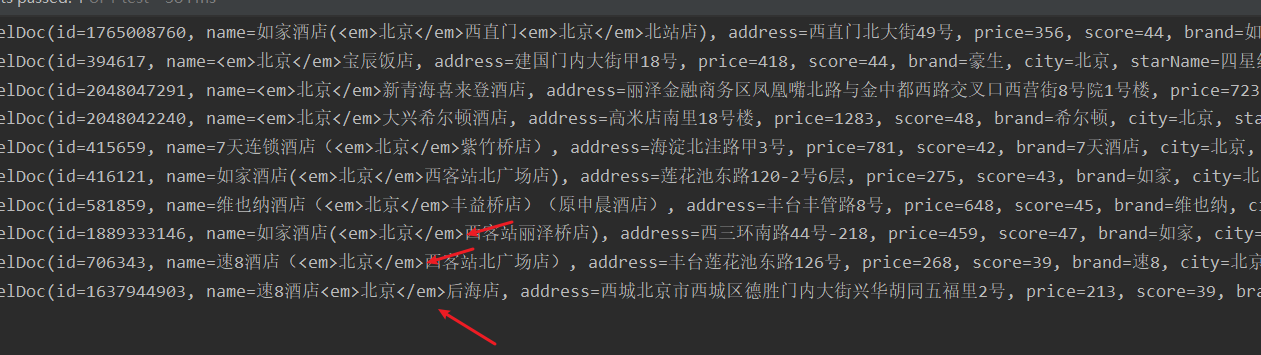
metaphlan f1.fastq.gz,r2.fastq.gz --bowtie2out f1r2.bowtie2.bz2 --nproc 60 --input_type fastq -o f1r2_mtphlan.txt####其中 f1.fastq.gz和r2.fastq.gz分别为样品的双端序列的两个压缩文件,最终我们想要的是f1r2_mtphlan.txt结果解释:
###前面几行已被注释,使用MetaPhlAn工具合并时会自动过滤掉
#anaconda3/envs/metaphlan4/bin/metaphlan 1.fastq,2.fastq --bowtie2out 1.bt2.bz2 --nproc 30 --input_type fastq -o 1.profiled.txt
#76553269 reads processed
#SampleID Metaphlan_Analysis
#clade_name NCBI_tax_id relative_abundance additional_species
k__Bacteria 2 99.92666
k__Archaea 2157 0.07334
k__Bacteria|p__Proteobacteria 2|1224 89.13284
k__Bacteria|p__Actinobacteria 2|201174 8.93442
k__Bacteria|p__Bacteroidetes 2|976 1.83546 私房菜:
###使用merge_metaphlan_tables.py将所有样品的注释结果合并(需要激活metaphlan4的conda环境)
merge_metaphlan_tables.py *.txt > merged_abundance_table.txt###使用下面语句从合并表中提取物种种水平下的物种注释信息
grep -E '(s__)|(clade_name)' merged_abundance_table.txt |grep -v 't__'|sed 's/^.*s__//g'|awk '{$2=null;print}'|sed 's/\ \ /\ /g'|sed 's/\ /\t/g' > merged_abundance_species.txt
###使用下面语句从合并表中提取物种属水平下的物种注释信息
grep -E '(g__)|(clade_name)' merged_abundance_table.txt |grep -v 's__'|sed 's/^.*g__//g'|awk '{$2=null;print}'|sed 's/\ \ /\ /g'|sed 's/\ /\t/g' > merged_abundance_genus.txt
###使用下面语句从合并表中提取物种科水平下的物种注释信息
grep -E '(f__)|(clade_name)' merged_abundance_table.txt |grep -v 'g__'|sed 's/^.*f__//g'|awk '{$2=null;print}'|sed 's/\ \ /\ /g'|sed 's/\ /\t/g' > merged_abundance_family.txt
###使用下面语句从合并表中提取物种目水平下的物种注释信息
grep -E '(o__)|(clade_name)' merged_abundance_table.txt |grep -v 'f__'|sed 's/^.*o__//g'|awk '{$2=null;print}'|sed 's/\ \ /\ /g'|sed 's/\ /\t/g' > merged_abundance_order.txt
###使用下面语句从合并表中提取物种纲水平下的物种注释信息
grep -E '(c__)|(clade_name)' merged_abundance_table.txt |grep -v 'o__'|sed 's/^.*c__//g'|awk '{$2=null;print}'|sed 's/\ \ /\ /g'|sed 's/\ /\t/g' > merged_abundance_class.txt
###使用下面语句从合并表中提取物种门水平下的物种注释信息
grep -E '(p__)|(clade_name)' merged_abundance_table.txt |grep -v 'c__'|sed 's/^.*p__//g'|awk '{$2=null;print}'|sed 's/\ \ /\ /g'|sed 's/\ /\t/g' > merged_abundance_phylum.txt注意事项:
1. MetaPhlAn 4需要消耗大量的计算资源,建议在具有足够内存和计算能力的计算机上运行。
2. MetaPhlAn 4的结果可能会受到样本质量、参考基因组库的完整性和准确性等多种因素的影响。可以根据需要选择合适的参考基因组库进行分析,以获得更准确的结果。
Metaphlan分析结果的使用建议:
1. 了解Metaphlan输出格式:Metaphlan输出文件包括两个文件,即<filename>.txt和<filename>.txt.bak。其中,<filename>.txt文件是包含分类结果和相对丰度值的文本文件,<filename>.txt.bak文件是二进制文件存储的序列信息。
2. 解释Metaphlan输出结果:Metaphlan输出文件提供了各个分类单元的相对丰度值,这些单元包括细菌、古菌、真菌和叶绿体等。通过观察这些相对丰度值,可以了解样本中各个分类单元的富集度和活跃度。
3. 与其他工具结合使用:Metaphlan结果可以与其他宏基因组分析工具结合使用,如PICRUSt、STAMP和LEfSe等,这些工具可以进一步分析样本中的生物学差异。
4. 结果可视化:Metaphlan的结果可以用不同的可视化工具展示,如Circos、R、STAMP和Phinch等。这些工具可以帮助用户更直观地理解样品之间的差异以及分类单元的富集度。
总之,Metaphlan是一个强大的宏基因组分析工具,可以帮助用户快速了解样品中的微生物组成。将其结果与其他工具结合使用,可以更深入地挖掘样品差异,为后续的实验设计提供重要参考。
相关文章:

202310-宏基组学物种分析工具-MetaPhlAn4安装和使用方法-Anaconda3- centos9 stream
MetaPhlAn 4是一种基于DNA序列的微生物组分析工具,它能够从宏基因组测序数据中识别和分离微生物的组成。以下是安装和使用MetaPhlAn 4的步骤: 安装MetaPhlAn 4: 裸机环境,手动安装 1. 安装依赖项: MetaPhlAn 4需要…...
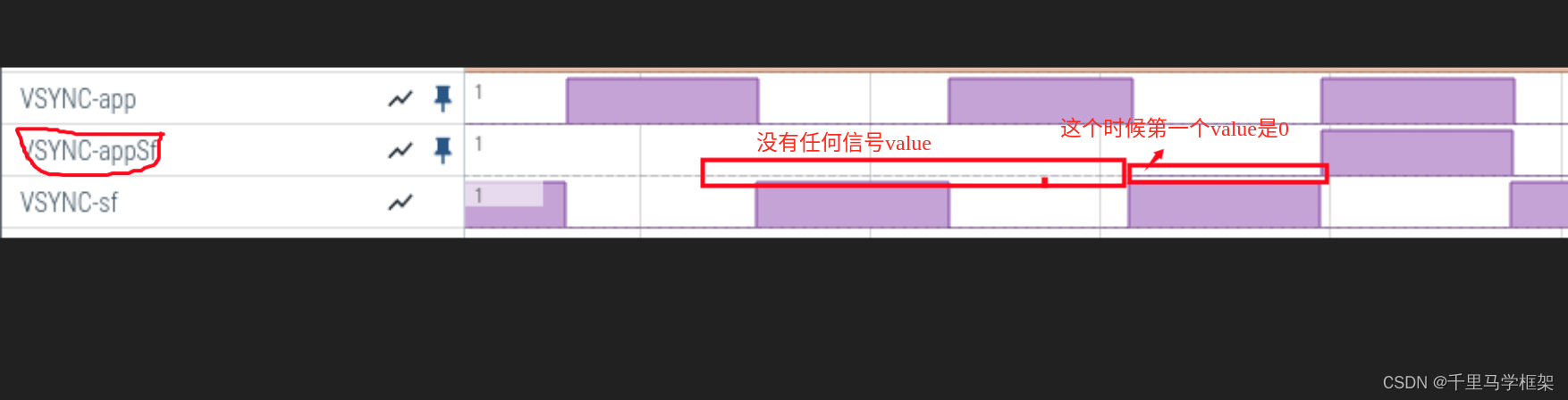
systrace/perfetto如何看surfaceflinger的vsync信号方法-android framework实战车载手机系统开发
背景: hi,粉丝朋友们: 大家好!近期分享了surfaceflinger相关的一些blog,有同学就对相关的一些内容产生了一些疑问。 比如:vsync查看问题,即怎么才可以说是vsync到来了。 比如perfetto中surfac…...
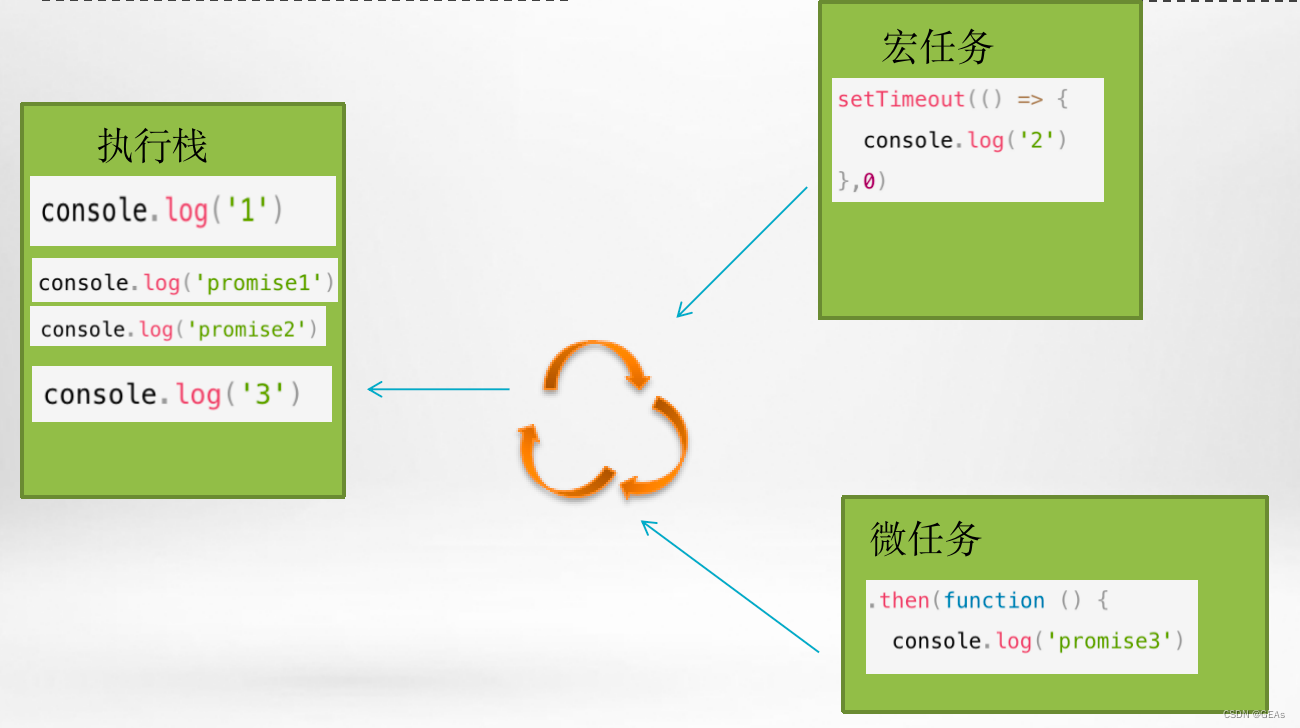
一文带你彻底弄懂js事件循环(Event Loop)
JavaScript事件循环是JavaScript运行时环境中处理异步操作的机制。它允许JavaScript在执行同步代码的同时处理异步任务,以避免阻塞线程并提供更好的用户体验。 本文将在浏览器异步执行原理基础上带你彻底弄懂js的事件循环机制。 浏览器JS异步执行原理 js是单线程…...
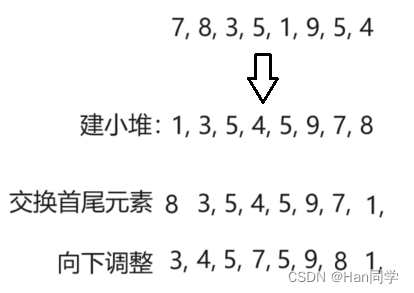
数据结构与算法:二叉树之“堆排序”
目录 一、树概念及结构 二、二叉树树概念及结构 特殊的二叉树 三、堆的概念及结构 四、堆的创建 1、声明结构体 2、初始化 3、销毁 4、添加新元素 5、交换元素 6、向上调整 7、判断堆是否为空 8、移除堆顶元素 9、向下调整 10、获取堆元素个数 五、使用堆排序…...
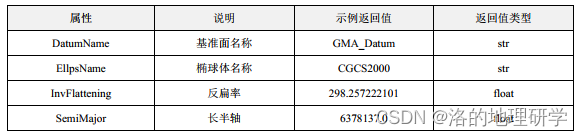
gma 2 教程(三)坐标参考系统:2.基准面/椭球体
安装 gma:pip install gma 地球是一个近似于椭球体的三维物体,而地球上的各种测量和计算都需要一个基准面来进行。基准面是一个虚拟的平面,用于测量和计算地球上的各种物理量。在地球科学中,基准面通常是一个参考椭球体࿰…...

【1day】复现广联达-Linkworks 协同办公管理平台信息泄露漏洞
注:该文章来自作者日常学习笔记,请勿利用文章内的相关技术从事非法测试,如因此产生的一切不良后果与作者无关。 目录 一、漏洞描述 二、影响版本 三、资产测绘 四、漏洞复现...
Spring Cloud之ElasticSearch的学习【详细】
目录 ElasticSearch 正向索引与倒排索引 数据库与elasticsearch概念对比 安装ES、Kibana与分词器 分词器作用 自定义字典 拓展词库 禁用词库 索引库操作 Mapping属性 创建索引库 查询索引库 删除索引库 修改索引库 文档操作 新增文档 查找文档 修改文档 全量…...
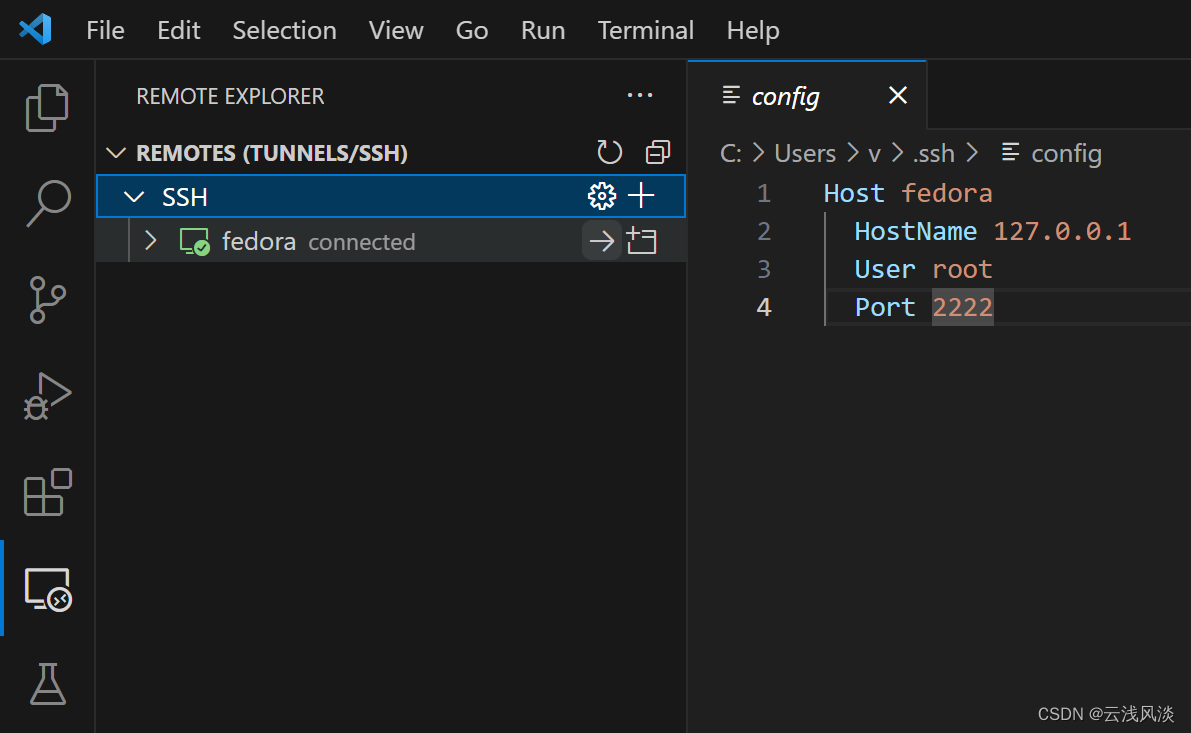
vscode免密码认证ssh连接virtual box虚拟机
文章目录 安装软件virtual box配置vscode配置创建并传递密钥连接虚拟机最后 安装软件 安装vscode和virtual box,直接官网下载对应软件包,下载之后,点击执行,最后傻瓜式下一步安装即可 virtual box配置 创建一个仅主机网络的网卡 …...

【Linux】Centos yum源替换
YUM是基于RPM包管理,能够从指定的服务器自动下载RPM包并且安装,可以自动处理依赖性关系,并且一次安装所有依赖的软件包,无须繁琐地一次次下载、安装。 CentOS 8操作系统版本结束了生命周期(EOL)࿰…...
)
uniapp组件初始化的销毁(监听隐藏事件)
onHide是监听隐藏事件onHide() {console.log("销毁");this.clearTimer(); }, onShow(){console.log("初始化");this.getOrderInfo() },...
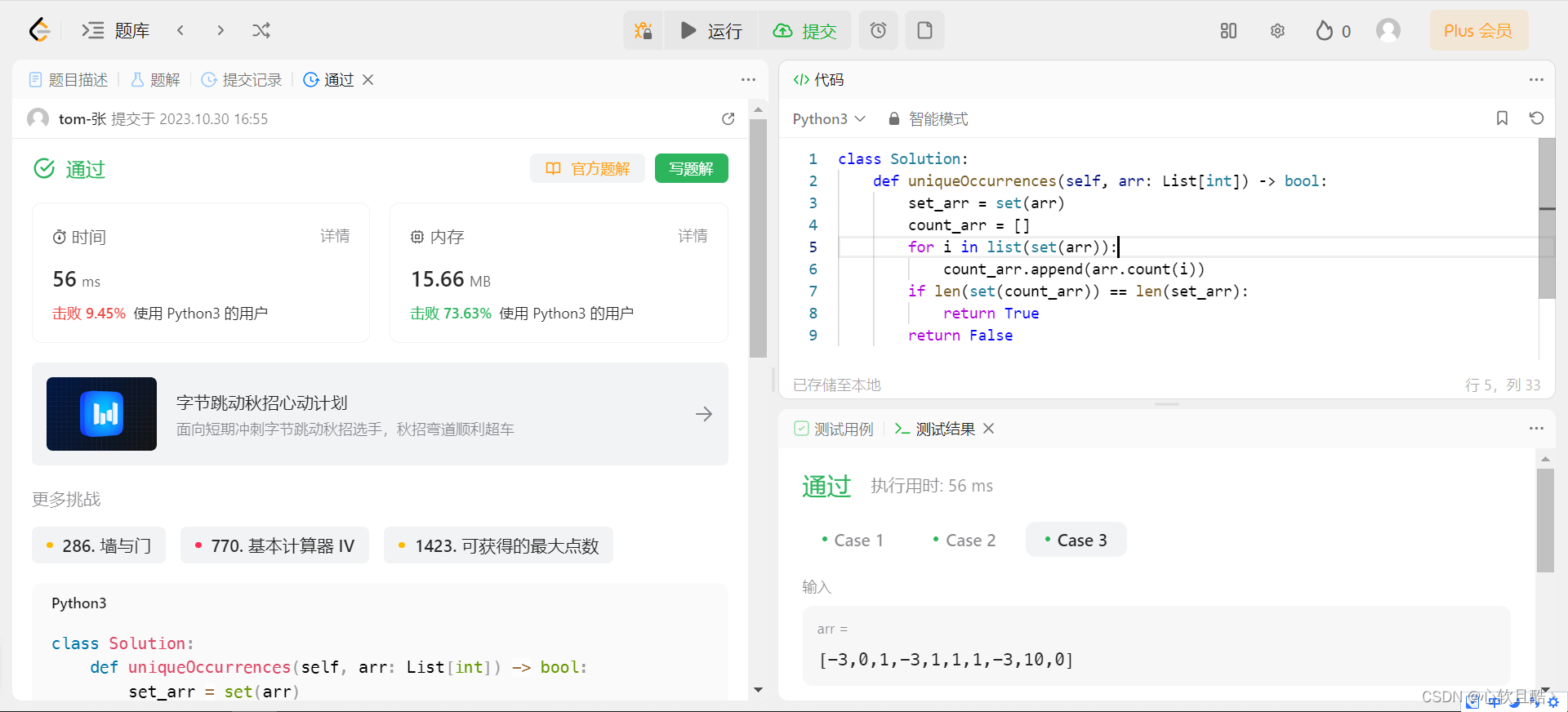
leetcode:1207. 独一无二的出现次数(python3解法)
难度:简单 给你一个整数数组 arr,请你帮忙统计数组中每个数的出现次数。 如果每个数的出现次数都是独一无二的,就返回 true;否则返回 false。 示例 1: 输入:arr [1,2,2,1,1,3] 输出:true 解释&…...

2023秋《论文写作》课程总结
2023秋《论文写作》课程总结 授课教师为闵帆教授,原文链接《论文写作》 文章目录 2023秋《论文写作》课程总结一、关于写作工具二、关于写作中的单词、短语、语法等三、关于论文题目四、关于摘要和关键词五、关于引言部分六、关于方法及实验部分七、关于结论八、关…...
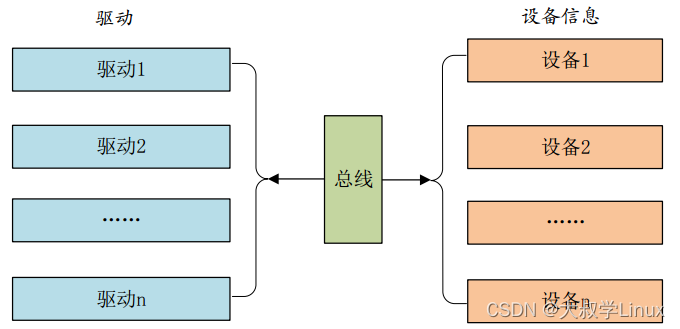
Linux学习第27天:Platform设备驱动开发: 专注与分散
Linux版本号4.1.15 芯片I.MX6ULL 大叔学Linux 品人间百味 思文短情长 专注与分散是我在题目中着重说明的一个内容。这是今天我们要学习分离与分层概念的延伸。专注是说我们要专注某层驱动的开发,而对于其他层则是芯片厂商…...

最长公共子序列
题目描述 给定两个字符串 text1 和 text2,返回这两个字符串的最长 公共子序列 的长度。如果不存在 公共子序列 ,返回 0 。 一个字符串的 子序列 是指这样一个新的字符串:它是由原字符串在不改变字符的相对顺序的情况下删除某些字符…...
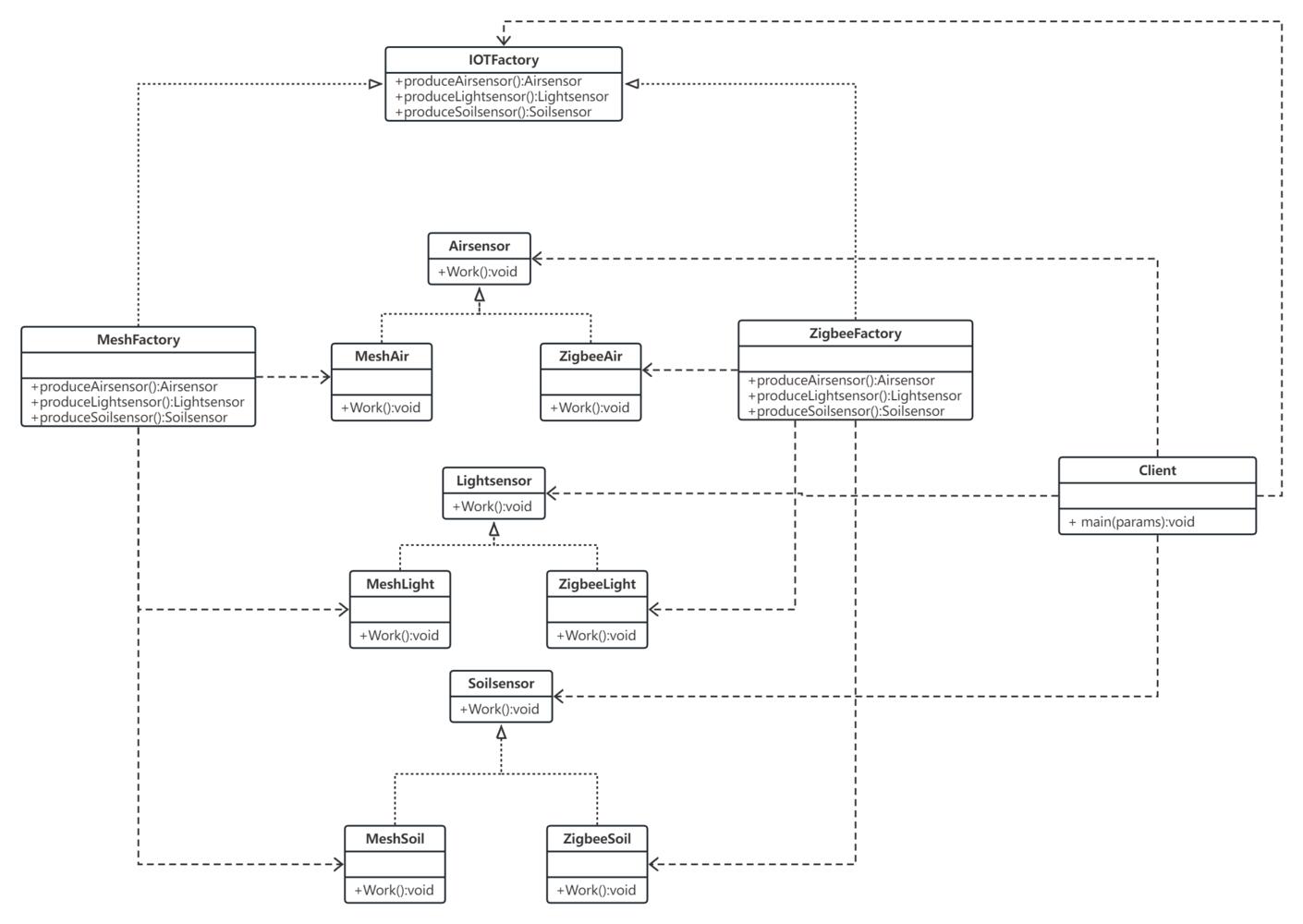
万字解析设计模式之工厂方法模式与简单工厂模式
一、概述 1.1简介 在java中,万物皆对象,这些对象都需要创建,如果创建的时候直接new该对象,就会对该对象耦合严重,假如我们要更换对象,所有new对象的地方都需要修改一遍,这显然违背了软件设计的…...

One-to-N N-to-One: Two Advanced Backdoor Attacks Against Deep Learning Models
One-to-N & N-to-One: Two Advanced Backdoor Attacks Against Deep Learning Models----《一对N和N对一:针对深度学习模型的两种高级后门攻击》 1对N: 通过控制同一后门的不同强度触发多个后门 N对1: 只有当所有N个后门都满足时才会触发…...
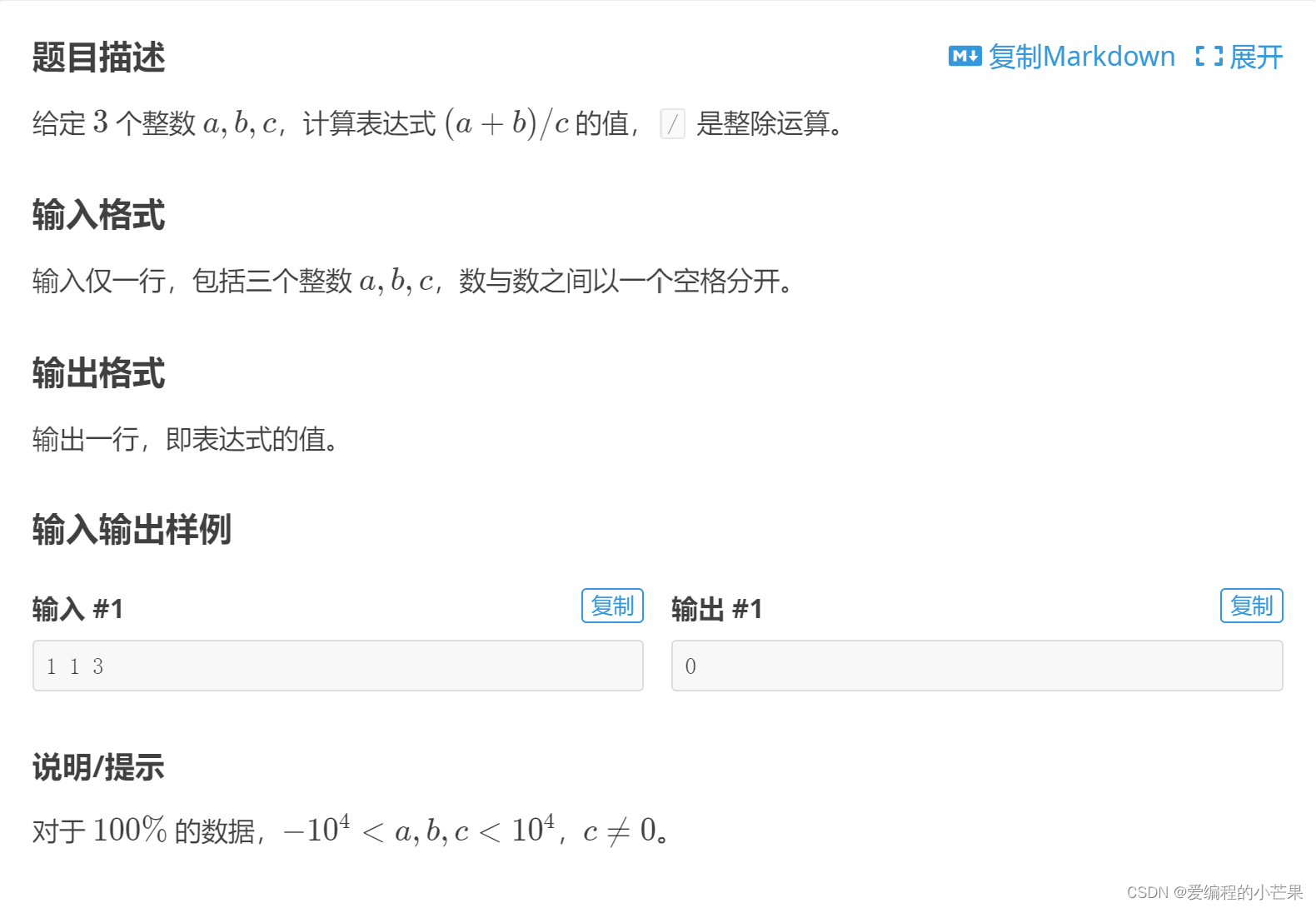
洛谷 B2009 计算 (a+b)/c 的值 C++代码
目录 题目描述 AC Code 切记 题目描述 题目网址:计算 (ab)/c 的值 - 洛谷 AC Code #include<bits/stdc.h> using namespace std; int main() {int a,b,c;cin>>a>>b>>c;cout<<(ab)/c<<endl;return 0; } 切记 不要复制题…...
)
Arduino驱动ME007-ULA防水测距模组(超声波传感器)
目录 1、传感器特性 2、控制器和传感器连线图 3、驱动程序 3.1、读取串口数据...

Linux 权限管理(二)
文件类型和访问权限(事物属性) linux前都会有一串这个字符,第二字符到第九字符分别表示拥有者,所属组,和other所对应的权限。那么第一个字符表示什么呢? 第一个字符表示文件类型: d:…...
线性代数 第一章 行列式
一、概念 不同行不同列元素乘积的代数和(共n!项) 二、性质 经转置行列式的值不变,即; 某行有公因数k,可把k提到行列式外。特别地,某行元素全为0,则行列式的值为0; 两行互换行列式…...
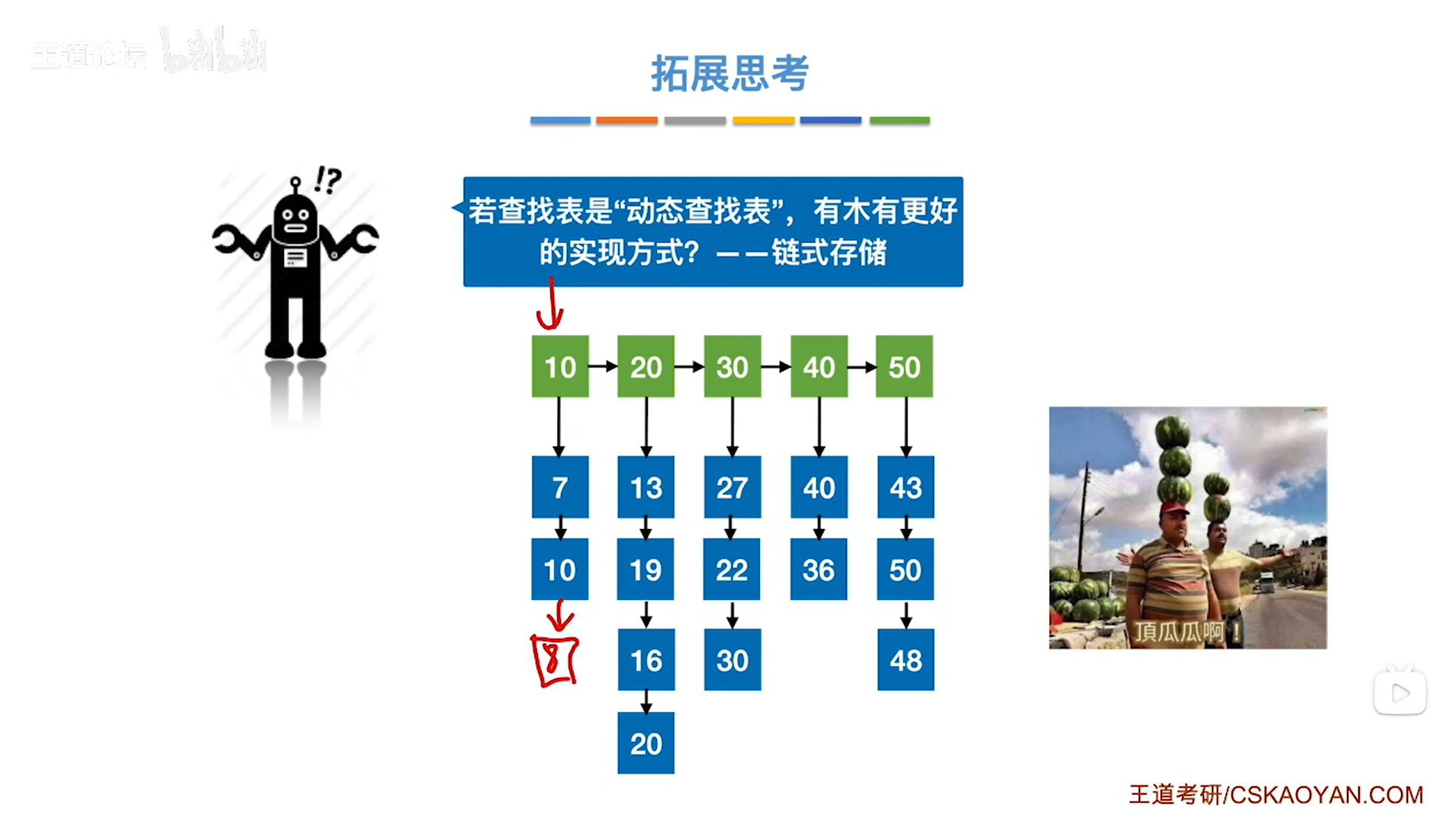
7.4.分块查找
一.分块查找的算法思想: 1.实例: 以上述图片的顺序表为例, 该顺序表的数据元素从整体来看是乱序的,但如果把这些数据元素分成一块一块的小区间, 第一个区间[0,1]索引上的数据元素都是小于等于10的, 第二…...

Admin.Net中的消息通信SignalR解释
定义集线器接口 IOnlineUserHub public interface IOnlineUserHub {/// 在线用户列表Task OnlineUserList(OnlineUserList context);/// 强制下线Task ForceOffline(object context);/// 发布站内消息Task PublicNotice(SysNotice context);/// 接收消息Task ReceiveMessage(…...
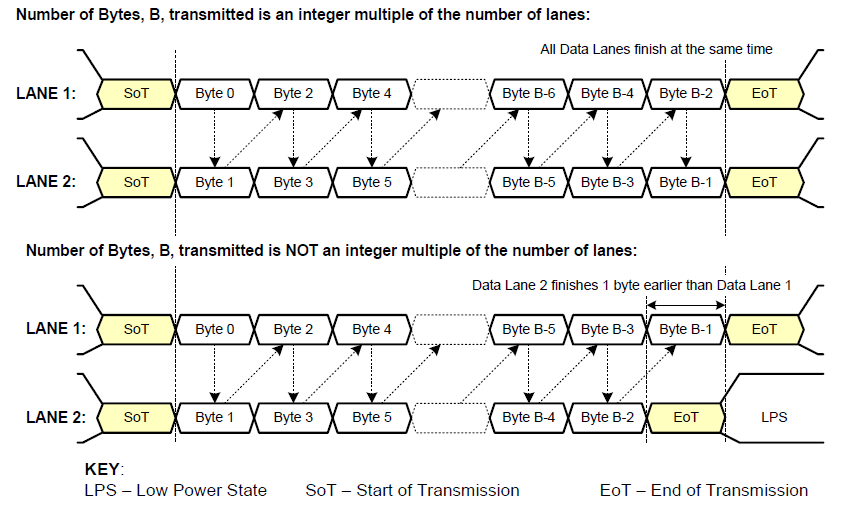
《从零掌握MIPI CSI-2: 协议精解与FPGA摄像头开发实战》-- CSI-2 协议详细解析 (一)
CSI-2 协议详细解析 (一) 1. CSI-2层定义(CSI-2 Layer Definitions) 分层结构 :CSI-2协议分为6层: 物理层(PHY Layer) : 定义电气特性、时钟机制和传输介质(导线&#…...

django filter 统计数量 按属性去重
在Django中,如果你想要根据某个属性对查询集进行去重并统计数量,你可以使用values()方法配合annotate()方法来实现。这里有两种常见的方法来完成这个需求: 方法1:使用annotate()和Count 假设你有一个模型Item,并且你想…...

Python如何给视频添加音频和字幕
在Python中,给视频添加音频和字幕可以使用电影文件处理库MoviePy和字幕处理库Subtitles。下面将详细介绍如何使用这些库来实现视频的音频和字幕添加,包括必要的代码示例和详细解释。 环境准备 在开始之前,需要安装以下Python库:…...
sipsak:SIP瑞士军刀!全参数详细教程!Kali Linux教程!
简介 sipsak 是一个面向会话初始协议 (SIP) 应用程序开发人员和管理员的小型命令行工具。它可以用于对 SIP 应用程序和设备进行一些简单的测试。 sipsak 是一款 SIP 压力和诊断实用程序。它通过 sip-uri 向服务器发送 SIP 请求,并检查收到的响应。它以以下模式之一…...

离线语音识别方案分析
随着人工智能技术的不断发展,语音识别技术也得到了广泛的应用,从智能家居到车载系统,语音识别正在改变我们与设备的交互方式。尤其是离线语音识别,由于其在没有网络连接的情况下仍然能提供稳定、准确的语音处理能力,广…...
MySQL的pymysql操作
本章是MySQL的最后一章,MySQL到此完结,下一站Hadoop!!! 这章很简单,完整代码在最后,详细讲解之前python课程里面也有,感兴趣的可以往前找一下 一、查询操作 我们需要打开pycharm …...

flow_controllers
关键点: 流控制器类型: 同步(Sync):发布操作会阻塞,直到数据被确认发送。异步(Async):发布操作非阻塞,数据发送由后台线程处理。纯同步(PureSync…...
聚六亚甲基单胍盐酸盐市场深度解析:现状、挑战与机遇
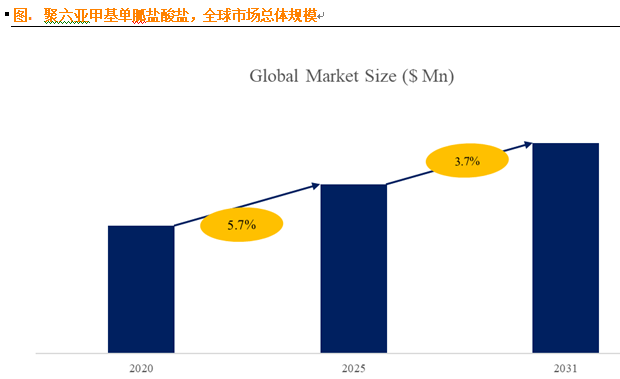
根据 QYResearch 发布的市场报告显示,全球市场规模预计在 2031 年达到 9848 万美元,2025 - 2031 年期间年复合增长率(CAGR)为 3.7%。在竞争格局上,市场集中度较高,2024 年全球前十强厂商占据约 74.0% 的市场…...
