梯度引导的分子生成扩散模型- GaUDI 评测
GaUDI模型来自于以色列理工Tomer Weiss的2023年发表在预印本ChemRxiv上的工作 《Guided Diffusion for Inverse Molecular Design》。原文链接:Guided Diffusion for Inverse Molecular Design | Materials Chemistry | ChemRxiv | Cambridge Open Engage
GaUDI模型有点像强化学习,目的都是生成特定性质的分子,且效果均非常显著,而且生成过程均存在迭代更新过程。
写在前面
GaUDI模型有点像强化学习,目的都是生成特定性质的分子,且效果均非常显著,而且生成过程均存在迭代更新过程。
但是,二者原理不同。
从迭代过程来说,强化学习生成特定属性的分子迭代发生在生成的批次分子之间,而梯度引导扩散模型是发生在生成一个分子的过程中,因此,从速度上来说,GaUDI模型非常快,很有优势。
从目标函数的形式来说,强化学习是使用目标函数计算生成的分子,获得reward。reward是 一个数值,而是通过数值,调节分子生成器的梯度强度,让分子生成器降低目标分子的损失,于是分子生成器生成更多特定属性的分子。GaUDI模型则不同,目标函数产生的梯度,并未返回到分子生成器中,而是直接用于引导Zt-1,使Zt-1亲向特定属性(方向)。因此,对于任何的分子性质要求,GaUDI模型的分子生成器,即扩散模型,都可以通用,无需重新训练。
从联合概率分布来说,GaUDI模型是联合概率分布,而强化学习不是。GaUDI模型生成的分子更加”平滑“,更具有实用性。强化学习更容易钻模型的漏洞,生成一些奇怪的分子。
一、背景介绍
GaUDI是用于逆向分子设计的一个引导扩散模型,因此文章的题目是《Guided Diffusion for Inverse Molecular Design》。GaUDI对扩散模型中的分子生成器进行了改造,将分子生成过程使用性质预测的函数进行梯度引导,更新Zt-1,使Zt-1更靠近我们期待的性质(例如:logP等)。我们期待的性质可以使单目标也可以是多目标,一切取决于我们的梯度函数f(x,t)。

引导分子生成的梯度来自于目标函数f(x,t), 函数f(x,t)用于根据输入的去噪过程中的Zt-1,预测Zt-1(即:分子)对应的性质。利用预测性质与目标性质之间的损失梯度,更新Zt-1,那么在去噪生成过程中,生成的Zt-1就会越来越靠近我们的期望的性质。当然目标函数f(x,t)需要另外训练。梯度对Zt-1的更新程度可使用调节系数(gradient scalar values, s)来控制。整个去噪采样逻辑如下图:

伪代码如下:

二、文章案例
2.1 多目标优化(全局最优)
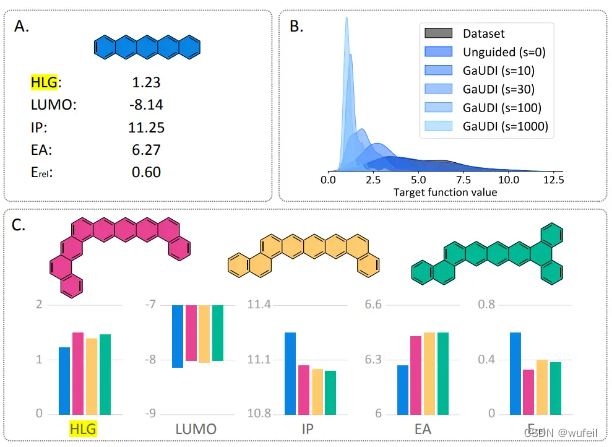
作者以 图 A 的 cc-PBHs 为例,目标是增加改分子的稳定性(更低的Erel)。这里作者是进行全局最优的例子,目标函数是生成分子与cc-PBHs 的LUMO, HLG, IP, EA 的MSE加上Erel。作者尝试了不同的调节系数(gradient scalar values, s)结果如图B所示。图C展示了一些具体的例子。
2.2 Out-of-distribution生成
验证GaUDI模型是否可以生成更高的HLG值的分子,限定条件是在相同的环数量下。结果如下图:

图A是数据集中不同环数下的最高HLG分子,图B是生成的分子,图C是不同调节系数下的结果。从结果上来看,生成分子的效果非常显著。
2.3 多目标优化
将目标设置为:ℓ(HLG,IP, EA) = 3 · HLG + IP − EA, 结果如下下图:
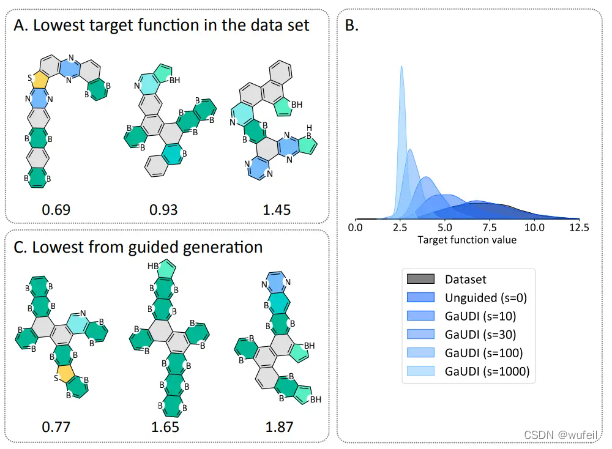
整体结果还是非常惊喜的。因为没有任何的强化学习,但是实现了类似的结果,效果非常明显。非常好的弥补了强化学习只能身材个核那个smiles的缺点。
原文代码开源:porannegroup / GaUDI · GitLab
三、模型应用测试
3.1 环境安装
首先,复制代码:
git clone https://github.com/tomer196/GaUDI.git代码目录:
.
├── analyze
├── cond_prediction
├── data
├── edm
├── environment.yml
├── eval_validity.py
├── GaUDI.png
├── generation_guidance.py
├── __init__.py
├── LICENSE
├── models_edm.py
├── __pycache__
├── README.md
├── sampling_edm.py
├── train_edm.py
└── utils安装环境目录,并激活环境:
conda env create -n GaUDI --file environment.yml
conda activate GaUDI如果不行,就分开安装rdkit, pytorch等,注意版本:
conda install pytorch==2.1.0 torchvision==0.16.0 torchaudio==2.1.0 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install -c conda-forge rdkit
pip install matplotlib networkx tensorboard pandas scipy tqdm imageio安装过程,比较顺利,没有特殊问题。但是centos和win系统比较麻烦,估计要摸索一下。
另外,需要安装几个附属模块:
pip install tensorboard
pip install imgaug3.2 数据准备&下载数据
作者使用到两个数据集:COMPAS 和 PASS,下载链接:
COMPAS:porannegroup / COMPAS · GitLab
PASS:PASS molecular dataset
需要从上述网址中,下载三个文件,分别是:PASs_csv.tar.gz, PASs_xyz.tar.gz, compas-main-COMPAS-1.zip
在data目录下,创建一个all_data文件夹,将下载的PASs_csv.tar.gz, PASs_xyz.tar.gz, compas-main-COMPAS-1.zip文件上传。然后,分别解压:
unzip compas-main-COMPAS-1.zip
tar -zxvf PASs_csv.tar.gz
tar -zxvf PASs_xyz.tar.gz#删除压缩文件
rm compas-main-COMPAS-1.zip PASs_csv.tar.gz PASs_xyz.tar.gz然后去更改./data/aromatic_dataloaders.py中get_path函数,关于数据的路径。
def get_paths(args):if not hasattr(args, "dataset"):csv_path = args.csv_filexyz_path = args.xyz_rootelif args.dataset == "cata":csv_path = "/home/tomerweiss/PBHs-design/data/COMPAS-1x.csv"xyz_path = "/home/tomerweiss/PBHs-design/data/peri-cata-89893-xyz"elif args.dataset == "peri":csv_path = "/home/tomerweiss/PBHs-design/data/peri-xtb-data-55821.csv"xyz_path = "/home/tomerweiss/PBHs-design/data/peri-cata-89893-xyz"elif args.dataset == "hetro":csv_path = "/home/tomerweiss/PBHs-design/data/db-474K-OPV-filtered.csv"xyz_path = "/home/tomerweiss/PBHs-design/data/db-474K-xyz"elif args.dataset == "hetro-dft":csv_path = "/home/tomerweiss/PBHs-design/data/db-15067-dft.csv"xyz_path = ""else:raise NotImplementedErrorreturn csv_path, xyz_path改为:
def get_paths(args):if not hasattr(args, "dataset"):csv_path = args.csv_filexyz_path = args.xyz_rootelif args.dataset == "cata":csv_path = "./data/all_data/compas-main-COMPAS-1/COMPAS-1/COMPAS-1x.csv"xyz_path = "/data/all_data/compas-main-COMPAS-1/COMPAS-1/pahs-cata-34072-xyz"elif args.dataset == "hetro":csv_path = "./data/all_data/db-474K-OPV-filtered.csv"xyz_path = "./data/all_data/db-474K-xyz"else:raise NotImplementedErrorreturn csv_path, xyz_path3.3 训练EDM 模型
由于作者没有提供预训练好的checkpoint, 所以需要我们自己训练。回到主目录,然后:
python train_edm.py训练过程了会被记录,将保存在./summary路径下,默认是:cata-test文件夹。整个训练过程,3090显卡需要5h左右。
训练完成后,./summary/cata-test内得到如下目录:
.
├── args.txt
├── epoch_0
├── epoch_100
├── epoch_150
├── epoch_200
├── epoch_250
├── epoch_300
├── epoch_350
├── epoch_400
├── epoch_450
├── epoch_50
├── epoch_500
├── epoch_550
├── epoch_600
├── epoch_650
├── epoch_700
├── epoch_750
├── epoch_800
├── epoch_850
├── epoch_900
├── epoch_950
├── events.out.tfevents.1700200741.a01.3529424.0
├── events.out.tfevents.1700200774.a01.3532044.0
└── model.pt #模型文件3.4 训练条件模型
同样的,作者没有给checkpoint,条件模型也需要训练。在主目录下,执行:
python cond_prediction/train_cond_predictor.py训练过程将保存在prediction_summary目录中的cata-test文件夹中。训练过程大约也需要4h。训练完成后, prediction_summary的目录如下:
.
└── cata-test├── args.txt├── events.out.tfevents.1700211860.a01.63329.0└── model.pt3.5 条件引导扩散的分子生成
3.5.1 使用generation_guidance.py
在完成EDM和条件模型的训练以后,现在可以进行条件引导分子扩散。
首先,需要修改generation_guidance.py文件中的EDM和条件模型路径。将如下内容:
args = get_edm_args("/home/tomerweiss/PBHs-design/summary/hetro_l9_c196_orientation2")
cond_predictor_args = get_cond_predictor_args(f"/home/tomerweiss/PBHs-design/prediction_summary/"f"cond_predictor/hetro_gap_homo_ea_ip_stability_polynomial_2_with_norm")根据我们前面训练获得的模型,更改为:
args = get_edm_args("./gaudi/summary/cata-test")
cond_predictor_args = get_cond_predictor_args(f"./gaudi/prediction_summary/cata-test")在189~193行可以修改,需要生成的分子数量和梯度引导强度。
##################### 设置引导梯度的强度,和想要的分子数量 ###################################
# Set controllable parameters
args.batch_size = 512 # number of molecules to generate
scale = 0.6 # gradient scale - the guidance strength
n_nodes = 10 # number of rings in the generated molecules
#########################################################################################然后执行:
python generation_guidance.py就可以生成分子。
但是,存在的问题是:
plot_graph_of_rings
plot_rdkit这两个函数一直无法通过。同时发现,改模型输出的结果不是xyz文件,也不是sdf文件,似乎是图片。这是因为,作者在定义图的时候与我们平常的方法不同,平常的,我们会将一个原子作为一个节点,但是这里作者是将一个环作为一个节点。因此输出的结果比较奇怪。需要其他的转化函数(plot_rdkit),才能输出我们能查看的RDKIT对象。但是我们在运行generation_guidance.py文件时,plot_rdkit文件报错了。所以无法继续运行。
3.5.2 generation_guidance.py文件修正
在研究了其代码和输出结果以后,我们发现代码里面存在变量错误和文件名命名错误。
将以下代码进行修改:
plot_graph_of_rings(x_stable[idx].detach().cpu(),atom_type_stable[idx].detach().cpu(),filename=f"{dir_name}/{i}.pdf",title=f"{best_str}\n {value}",dataset=args.dataset,)plot_rdkit(x_stable[idx].detach().cpu(),atom_type_stable[idx].detach().cpu(),filename=f"{dir_name}/mol_{i}.pdf",title=f"{best_str}\n {value}",dataset=args.dataset,)修改为:
try:plot_graph_of_rings(x_stable[idx].detach().cpu(),atom_type[idx].detach().cpu(),filename=f"{dir_name}/{i}.png",title=f"{best_str}\n {value}",dataset=args.dataset,)plot_rdkit(x_stable[idx].detach().cpu(),atom_type[idx].detach().cpu(),filename=f"{dir_name}/mol_{i}.png",title=f"{best_str}\n {value}",dataset=args.dataset,)except:pass使用try语句的原因是,及时通过了有效检测,但是仍然有分子是画不出来的。
生成分子:
python generation_guidance.py运行结束后,将产生./best文件夹目录,有一个以运行日期为名的文件夹,例如:1120_00:42:46_0.6,其中保存对于 Top 5 最优分子的结果,包括节点图以及分子结构。
生成目录示例:
./best
└── 1120_00:42:46_0.6├── 0.png├── 1.png├── 2.png├── 3.png├── 4.png├── all.png└── mol_0.png生成节点图:
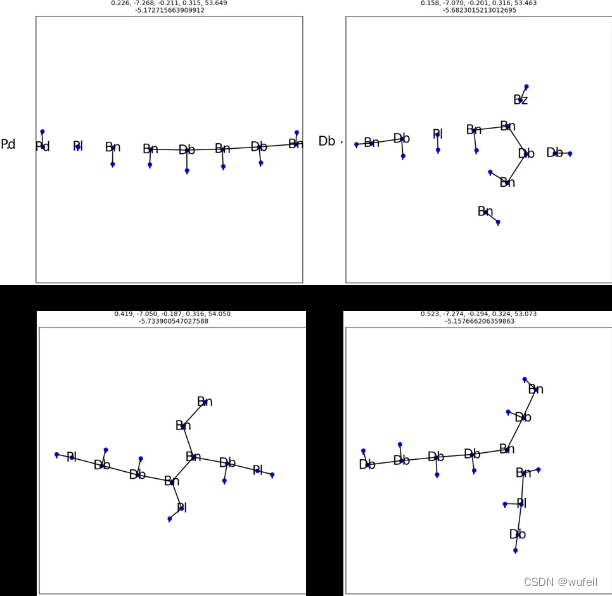
目标函数分子的gap最大,最优分子结构:
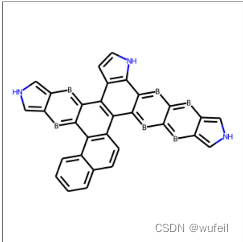
由于生成分子结构代码的问题,最终只生成了一个分子的结构图,如下图所示:
scale=0.8
stability_dict['mol_valid']=82.00% out of 50
Mean target function value: -4.7209
best value: -5.982420921325684, pred: tensor([ 0.2653, -6.9535, -0.1940, 0.3079, 53.0093])
Mean target function value (from valid): -4.7066根据输出的结果,最优的分子的目标函数值,gap值为:5.98,但是由于画图的原因,导致无法输出分子结构。能输出分子结构的目标函数值是5.488。
因为在没有引导的时候,即scale参数为0时(在generation_guidance.py文件内调整scale参数的值),相当于直接使用EDM进行分子生成,运行代码输出:
scale=0
stability_dict['mol_valid']=100.00% out of 50
Mean target function value: 0.0738
best value: -1.7954111099243164, pred: tensor([ 1.1055, -8.5804, -0.2251, 0.3761, 55.9974])
Mean target function value (from valid): 0.0738对应的gap的最大值只有1.79,远小于scale为0.8的结果,说明作者提出的梯度引导很符合target函数的要求,效果还不错。但是牺牲的代价是分子的有效率降低了。
3.6 scale梯度调节参数
刚才我们提到scale参数,scale参数是调节引导强度的超参数,当这个值越大时,则生成的分子越满足,越靠近我们的目标函数需求,但是分子的有效率会大大降低,例如,我们将scale设置为2时:只有6%的分子有效。
scale=2
stability_dict['mol_valid']=6.00% out of 50
Mean target function value: -5.2808
best value: -6.993871212005615, pred: tensor([ 0.2147, -6.5605, -0.1836, 0.2928, 52.0480])
Mean target function value (from valid): -5.1165
best value (from stable): tensor([ 0.2562, -7.1488, -0.2008, 0.3158, 52.8709]), score: -5.479823112487793
best value (from stable): tensor([ 0.3591, -7.2578, -0.1995, 0.3182, 53.5928]), score: -5.1993408203125
best value (from stable): tensor([ 0.1914, -7.4633, -0.2178, 0.3219, 54.1037]), score: -4.670310020446777我们将scale设置为4时,报错了,生成了一些奇怪的分子无法计算其gap等分子属性结果,分子的有效率直接降为0:
scale=4
stability_dict['mol_valid']=0.00% out of 50
Mean target function value: -2.2682
best value: -6.642547130584717, pred: tensor([ 0.3690, -6.6970, -0.1769, 0.3109, 51.9043])
Mean target function value (from valid): nan
~/anaconda3/envs/GaUDI/lib/python3.8/site-packages/numpy/lib/histograms.py:883: RuntimeWarning: invalid value encountered in dividereturn n/db/n.sum(), bin_edges3.7 条件引导扩散的分子生成(jupyter notebook版本)
作者也提供了一个jupyter notebook的版本,详见文件中的Case_Test.ipynb文件。
导入相关模块:
import shutil
import os
import matplotlib.pyplot as plt
import torch
import numpy as np
import gdown
from time import time
from types import SimpleNamespacefrom cond_prediction.train_cond_predictor import get_cond_predictor_model
from data.aromatic_dataloader import create_data_loaders
from models_edm import get_model
from utils.helpers import switch_grad_off, get_edm_args, get_cond_predictor_args
加载预训练模型:
edm_model - 扩散模型,一种在每次迭代时稍微清洁分子的降噪器。
cond_predictor - 条件预测器,预测生成过程中噪声分子的属性。 以时间为条件。
from cond_prediction.train_cond_predictor import get_cond_predictor_model
from data.aromatic_dataloader import create_data_loaders
from models_edm import get_model
from utils.helpers import switch_grad_off, get_edm_args, get_cond_predictor_args# 加载EDM 参数配置
args = get_edm_args("pre_trained/EDM")
# 加载条件控制模型参数配置
cond_predictor_args = get_cond_predictor_args("pre_trained/cond_pred")# 数据集的模拟,我们仅需要它来了解数据的形状及其统计数据。
dataset_mock = SimpleNamespace( num_node_features=12,num_targets=5,mean=torch.tensor([ 1.1734, -9.2780, -0.2356, 0.4042, 53.9615]),std=torch.tensor([0.5196, 0.3886, 0.0179, 0.0152, 1.6409]),
)
train_loader_mock = SimpleNamespace(dataset=dataset_mock)# 加载EDN模型
edm_model, nodes_dist, prop_dist = get_model(args, train_loader_mock, only_norm=True)
# 加载条件控制模型
cond_predictor = get_cond_predictor_model(cond_predictor_args, dataset_mock)
# 设置模型中的参数为不可训练
switch_grad_off([edm_model, cond_predictor])定义生成的内容:
batch_size - 有多少分子 - 影响运行时间。
n_rings 每个分子中环的数量。
Guiding_scale - 对具有较低目标功能的分子的引导强度 - 梯度标量影响生成分子的有效性与其目标分数之间的权衡。
它越高,有效分子的百分比就会减少。 然而,生成的有效分子将更接近定义的目标属性。
目标函数 - 我们想要最小化的函数。 预测属性的任意组合。
### 定义分子限制和目标函数 ###
batch_size = 10
n_rings = 8
guidance_scale = 1def target_function(_input, _node_mask, _edge_mask, _t):pred = cond_predictor(_input, _node_mask, _edge_mask, _t)pred = prop_dist.unnormalize(pred)gap = pred[:, 0]homo = pred[:, 1]ea = pred[:, 2] * 27.2114 # hartree to eVip = pred[:, 3] * 27.2114 # hartree to eVreturn -gap# return ip + ea + 3 * gap生成分子:
from sampling_edm import sample_guidance# 初始化生成分子,由于这里的模型是一个节点是一个环,所以节点比较奇怪
nodesxsample = torch.Tensor([n_rings] * batch_size).long()
# nodesxsample = nodes_dist.sample(args.batch_size)start_time = time()
x, one_hot, node_mask, edge_mask = sample_guidance(args,edm_model,target_function,nodesxsample,scale=guidance_scale,
)
print(f"Generated {x.shape[0]} molecules in {time() - start_time:.2f} seconds")使用 RDKit 评估分子的有效性并过滤掉无效分子:
target_function_values = get_target_function_values(x, one_hot, target_function, node_mask, edge_mask, edm_model)
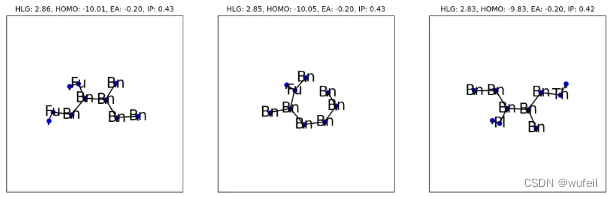
pred = prop_dist.unnormalize(predict(cond_predictor, x, one_hot, node_mask, edge_mask, edm_model))绘制最佳分子(具有较低的目标函数值)。 生成的分子表示为环图 (GOR),并带有表示环方向的加法节点。
from utils.plotting import plot_grap_of_rings_inner, plot_rdkitbest_idxs = target_function_values.argsort()
n_plot = min(3, x.shape[0])
fig, axes = plt.subplots(1, n_plot, figsize=(5*n_plot, 7))
for i, idx in enumerate(best_idxs[:n_plot]):pred_i = pred[idx]title = f"HLG: {pred_i[0]:.2f}, HOMO: {pred_i[1]:.2f}, EA: {pred_i[2]:.2f}, IP: {pred_i[3]:.2f}"plot_grap_of_rings_inner(axes[i], x[idx], one_hot.argmax(2)[idx], title=title, dataset=args.dataset, )
plt.show()
输出:
转换成完整的分子结构:
fig, axes = plt.subplots(1, n_plot, figsize=(5*n_plot, 7))
for i, idx in enumerate(best_idxs[:n_plot]):pred_i = pred[idx]title = f"HLG: {pred_i[0]:.2f}, HOMO: {pred_i[1]:.2f}, EA: {pred_i[2]:.2f}, IP: {pred_i[3]:.2f}"plot_rdkit(x[idx], one_hot.argmax(2)[idx], axes[i], title=title, dataset=args.dataset)
plt.show()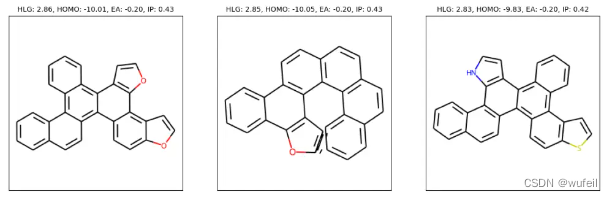
四、总结
1. GaUDI是一个梯度引导的分子生成扩散模型,可以在不重新训练分子生成模型的基础上,通过目标函数的梯度引导分子生成,从而产生特定属性的分子。文中以Gap等分子属性作为目标,验证了其方法的可行性。
2. 对于药物设计,分子设计等而言,该方法不能直接使用。首先,该模型对分子的表示,以环为节点,不符合我们几何神经网络中关于分子的设定,导致分子还原存在困难。这也是为什么上述结果中,有着GOA图的原因,以及有大量分子无法输出结构的原因。数据集中的分子均为多环结构,不是药物设计中常用的分子结构,模型的偏差很严重。
结论是,该模型不推荐,难用不友好,但是该方法提供了非常好的概念验证,值得进一步研究学习。
相关文章:

梯度引导的分子生成扩散模型- GaUDI 评测
GaUDI模型来自于以色列理工Tomer Weiss的2023年发表在预印本ChemRxiv上的工作 《Guided Diffusion for Inverse Molecular Design》。原文链接:Guided Diffusion for Inverse Molecular Design | Materials Chemistry | ChemRxiv | Cambridge Open Engage GaUDI模型…...

2023 年 亚太赛 APMCM ABC题 国际大学生数学建模挑战赛 |数学建模完整代码+建模过程全解全析
当大家面临着复杂的数学建模问题时,你是否曾经感到茫然无措?作为2022年美国大学生数学建模比赛的O奖得主,我为大家提供了一套优秀的解题思路,让你轻松应对各种难题。 以五一杯 A题为例子,以下是咱们做的一些想法呀&am…...
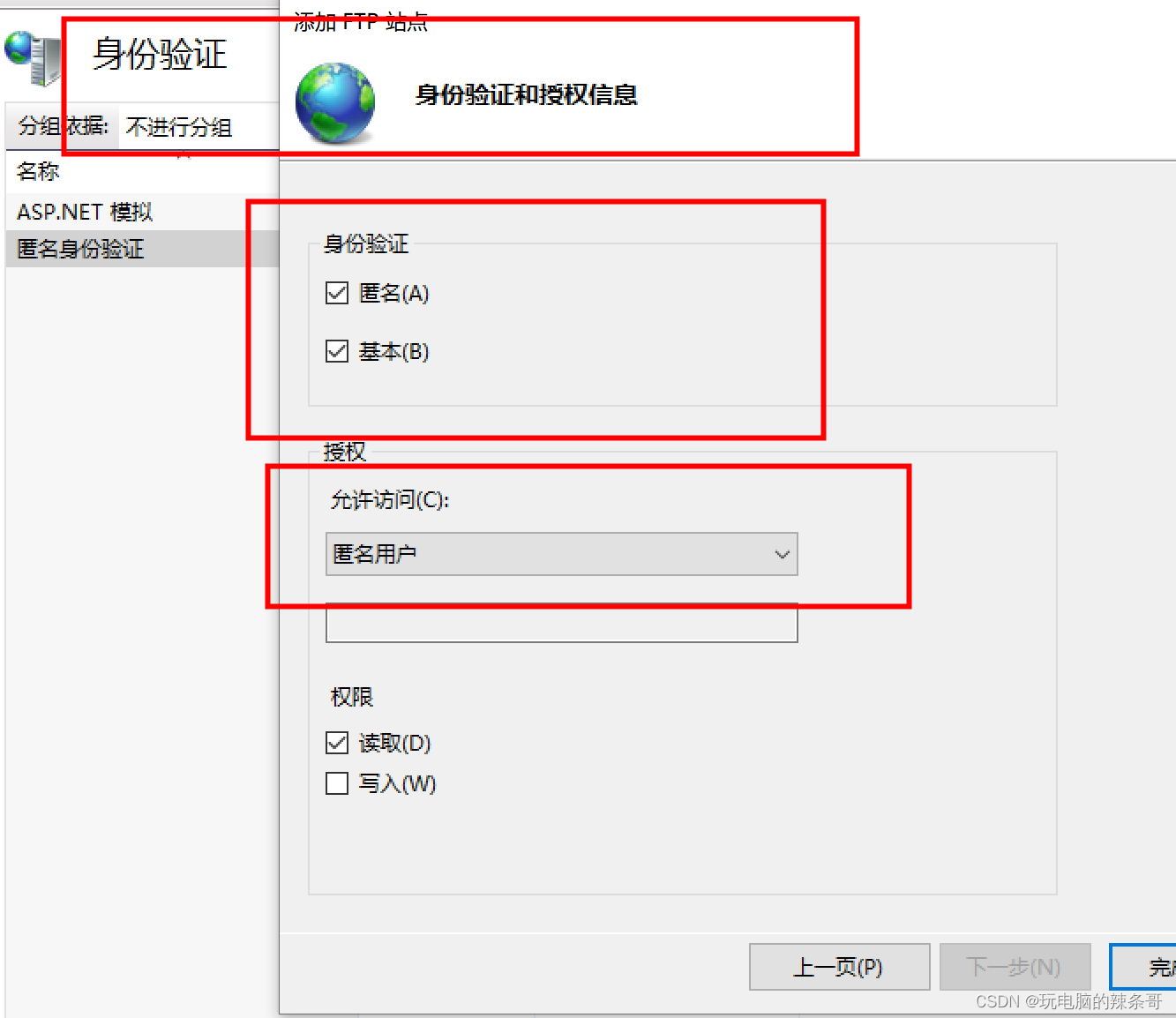
如何用cmd命令快速搭建FTP服务
环境: Win10专业版 问题描述: 如何用cmd命令快速搭建FTP服务 解决方案: 1.输入以下命令来安装IIS(Internet Information Services): dism /online /enable-feature /featurename:IIS-FTPServer /all …...
数据结构学习笔记——多维数组、矩阵与广义表
目录 一、多维数组(一)数组的定义(二)二维数组(三)多维数组的存储(四)多维数组的下标的相关计算 二、矩阵(一)特殊矩阵和稀疏矩阵(二)…...
C++之常用的排序算法
C之常用的排序算法 sort #include<iostream> using namespace std; #include<vector> #include<algorithm> #include<functional> void Myptint(int val) {cout << val << " "; }void test() {vector<int> v;v.push_back(…...
Mac中LaTex无法编译的问题
最近在使用TexStudio时,遇到一个棘手的问题: 无法编译,提示如下: kpathsea: Running mktexfmt xelatex.fmt /Library/TeX/texbin/mktexfmt: kpsewhich -var-valueTEXMFROOT failed, aborting early. BEGIN failed–compilation a…...
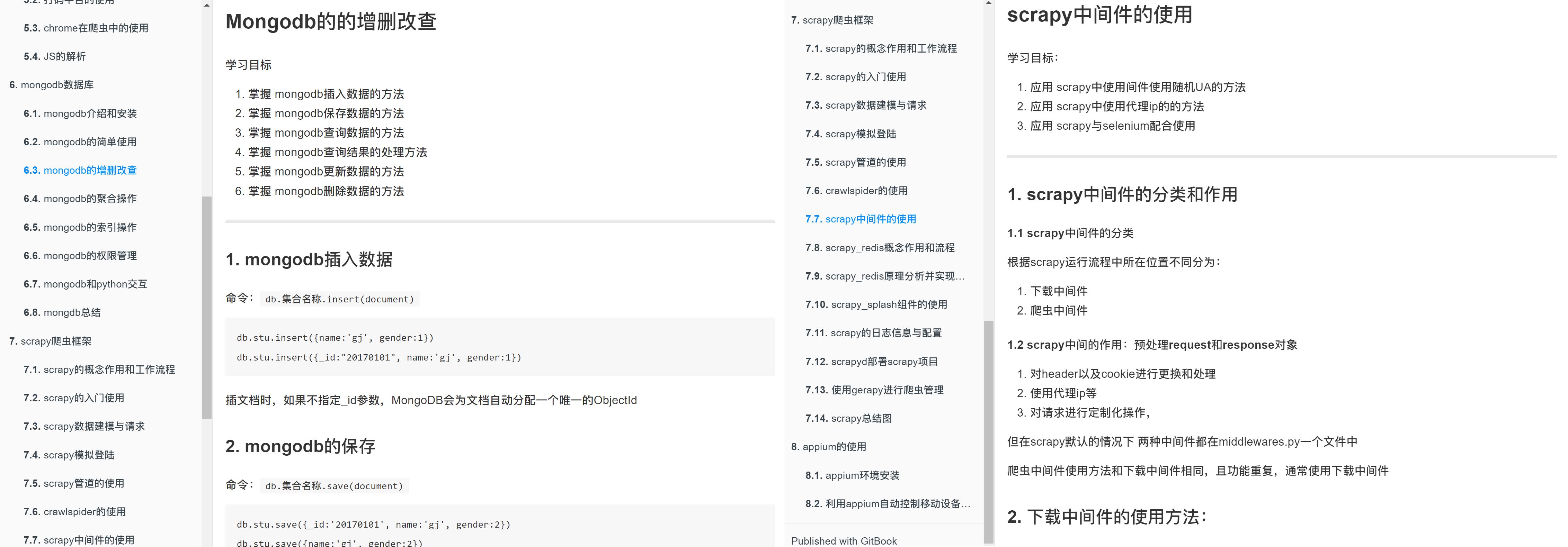
【Python爬虫】8大模块md文档集合从0到scrapy高手,第7篇:selenium 数据提取详解
本文主要学习一下关于爬虫的相关前置知识和一些理论性的知识,通过本文我们能够知道什么是爬虫,都有那些分类,爬虫能干什么等,同时还会站在爬虫的角度复习一下http协议。 爬虫全套笔记地址: 请移步这里 共 8 章&#x…...

【python基础(三)】操作列表:for循环、正确缩进、切片的使用、元组
文章目录 一. 遍历整个列表1. 在for循环中执行更多操作2. 在for循环结束后执行一些操作 二. 避免缩进错误三. 创建数值列表1. 使用函数range()2. 使用range()创建数字列表3. 指定步长。4. 对数字列表执行简单的统计计算5. 列表解析 五. 使用列表的一部分-切片1. 切片2. 遍历切片…...

使用VSCode调试全志R128的C906 RISC-V核心
使用 VSCode 调试 调试 XuanTie C906 核心 准备工具 T-Head DebugServer(CSkyDebugServer) - 搭建调试服务器 下载地址:T-Head DebugServer手册:T-Head Debugger Server User Guide驱动:cklink_dirvers VSCode - 开…...

Node.js之http模块
http模块是什么? http 模块是 Node,js 官方提供的、用来创建 web 服务器的模块。通过 http 模块提供的 http.createServer() 方法,就能方便的把一台普通的电脑,变成一台Web 服务器,从而对外提供 Web 资源服务。 如果我们想在node…...
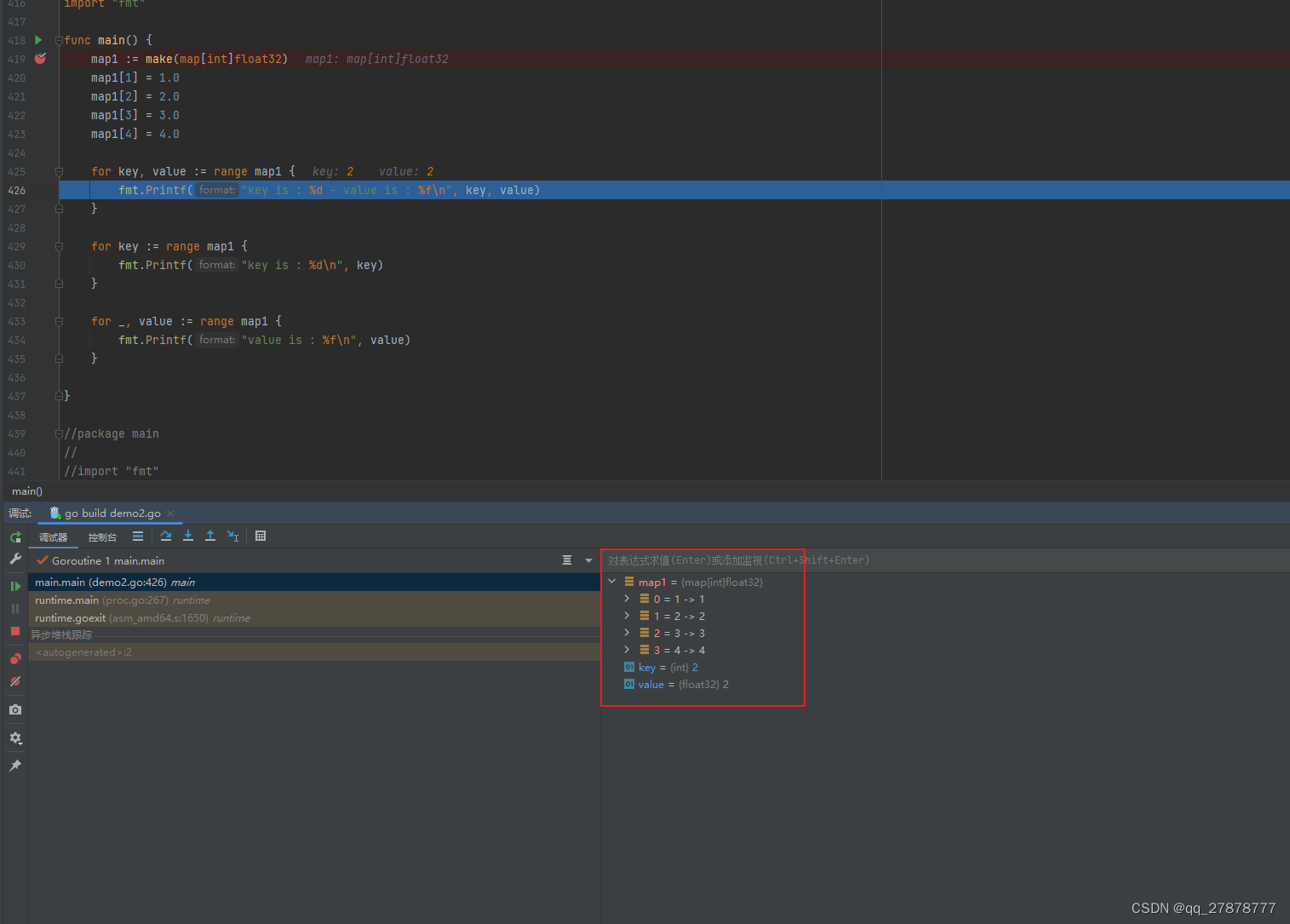
golang 断点调试
1.碰见如下报错,调试器没有打印变量信息 Delve is too old for Go version 1.21.2 (maximum supported version 1.19) 2. 解决办法 升级delve delve是go语言的debug工具。 go install github.com/go-delve/delve/cmd/dlvlatest报错 Get “https://proxy.golang.org/github…...

定时器如何计算触发频率?
定时器触发频率的计算公式为:定时器时钟频率/(预分频系数*计数周期1)。其中,定时器时钟频率是指定时器所连接的总线频率,预分频系数和计数周期需要根据具体的需求进行设置。预分频系数用于将总线频率分频,计…...

【数据库】数据库中的检查点Checkpoint,数据落盘的重要时刻
检查点(checkpoint) 专栏内容: 手写数据库toadb 本专栏主要介绍如何从零开发,开发的步骤,以及开发过程中的涉及的原理,遇到的问题等,让大家能跟上并且可以一起开发,让每个需要的人成为参与者。 本专栏会定…...
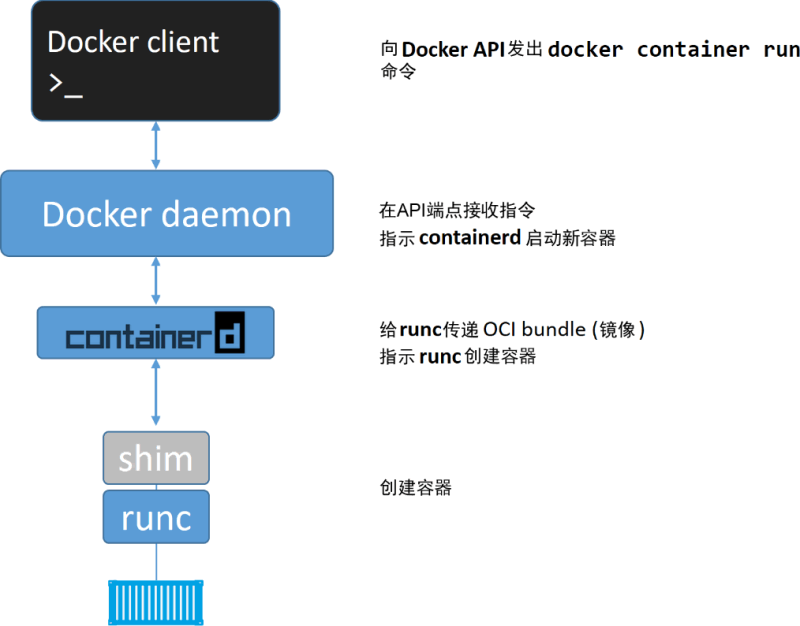
关于 Docker
关于 Docker 1. 术语Docker Enginedockerd(Docker daemon)containerdOCI (Open Container Initiative)runcDocker shimCRI (Container Runtime Interface)CRI-O 2. 容器启动过程在 Linux 中的实现daemon 的作用 Docker 是个划时代的开源项目,…...

LeetCode解法汇总2342. 数位和相等数对的最大和
目录链接: 力扣编程题-解法汇总_分享记录-CSDN博客 GitHub同步刷题项目: https://github.com/September26/java-algorithms 原题链接:力扣(LeetCode)官网 - 全球极客挚爱的技术成长平台 描述: 给你一个下…...

数据库的级联删除
级联删除是指在数据库中删除一个对象时,与该对象有关的其他对象也被自动删除。在 Django 中,级联删除通常通过在模型中定义外键时使用 on_delete 参数来实现。以下是一些常见的 on_delete 选项: 1.models.CASCADE: 当关联的对象被删除时&…...

【Python 千题 —— 基础篇】奇数列表
题目描述 题目描述 创建奇数列表。使用 for 循环创建一个包含 20 以内奇数的列表。 输入描述 无输入。 输出描述 输出创建的列表。 示例 示例 ① 输出: 创建的奇数列表为: [1, 3, 5, 7, 9, 11, 13, 15, 17, 19]代码讲解 下面是本题的代码: #…...

当npm下载库失败时可以用cnpm替代
下载cnpm npm install -g cnpm --registryhttp://registry.npmmirror.com 然后使用cnpm代替npm下载即可 cnpm install...

PyTorch多GPU训练时同步梯度是mean还是sum?
PyTorch 通过两种方式可以进行多GPU训练: DataParallel, DistributedDataParallel. 当使用DataParallel的时候, 梯度的计算结果和在单卡上跑是一样的, 对每个数据计算出来的梯度进行累加. 当使用DistributedDataParallel的时候, 每个卡单独计算梯度, 然后多卡的梯度再进行平均.…...

Spring Framework IoC依赖注入-按Bean类型注入
Spring Framework 作为一个领先的企业级开发框架,以其强大的依赖注入(Dependency Injection,DI)机制而闻名。DI使得开发者可以更加灵活地管理对象之间的关系,而不必过多关注对象的创建和组装。在Spring Framework中&am…...

日语AI面试高效通关秘籍:专业解读与青柚面试智能助攻
在如今就业市场竞争日益激烈的背景下,越来越多的求职者将目光投向了日本及中日双语岗位。但是,一场日语面试往往让许多人感到步履维艰。你是否也曾因为面试官抛出的“刁钻问题”而心生畏惧?面对生疏的日语交流环境,即便提前恶补了…...
:OpenBCI_GUI:从环境搭建到数据可视化(下))
脑机新手指南(八):OpenBCI_GUI:从环境搭建到数据可视化(下)
一、数据处理与分析实战 (一)实时滤波与参数调整 基础滤波操作 60Hz 工频滤波:勾选界面右侧 “60Hz” 复选框,可有效抑制电网干扰(适用于北美地区,欧洲用户可调整为 50Hz)。 平滑处理&…...

ubuntu搭建nfs服务centos挂载访问
在Ubuntu上设置NFS服务器 在Ubuntu上,你可以使用apt包管理器来安装NFS服务器。打开终端并运行: sudo apt update sudo apt install nfs-kernel-server创建共享目录 创建一个目录用于共享,例如/shared: sudo mkdir /shared sud…...

Python:操作 Excel 折叠
💖亲爱的技术爱好者们,热烈欢迎来到 Kant2048 的博客!我是 Thomas Kant,很开心能在CSDN上与你们相遇~💖 本博客的精华专栏: 【自动化测试】 【测试经验】 【人工智能】 【Python】 Python 操作 Excel 系列 读取单元格数据按行写入设置行高和列宽自动调整行高和列宽水平…...

工程地质软件市场:发展现状、趋势与策略建议
一、引言 在工程建设领域,准确把握地质条件是确保项目顺利推进和安全运营的关键。工程地质软件作为处理、分析、模拟和展示工程地质数据的重要工具,正发挥着日益重要的作用。它凭借强大的数据处理能力、三维建模功能、空间分析工具和可视化展示手段&…...
srs linux
下载编译运行 git clone https:///ossrs/srs.git ./configure --h265on make 编译完成后即可启动SRS # 启动 ./objs/srs -c conf/srs.conf # 查看日志 tail -n 30 -f ./objs/srs.log 开放端口 默认RTMP接收推流端口是1935,SRS管理页面端口是8080,可…...

基于Docker Compose部署Java微服务项目
一. 创建根项目 根项目(父项目)主要用于依赖管理 一些需要注意的点: 打包方式需要为 pom<modules>里需要注册子模块不要引入maven的打包插件,否则打包时会出问题 <?xml version"1.0" encoding"UTF-8…...
Map相关知识
数据结构 二叉树 二叉树,顾名思义,每个节点最多有两个“叉”,也就是两个子节点,分别是左子 节点和右子节点。不过,二叉树并不要求每个节点都有两个子节点,有的节点只 有左子节点,有的节点只有…...
Yolov8 目标检测蒸馏学习记录
yolov8系列模型蒸馏基本流程,代码下载:这里本人提交了一个demo:djdll/Yolov8_Distillation: Yolov8轻量化_蒸馏代码实现 在轻量化模型设计中,**知识蒸馏(Knowledge Distillation)**被广泛应用,作为提升模型…...

Git 3天2K星标:Datawhale 的 Happy-LLM 项目介绍(附教程)
引言 在人工智能飞速发展的今天,大语言模型(Large Language Models, LLMs)已成为技术领域的焦点。从智能写作到代码生成,LLM 的应用场景不断扩展,深刻改变了我们的工作和生活方式。然而,理解这些模型的内部…...
