单细胞转录组 —— kb-python 原始数据处理
单细胞转录组 —— kb-python 原始数据处理
前言
kallisto|bustools 是一种用于预处理 scRNA-seq 数据的工作流程。
数据预处理步骤包括:
- 将
reads与其来源细胞关联起来; - 根据唯一分子标识符(
UMI)对reads进行去重; - 从
reads中生成基因或特征计数,以生成 细胞x基因 矩阵。
使用 kallisto|bustools 有以下几点优势
- 生成与 细胞x基因 或 细胞x转录本 等价的计数矩阵
- 执行
RNA速率分析和snRNA-seq分析 - 对
10x、inDrops和Dropseq等多种技术的数据进行定量。 - 为新技术和方案定制工作流程。
- 处理特征条码数据,如
CITE-seq、REAP-seq、MULTI-seq、Clicktags和Perturb-seq。 - 生成
QC报告 - 速度飞快
使用
kb-python 是一个用于处理 scRNA 序列的 python 软件包,它封装了 kallisto|bustools 单细胞 RNA 分析流程。
可以使用 pip 安装
pip install kb-python
也可以使用 conda 安装
conda install -c bioconda kb-python
或者安装开发版
pip install git+https://github.com/pachterlab/kb_python
命令行输入 kb,输出类似如下信息
usage: kb [-h] [--list] <CMD> ...kb_python 0.28.2positional arguments:<CMD>info Display package and citation informationcompile Compile `kallisto` and `bustools` binaries from sourceref Build a kallisto index and transcript-to-gene mappingcount Generate count matrices from a set of single-cell FASTQ filesoptional arguments:-h, --help Show this help message and exit--list Display list of supported single-cell technologies
主要包含 4 个子命令,我们主要使用后两个。
构建索引
可通过 ref 子命令,使用 kallisto 建立转录组索引。只需传入参考基因组和基因组注释文件即可。
重要参数如下

流程主要包含 4 种类型,默认是 standard,RNA 速率分析用 nac,kite 主要用于 Feature Barcode 测序,如 10x Feature Barcode 技术可以同时分析基因表达和免疫受体序列(BCR 和 TCR)。
也可以使用 custom 模式,定制自己的参考基因组索引。
定量
重要参数如下

kb-python 支持的单细胞技术可以使用下面的命令查看
kb --list

其中,后三列的数字代表索引位置信息:文件索引(0 表示 R1), 起始位置, 结束位置。如果为 None,说明整个文件都是。
例如,10XV2 表示 10x 的 V2 版,barcode 和 UMI 都在 R1 中,前 16 位为 barcode 序列,后 10 位为 UMI 序列,R2 都是 cDNA 序列
实战
10x scRNA-seq
我们以昨天的数据为例,使用 kb-python 来进行原始数据的处理
创建参考基因组的索引,使用 standard 模式
kb ref -i mm10.standard.idx -g t2g.txt \-f1 cdna.fa -f2 intron.fa \--workflow standard \mm10.fa \mm10.refGene.gtf
定量分析,多个 FASTQ 文件要按照文件排序,同一个 lane 的文件要先 R1 再 R2
kb count -i mm10.standard.idx \-g t2g.txt -x 10xv2 \-o output -t 2 --workflow standard \--h5ad --cellranger --filter bustools \GSM4812353_S1_L001_R1_001.fastq.gz \GSM4812353_S1_L001_R2_001.fastq.gz \... \GSM4812353_S1_L002_R1_001.fastq.gz \GSM4812353_S1_L002_R2_001.fastq.gz
输出结果的结构大致如下
├── 10x_version2_whitelist.txt
├── counts_filtered
│ ├── adata.h5ad
│ ├── cells_x_genes.barcodes.txt
│ ├── cells_x_genes.genes.names.txt
│ ├── cells_x_genes.genes.txt
│ └── cells_x_genes.mtx
├── counts_unfiltered
│ ├── adata.h5ad
│ ├── cellranger
│ │ ├── barcodes.tsv
│ │ ├── genes.tsv
│ │ └── matrix.mtx
│ ├── cells_x_genes.barcodes.txt
│ ├── cells_x_genes.genes.names.txt
│ ├── cells_x_genes.genes.txt
│ └── cells_x_genes.mtx
├── filter_barcodes.txt
├── inspect.json
├── kb_info.json
├── matrix.ec
├── output.bus
├── output.filtered.bus
├── output.unfiltered.bus
├── run_info.json
└── transcripts.txt
可以使用 counts_filtered/adata.h5ad 作为下游分析的起点。
因为我们设置了 --cellranger 参数,所以输出目录中也有 cellranger 结构的输出结果。
不推荐使用 --report 输出报告,使用时报了很多错误。
10x snRNA-seq
我们以 10x 的测序数据 1k Brain Nuclei from an E18 Mouse 为例。
我们将生成两个矩阵:一个是剪接转录本矩阵,另一个是未剪接转录本矩阵,然后将它们相加得出核转录本总数。
首先,下载数据
wget https://caltech.box.com/shared/static/j337aflq9ublmwaripkepob41mr23216.txt -O checksums.txt
wget https://caltech.box.com/shared/static/2j8shgwmalzcjawuow51678a8yssvdef.gz -O nuclei_900_S1_L001_R1_001.fastq.gz
wget https://caltech.box.com/shared/static/k2yydqlz2jtckw1shk5h536mxn47nm9n.gz -O nuclei_900_S1_L001_R2_001.fastq.gz
wget https://caltech.box.com/shared/static/tlqdm0w3tvy8ogyktsz7ahggwurc6kkj.gz -O nuclei_900_S1_L002_R1_001.fastq.gz
wget https://caltech.box.com/shared/static/gqrvkqllr9d7zq4e3yfrng9kgfbejowe.gz -O nuclei_900_S1_L002_R2_001.fastq.gz
校验下载文件是否完整
md5sum -c checksums.txt --ignore-missing
# nuclei_900_S1_L001_R1_001.fastq.gz: 成功
# nuclei_900_S1_L001_R2_001.fastq.gz: 成功
# nuclei_900_S1_L002_R1_001.fastq.gz: 成功
# nuclei_900_S1_L002_R2_001.fastq.gz: 成功
下载参考基因组信息,如果已经下载过,可以直接使用本地文件
wget ftp://ftp.ensembl.org/pub/release-98/fasta/mus_musculus/dna/Mus_musculus.GRCm38.dna.primary_assembly.fa.gz
wget ftp://ftp.ensembl.org/pub/release-98/gtf/mus_musculus/Mus_musculus.GRCm38.98.gtf.gz
构建小鼠的 DNA 和内含子索引,使用 nac 模式,后续可以进行 RNA 速率分析
kb ref -i index.idx -g t2g.txt \-f1 cdna.fa -f2 intron.fa \-c1 cdna_t2c.txt -c2 intron_t2c.txt \--workflow nac \Mus_musculus.GRCm38.dna.primary_assembly.fa.gz \Mus_musculus.GRCm38.98.gtf.gz
定量分析
kb count -i index.idx \-g t2g.txt -c1 cdna_t2c.txt \-c2 intron_t2c.txt -x 10xv2 \-o output -t 2 --workflow nac --h5ad \nuclei_900_S1_L001_R1_001.fastq.gz \nuclei_900_S1_L001_R2_001.fastq.gz \nuclei_900_S1_L002_R1_001.fastq.gz \nuclei_900_S1_L002_R2_001.fastq.gz
10x Feature Barcode
我们使用 Kallisto Indexing and Tag Extraction (KITE) 流程对 10x Genomics pbmc_1k_protein_v3 特征条形码数据集进行预处理和分析。
在特征条形码芯片是建立在 scRNA-seq 的基础上,可以同时获得大量单个细胞中基因的表达量和细胞表面蛋白的表达情况,并将细胞数据记录为短 DNA 序列
KITE 处理流程会生成 错配图(Mismatch Map),其中包含实验中使用的所有特征条形码序列及其所有单碱基错配。
错配图用于生成转录本到基因的映射文件(.t2g)和 fasta文件,作为 kallisto 的输入。
使用 kallisto index 建立索引后,kallisto|bustools 就能有效地搜索测序数据,查找错配图中的序列。
下载数据
wget -q https://caltech.box.com/shared/static/asmj4nu90ydhsrk3pm7aaxu00cnnfige.txt -O checksums.txt
wget -q https://caltech.box.com/shared/static/mp2vr3p6dztdyatuag8ir3cektmrztg8.gz -O pbmc_1k_protein_v3_antibody_S2_L001_R1_001.fastq.gz
wget -q https://caltech.box.com/shared/static/f3payi1za7mn0jfai7vm10sy3yqwgpqh.gz -O pbmc_1k_protein_v3_antibody_S2_L001_R2_001.fastq.gz
wget -q https://caltech.box.com/shared/static/e112bbczh9o1rl6gfin36bqp0ga7uvdy.gz -O pbmc_1k_protein_v3_antibody_S2_L002_R1_001.fastq.gz
wget -q https://caltech.box.com/shared/static/3ve2axc8dr8v5nnrhmynrdgpqj6xg42k.gz -O pbmc_1k_protein_v3_antibody_S2_L002_R2_001.fastq.gz
检查数据的完整性
md5sum -c checksums.txt --ignore-missing
创建错配索引
kb 能够生成一个 FASTA 文件,其中包含所有汉明距离小于 2 的特征条形码变体,并创建这些序列的 kallisto 索引。
要做到这一点,我们首先需要准备一个 TSV 文件,其中第一列包含特征条形码序列,第二列包含特征条形码名称。
首先,我们下载 10x Genomics 提供的 feature reference文件。
wget -q http://cf.10xgenomics.com/samples/cell-exp/3.0.0/pbmc_1k_protein_v3/pbmc_1k_protein_v3_feature_ref.csv
使用 pandas 处理成 kb-python 接受的输入形式
import pandas as pddf = pd.read_csv('pbmc_1k_protein_v3_feature_ref.csv')
df[['sequence', 'id']].to_csv('features.tsv', index=None, header=None, sep='\t')
创建索引
kb ref -i mismatch.idx -f1 mismatch.fa -g t2g.txt --workflow kite features.tsv
定量
kb count --h5ad -i mismatch.idx \-g t2g.txt -x 10xv3 --workflow kite -t 8 \pbmc_1k_protein_v3_antibody_S2_L001_R1_001.fastq.gz \pbmc_1k_protein_v3_antibody_S2_L001_R2_001.fastq.gz \pbmc_1k_protein_v3_antibody_S2_L002_R1_001.fastq.gz \pbmc_1k_protein_v3_antibody_S2_L002_R2_001.fastq.gz
Drop-seq
数据集中 GSE178612 研究的是 FoxM1 与 Rb 基因在小鼠乳腺癌中的相互作用。
我们选择其中一个样本 GSM5394388,下载其原始数据
prefetch SRR14872449
解压
fastq-dump SRR14872449/SRR14872449.sra --split-files --gzip -O SRR14872449
表达定量,使用之前构建好的小鼠的索引
kb count --h5ad -i mm10.standard.idx \-g t2g.txt -x DROPSEQ --workflow standard -t 8 \--h5ad --cellranger --filter bustools \SRR14872449/SRR14872449_1.fastq.gz \SRR14872449/SRR14872449_2.fastq.gz \
Indrop
GSE111672 数据集中包含 6 例原发性胰腺癌组织的单细胞 RNA 测序和空间转录组学,
我们随便选择一个样本 GSM3036909 下载原始数据
prefetch SRR6825055
解压
fastq-dump SRR6825055/SRR6825055.sra --split-files --gzip -O SRR6825055
其中 R1 长度为 35,R2 长度为 51,是 Indrop-seq 的 V2 版
不同版本之间的差别:
v1:原始版,其中R2为cDNA序列,R1为元数据(UMI和barcode)。v2:v1的反转,R1和R2的内容互换v3:2016年夏季重新设计,需要手动解复用。R1是cDNA序列,R2包含凝胶条形码的前半部分,R3包含文库索引,R4包含凝胶条形码的后半部分、UMI和部分polyA尾部。
下载构建好的人类参考基因组索引
mkdir human
kb ref -d human \-i human/kb_ref.idx \-g human/t2g.txt
表达定量
kb count --h5ad -i human/kb_ref.idx \-g human/t2g.txt -x INDROPSV2 \--workflow standard -t 8 \--h5ad --cellranger --filter bustools \SRR6825055/SRR6825055_1.fastq.gz \SRR6825055/SRR6825055_2.fastq.gz \
SMART-seq2
我们从研究人类皮质球体内星形胶质细胞的成熟数据 GSE99951 中,下载 GSM2665701 样本进行分析
prefetch SRR5676730
解压
fastq-dump SRR5676730/SRR5676730.sra --split-files --gzip -O SRR5676730
表达定量,不支持 --filter 参数,同时必须加上 --parity 参数
kb count --h5ad -i human/kb_ref.idx \-g human/t2g.txt -x SMARTSEQ2 -t 8 \--parity paired --workflow standard \--h5ad --cellranger \SRR5676730/SRR5676730_1.fastq.gz \SRR5676730/SRR5676730_2.fastq.gz \
其他测序数据可以自行探索一下,前面几种用法基本涵盖了。
相关文章:

单细胞转录组 —— kb-python 原始数据处理
单细胞转录组 —— kb-python 原始数据处理 前言 kallisto|bustools 是一种用于预处理 scRNA-seq 数据的工作流程。 数据预处理步骤包括: 将 reads 与其来源细胞关联起来;根据唯一分子标识符(UMI)对 reads 进行去重࿱…...
全同态加密算法概览
我们前面有谈到《Paillier半同态加密算法》,半同态加密算法除了支持密文加法运算的 Paillier 算法,还有支持密文乘法计算的 RSA 算法,早期的PSI(隐私求交)和PIR(匿踪查询)都有使用基于RSA盲签名技术来实现。今天我们来谈谈能够有效支持任意函…...
)
leetcode 刷题day38动态规划Part07 打家劫舍(198.打家劫舍、213.打家劫舍II、337.打家劫舍III)
198.打家劫舍 思路: 1、dp[i]为到第i家偷到的最高金额。 2、如果偷第i家,那么dp[i]dp[i-2]nums[i],如果不偷,则dp[i]dp[i-1],所以递推公式dp[i]max(dp[i-2]nums[i],dp[i-1])。 3、初始值,根据递推公式,我们…...

C0010.Qt5.15.2下载及安装方法
1. 下载及安装 Qt 添加链接描述下载地址:http://download.qt.io/ 选择 archive 目录 安装Qt **注意:**本人使用的是Qt5.15.2版本,可以按如下方法找到该版本;...

制造企业MES管理系统的应用策略与实施路径
在智能制造浪潮的席卷之下,MES管理系统作为连接生产计划与车间操作的核心桥梁,其战略地位愈发显著。本文旨在深入剖析MES管理系统在智能制造转型中的核心价值、实施策略及实践路径,为制造企业探索智能化生产之路提供实践指导与灵感启发。 MES…...

Halcon 3D应用 - 胶路提取
1. 需求 本文基于某手环(拆机打磨处理)做的验证性工作,为了项目保密性,只截取部分数据进行测试。 这里使用的是海康3D线激光轮廓相机直线电机的方式进行的高度数据采集,我们拿到的是高度图亮度图数据。 提取手环上的胶…...

【Redis】Redis线程模型
目录 1. Redis 是单线程的,还是多线程的?2. Redis单线程模式是怎么样的?Redis 单线程模式的优势Redis 单线程的局限性Redis 单线程的优化策略 3. Redis采用单线程为什么还这么快4. Redis 6.0 之前为什么使用单线程?5. Redis 6.0 之…...
Electron构建桌面应用程序,服务于项目的自主学习记录(持续更新...
无所畏惧地面对未知,并将其视为成长的机会 大纲官网快速入门1.安装node.js -- 这里推荐用nvm管理2.脚手架创建3.electron 包安装到应用的开发依赖4.创建主进程(main.js)并启动项目1.创建页面2.配置main.js3.启动项目 -- 效果 进阶 -- 基于项目场景功能使用场景一&am…...

linux Load Average 计算
在内核代码 kernel/sched/loadavg.c 中有一个公式: a1 a0 * e a * (1 - e) 此算法是指数加权移动平均法(Exponential Weighted Moving Average,EMWA),是一种特殊的加权移动平均法,它考虑当前和历史的所有数据&#…...

pandas常用数据格式IO性能对比
前言 本文对pandas支持的一些数据格式进行IO(读写)的性能测试,大数据时代以数据为基础,经常会遇到操作大量数据的情景,数据的IO性能尤为重要,本文对常见的数据格式csv、feather、hdf5、jay、parquet、pick…...

【D3.js in Action 3 精译_031】3.5.2 DIY实战:在 Observable 平台实现带数据标签的 D3 条形图并改造单元测试模块
当前内容所在位置(可进入专栏查看其他译好的章节内容) 第一部分 D3.js 基础知识 第一章 D3.js 简介(已完结) 1.1 何为 D3.js?1.2 D3 生态系统——入门须知1.3 数据可视化最佳实践(上)1.3 数据可…...

华为OD机试真题-字符串分割
题目描述: 给定非空字符串s,将该字符串分割成一些子串,使每个子串的ASCII码值的和均为水仙花数。 1、若分割不成功,则返回0。 2、若分割成功且分割结果不唯一,则返回-1。 3、若分割成功且分割结果唯一,则返…...
)
编程技巧:提高代码健壮性与可维护性的关键方法(以 Shell 为例)
在脚本编写和自动化工作中,良好的编程技巧对于确保代码的健壮性和可维护性至关重要。以下是一些关键的编程技巧,包括模块化设计、单元测试、版本控制、处理边界条件、错误处理、中间值保存和创建 Flag。本文将通过 Shell 脚本示例来阐述这些技巧的应用。 1. 模块化设计 **定…...

【无标题】ReadableStream is not defined
升级 node 版本到 18 及以上即可解决...

【JVM】高级篇
1 GraalVM 1.1 什么是GraalVM GraalVM是Oracle官方推出的一款高性能JDK,使用它享受比OpenJDK或者OracleJDK更好的性能。 GraalVM的官方网址:https://www.graalvm.org/ 官方标语:Build faster, smaller, leaner applications。 更低的CPU…...

nacos1.4源码-服务发现、心跳机制
nacos的服务发现主要采用服务端主动推送客户端定时拉取;心跳机制通过每5s向服务端发送心跳任务来保活,当超过15s服务端未接收到心跳任务时,将该实例设置为非健康状态;当超过30s时,删除该实例。 1.服务发现 nacos主要采…...

C++ 2D平台游戏开发案例
关于2D平台游戏的C开发案例,包括游戏设计、实现细节、图形渲染和音效处理等内容。虽然无法一次性提供3000字,但我会尽量详细描述各个部分,并确保有足够的深度和广度。 2D平台游戏开发案例 一、游戏设计 游戏概述 游戏名称:“冒险…...

【Webpack--019】TreeShaking
🤓😍Sam9029的CSDN博客主页:Sam9029的博客_CSDN博客-前端领域博主 🐱🐉若此文你认为写的不错,不要吝啬你的赞扬,求收藏,求评论,求一个大大的赞!👍* &#x…...

Docker基本操作命令
Docker 是一个开源的应用容器引擎,允许开发者打包应用以及其依赖包到一个可移植的容器中,然后发布到任何流行的 Linux 机器上,也可以实现虚拟化。容器是完全使用沙箱机制,相互之间不会有任何接口。主要功能是为开发者提供一个简单…...

开源计算器应用的全面测试计划:确保功能性和可靠性
✅作者简介:2022年博客新星 第八。热爱国学的Java后端开发者,修心和技术同步精进。 🍎个人主页:Java Fans的博客 🍊个人信条:不迁怒,不贰过。小知识,大智慧。 💞当前专栏…...
eNSP-Cloud(实现本地电脑与eNSP内设备之间通信)
说明: 想象一下,你正在用eNSP搭建一个虚拟的网络世界,里面有虚拟的路由器、交换机、电脑(PC)等等。这些设备都在你的电脑里面“运行”,它们之间可以互相通信,就像一个封闭的小王国。 但是&#…...
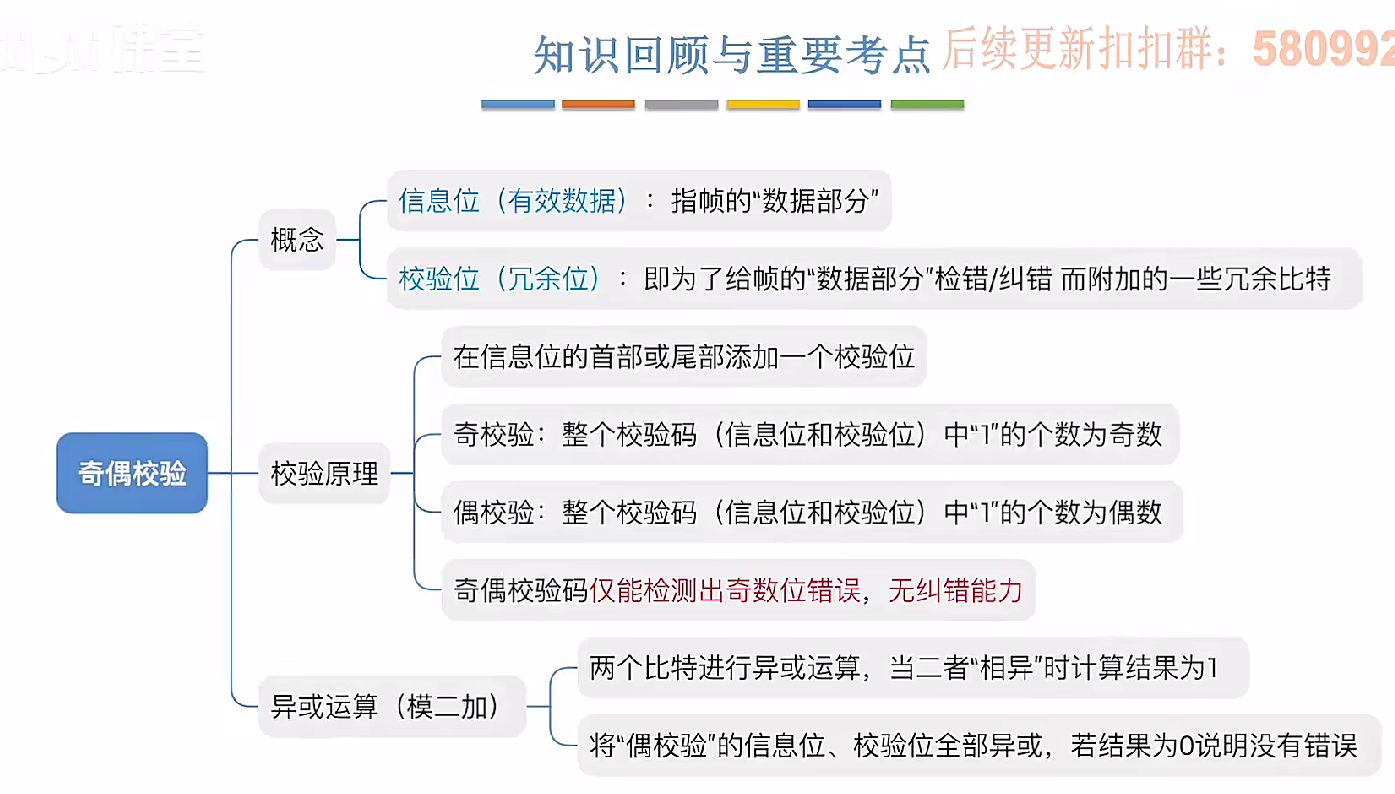
3.3.1_1 检错编码(奇偶校验码)
从这节课开始,我们会探讨数据链路层的差错控制功能,差错控制功能的主要目标是要发现并且解决一个帧内部的位错误,我们需要使用特殊的编码技术去发现帧内部的位错误,当我们发现位错误之后,通常来说有两种解决方案。第一…...
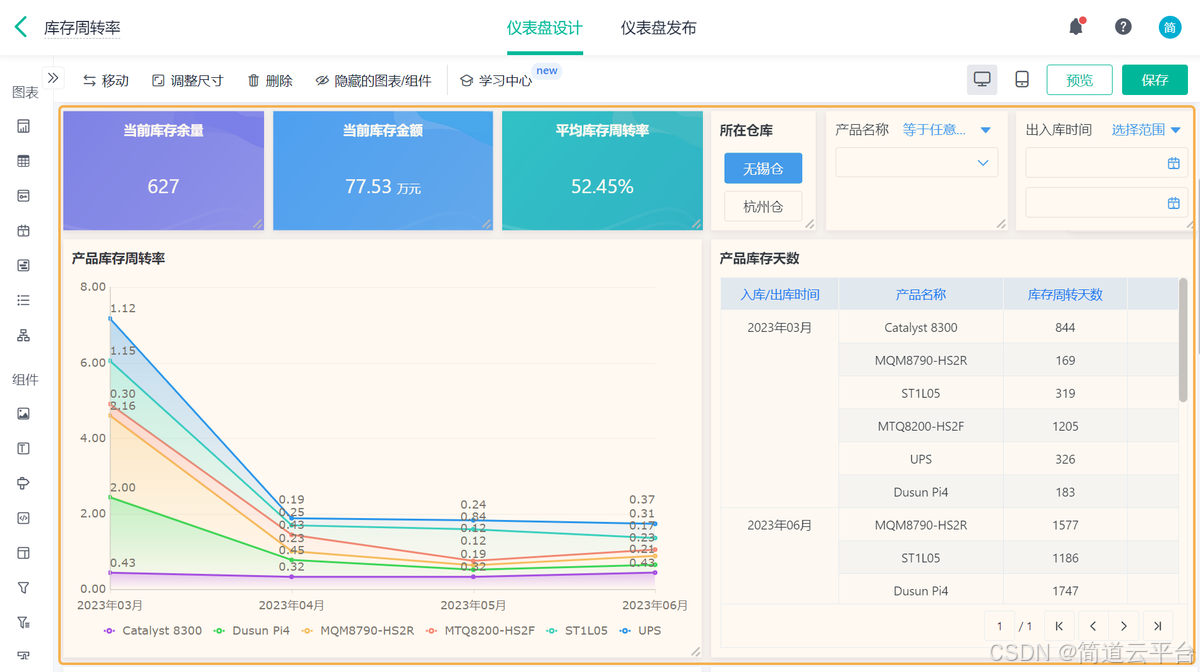
什么是库存周转?如何用进销存系统提高库存周转率?
你可能听说过这样一句话: “利润不是赚出来的,是管出来的。” 尤其是在制造业、批发零售、电商这类“货堆成山”的行业,很多企业看着销售不错,账上却没钱、利润也不见了,一翻库存才发现: 一堆卖不动的旧货…...
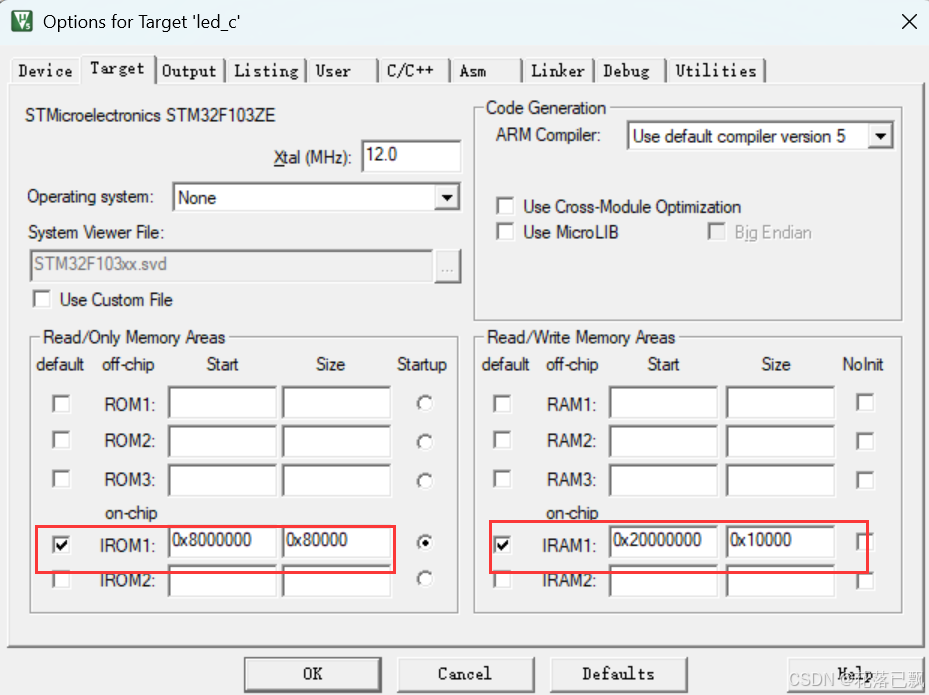
Keil 中设置 STM32 Flash 和 RAM 地址详解
文章目录 Keil 中设置 STM32 Flash 和 RAM 地址详解一、Flash 和 RAM 配置界面(Target 选项卡)1. IROM1(用于配置 Flash)2. IRAM1(用于配置 RAM)二、链接器设置界面(Linker 选项卡)1. 勾选“Use Memory Layout from Target Dialog”2. 查看链接器参数(如果没有勾选上面…...
基础光照(Basic Lighting))
C++.OpenGL (10/64)基础光照(Basic Lighting)
基础光照(Basic Lighting) 冯氏光照模型(Phong Lighting Model) #mermaid-svg-GLdskXwWINxNGHso {font-family:"trebuchet ms",verdana,arial,sans-serif;font-size:16px;fill:#333;}#mermaid-svg-GLdskXwWINxNGHso .error-icon{fill:#552222;}#mermaid-svg-GLd…...
)
【服务器压力测试】本地PC电脑作为服务器运行时出现卡顿和资源紧张(Windows/Linux)
要让本地PC电脑作为服务器运行时出现卡顿和资源紧张的情况,可以通过以下几种方式模拟或触发: 1. 增加CPU负载 运行大量计算密集型任务,例如: 使用多线程循环执行复杂计算(如数学运算、加密解密等)。运行图…...

EtherNet/IP转DeviceNet协议网关详解
一,设备主要功能 疆鸿智能JH-DVN-EIP本产品是自主研发的一款EtherNet/IP从站功能的通讯网关。该产品主要功能是连接DeviceNet总线和EtherNet/IP网络,本网关连接到EtherNet/IP总线中做为从站使用,连接到DeviceNet总线中做为从站使用。 在自动…...

【JavaSE】绘图与事件入门学习笔记
-Java绘图坐标体系 坐标体系-介绍 坐标原点位于左上角,以像素为单位。 在Java坐标系中,第一个是x坐标,表示当前位置为水平方向,距离坐标原点x个像素;第二个是y坐标,表示当前位置为垂直方向,距离坐标原点y个像素。 坐标体系-像素 …...
中的KV缓存压缩与动态稀疏注意力机制设计)
大语言模型(LLM)中的KV缓存压缩与动态稀疏注意力机制设计
随着大语言模型(LLM)参数规模的增长,推理阶段的内存占用和计算复杂度成为核心挑战。传统注意力机制的计算复杂度随序列长度呈二次方增长,而KV缓存的内存消耗可能高达数十GB(例如Llama2-7B处理100K token时需50GB内存&a…...
)
Angular微前端架构:Module Federation + ngx-build-plus (Webpack)
以下是一个完整的 Angular 微前端示例,其中使用的是 Module Federation 和 npx-build-plus 实现了主应用(Shell)与子应用(Remote)的集成。 🛠️ 项目结构 angular-mf/ ├── shell-app/ # 主应用&…...
