日常吐槽。
一、写在前面
stereopy日常出bug(github issue里得有一半的问题是我提的,当然也有可能是因为我菜),stereopy自己生成的anndata自己不能计算空间共现关系,还是靠squidpy才能计算。另外还要一些函数一开并行计算就报错,这里留一些stereopy对象的转换/输出方式,以备大家不(sui)时之(pao)需(lu)。更多空转教程可见:空间转录组学习手册合辑

二、stereopy相关数据读写
Stereopy是一个基础且全面的空间转录组分析工具,其支持空间高变基因计算、RNA Velocity、批次整合、SingleR注释、细胞通讯、基因调控网络、轨迹推断等基础/进阶分析。此外,针对多样本的数据,Stereopy还支持3D细胞通讯、3D轨迹推断、3D基因调控网络等分析。学习下面的内容前需要大家先掌握python知识:生信Python速查手册
文件读取、输出、转换:
1.1 GEM与GEF格式文件
一共有GEM、GEF两种格式的数据。GEM文件包括:切片中x、y轴空间坐标信息,MIDCount为基因表达量,ExonCount为由SAW(Version >= 5.1.3)中的spatial_RNA_visualization_v5生成的外显子表达量。
数据格式包含以下信息:

GEF分为Square Bin与Cell Bin两种,前者的前缀一般为.raw.gef/.gef/.tissue.gef,后者的前缀一般为.cellbin.gef。(Stereo-seq技术分辨率可达纳米级别bin1的纳米孔半径约为250nm。通过bin_size参数,可以将一定范围内的纳米孔数据合并为一个bin unit(相当于一个分类单元)。例如,当bin_size设置为20时,bin unit的边长大约为10/14 μm(取决于纳米孔中心的距离:500nm或715nm)。在bin_size确定后,可自动生成StereoExpData用于下游分析。由于计算性能、生物学意义等问题,在分析过程中可能需要跳回“分bin”的步骤,优化参数后继续处理。)
此外stereopy还可以读取来自Anndata、Scanpy、Seurat的h5ad格式文件。
1.2 数据读取
# 导入包
import stereo as st
import warnings
warnings.filterwarnings('ignore')1.2.1 GEM文件读取
# gem文件读取
data_path = 'Demo_MouseBrain/SS200000135TL_D1.cellbin.gem'
data = st.io.read_gem(file_path=data_path,sep='\t',bin_type='cell_bins',is_sparse=True,)
data
# [2023-09-19 06:31:29][Stereo][3273491][MainThread][139900354834816][reader][84][INFO]: the martrix has 57133 cells, and 22406 genes.
# StereoExpData object with n_cells X n_genes = 57133 X 22406
# bin_type: cell_bins
# offset_x = 3206# offset_y = 6174
# cells: ['cell_name', 'cell_point']
# genes: ['gene_name']1.2.2 GEF文件读取
# suqre bin读取
data_path = 'Demo_MouseBrain/SS200000135TL_D1.tissue.gef'
data = st.io.read_gef(file_path=data_path,bin_type='bins',bin_size=100,is_sparse=True,)
data
# [2023-09-19 06:33:03][Stereo][3273491][MainThread][139900354834816][reader][1001][INFO]: read_gef begin ...
# [2023-09-19 06:33:23][Stereo][3273491][MainThread][139900354834816][reader][1088][INFO]: the matrix has 9124 cells, and 24302 genes.
# [2023-09-19 06:33:25][Stereo][3273491][MainThread][139900354834816][reader][1096][INFO]: read_gef end.
# StereoExpData object with n_cells X n_genes = 9124 X 24302
# bin_type: bins
# bin_size: 100
# offset_x = 0
# offset_y = 0
# cells: ['cell_name']
# genes: ['gene_name']# cell bin读取
data_path = './Demo_MouseBrain/SS200000135TL_D1.cellbin.gef'
data = st.io.read_gef(file_path=data_path,is_sparse=True,bin_type='cell_bins',)
# [2023-09-19 06:33:54][Stereo][3273491][MainThread][139900354834816][reader][1001][INFO]: read_gef begin ...
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1038][INFO]: the matrix has 57133 cells, and 24670 genes.
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1161][INFO]: This is GEF file which contains cell bin infomation.
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1162][INFO]: bin_type: cell_bins
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1168][INFO]: Number of cells: 57133
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1171][INFO]: Number of gene: 24670
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1174][INFO]: Resolution: 500
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1177][INFO]: offsetX: 0
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1180][INFO]: offsetY: 0
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1183][INFO]: Average number of genes: 223.460693359375
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1186][INFO]: Maximum number of genes: 1046
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1189][INFO]: Average expression: 399.17034912109375
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1192][INFO]: Maximum expression: 2337
# [2023-09-19 06:33:58][Stereo][3273491][MainThread][139900354834816][reader][1096][INFO]: read_gef end.1.2.3 Stereopy来源的h5ad文件读取
data_path = './Demo_MouseBrain/StereoExpData_stereopy_mykey.h5ad'
data = st.io.read_stereo_h5ad(file_path=data_path,use_raw=True,use_result=True,)
data
# StereoExpData object with n_cells X n_genes = 35998 X 3000
# bin_type: bins
# bin_size: 50
# offset_x = None
# offset_y = None
# cells: ['cell_name', 'total_counts', 'pct_counts_mt', 'n_genes_by_counts', 'leiden', 'louvain']
# genes: ['gene_name', 'n_cells', 'n_counts']
# key_record: {'cluster': ['leiden', 'louvain'], 'gene_exp_cluster': ['gene_exp_leiden', 'gene_exp_louvain']}1.2.4 Anndata来源的h5ad文件读取
ann_h5ad = './Demo_MouseBrain/anndata_output.h5ad'
data = st.io.read_ann_h5ad(file_path=ann_h5ad,spatial_key=None,)
data
# StereoExpData object with n_cells X n_genes = 35998 X 24302
# bin_type: bins
# bin_size: 50
# offset_x = None
# offset_y = None
# cells: ['cell_name']
# genes: ['gene_name']# Anndata来源的h5ad文件也可用于scanpy的分析
import scanpy as sc
# 直接利用scanpy读取:
data = sc.read_h5ad('Demo_MouseBrain/anndata_output.h5ad')
data
# AnnData object with n_obs × n_vars = 35998 × 24302
# obs: 'orig.ident', 'x', 'y'
# uns: 'bin_size', 'bin_type', 'raw_cellname', 'raw_counts', 'raw_genename', 'resolution', 'sn'
# obsm: 'spatial'1.3 文件输出
虽然输出的文件均为h5ad文件,但不同格式输出的h5ad文件具有差异,需要区别对待,输出对应软件需要的版本。
1.3.1 用于stereopy读取的h5ad输出¶
# 读取GEF文件data_path = './Demo_MouseBrain/SS200000135TL_D1.tissue.gef'data = st.io.read_gef(file_path=data_path, bin_size=50)# 预处理data.tl.cal_qc()data.tl.raw_checkpoint()data.tl.sctransform(res_key='sctransform', inplace=True)# 聚类data.tl.pca(use_highly_genes=False, n_pcs=30, res_key='pca')data.tl.neighbors(pca_res_key='pca', n_pcs=30, res_key='neighbors')data.tl.umap(pca_res_key='pca', neighbors_res_key='neighbors', res_key='umap')data.tl.leiden(neighbors_res_key='neighbors', res_key='leiden')data.tl.louvain(neighbors_res_key='neighbors', res_key='louvain')# data.tl.key_record为运行函数时自动生成的字典,收录的为res_key参数返回的内容,我们在后面正式的数据分析教程中会介绍print(data.tl.key_record)# [2023-09-19 08:09:11][Stereo][3273491][MainThread][139900354834816][st_pipeline][40][INFO]: umap end, consume time 24.3978s.# [2023-09-19 08:09:11][Stereo][3273491][MainThread][139900354834816][st_pipeline][37][INFO]: start to run leiden...# [2023-09-19 08:09:20][Stereo][3273491][MainThread][139900354834816][st_pipeline][40][INFO]: leiden end, consume time 9.2709s.# [2023-09-19 08:09:21][Stereo][3273491][MainThread][139900354834816][st_pipeline][37][INFO]: start to run louvain...# [2023-09-19 08:09:21][Stereo][3273491][MainThread][139900354834816][_louvain][100][INFO]: using the "louvain" package of Traag (2017)# [2023-09-19 08:09:35][Stereo][3273491][MainThread][139900354834816][st_pipeline][40][INFO]: louvain end, consume time 14.8895s.# {'hvg': [], 'pca': ['pca'], 'neighbors': ['neighbors'], 'umap': ['umap'], 'cluster': ['leiden', 'louvain'], 'marker_genes': [], 'sct': ['sctransform'], 'gene_exp_cluster': ['gene_exp_leiden', 'gene_exp_louvain']}
# 将StereoExpData对象写为h5ad, 如果key_record = None, 则会自动采用data.tl.key_recordst.io.write_h5ad(data,use_raw=True,use_result=True,key_record=None,output='./Demo_MouseBrain/StereoExpData_stereopy.h5ad')# 原gef大约600MB,输出后的h5ad约为3GB
# 当然你也可以建立一个字典保存指定的key_recordoutkey_record = {'cluster':['leiden','louvain'],}st.io.write_h5ad(data,use_raw=True,use_result=True,key_record=outkey_record,output='./Demo_MouseBrain/StereoExpData_stereopy_mykey.h5ad',)# 指定key_record后的输出文件大小为1.1GB,需要节省磁盘空间的同学可以尝试这种方法
1.3.2 用于Anndata读取的h5ad输出
# 读取gef文件:data_path = './Demo_MouseBrain/SS200000135TL_D1.tissue.gef'data = st.io.read_gef(file_path=data_path, bin_size=50)data.tl.raw_checkpoint()# 转换输出adata = st.io.stereo_to_anndata(data,flavor='seurat',output='Demo_MouseBrain/anndata_output.h5ad')# [2023-09-19 07:07:41][Stereo][3273491][MainThread][139900354834816][reader][1001][INFO]: read_gef begin ...# [2023-09-19 07:08:00][Stereo][3273491][MainThread][139900354834816][reader][1088][INFO]: the matrix has 35998 cells, and 24302 genes.# [2023-09-19 07:08:02][Stereo][3273491][MainThread][139900354834816][reader][1096][INFO]: read_gef end.# [2023-09-19 07:08:03][Stereo][3273491][MainThread][139900354834816][reader][756][INFO]: Adding sample in adata.obs['orig.ident'].# [2023-09-19 07:08:03][Stereo][3273491][MainThread][139900354834816][reader][759][INFO]: Adding data.position as adata.obsm['spatial'] .# [2023-09-19 07:08:03][Stereo][3273491][MainThread][139900354834816][reader][764][INFO]: Adding data.position as adata.obs['x'] and adata.obs['y'] .# [2023-09-19 07:08:03][Stereo][3273491][MainThread][139900354834816][reader][853][INFO]: Adding data.tl.raw.exp_matrix as adata.uns['raw_counts'] .# [2023-09-19 07:08:03][Stereo][3273491][MainThread][139900354834816][reader][884][INFO]: Rename QC info.# [2023-09-19 07:08:03][Stereo][3273491][MainThread][139900354834816][reader][900][INFO]: Finished conversion to anndata.# [2023-09-19 07:08:06][Stereo][3273491][MainThread][139900354834816][reader][904][INFO]: Finished output to Demo_MouseBrain/anndata_output.h5ad
1.3.3 用于Seurat读取的h5ad输出
Seurat对象可谓是单细胞分析界的硬通货,一定要学会转化。
# 读取数据data_path = './Demo_MouseBrain/SS200000135TL_D1.tissue.gef'data = st.io.read_gef(file_path=data_path, bin_size=50)# 预处理一下data.tl.cal_qc()data.tl.raw_checkpoint()# 存为Seurat友好型的h5adadata = st.io.stereo_to_anndata(data,flavor='seurat',output='Demo_MouseBrain/seurat_out.h5ad')# [2023-09-19 08:32:39][Stereo][3273491][MainThread][139900354834816][reader][1001][INFO]: read_gef begin ...# [2023-09-19 08:32:58][Stereo][3273491][MainThread][139900354834816][reader][1088][INFO]: the matrix has 35998 cells, and 24302 genes.# [2023-09-19 08:33:00][Stereo][3273491][MainThread][139900354834816][reader][1096][INFO]: read_gef end.# [2023-09-19 08:33:00][Stereo][3273491][MainThread][139900354834816][st_pipeline][37][INFO]: start to run cal_qc...# [2023-09-19 08:33:00][Stereo][3273491][MainThread][139900354834816][st_pipeline][40][INFO]: cal_qc end, consume time 0.7293s.# [2023-09-19 08:33:01][Stereo][3273491][MainThread][139900354834816][reader][756][INFO]: Adding sample in adata.obs['orig.ident'].# [2023-09-19 08:33:01][Stereo][3273491][MainThread][139900354834816][reader][759][INFO]: Adding data.position as adata.obsm['spatial'] .# [2023-09-19 08:33:01][Stereo][3273491][MainThread][139900354834816][reader][764][INFO]: Adding data.position as adata.obs['x'] and adata.obs['y'] .# [2023-09-19 08:33:01][Stereo][3273491][MainThread][139900354834816][reader][853][INFO]: Adding data.tl.raw.exp_matrix as adata.uns['raw_counts'] .# [2023-09-19 08:33:01][Stereo][3273491][MainThread][139900354834816][reader][884][INFO]: Rename QC info.# [2023-09-19 08:33:01][Stereo][3273491][MainThread][139900354834816][reader][900][INFO]: Finished conversion to anndata.# [2023-09-19 08:33:04][Stereo][3273491][MainThread][139900354834816][reader][904][INFO]: Finished output to Demo_MouseBrain/seurat_out.h5ad
后续h5ad在Seurat中的读取可以参考:单细胞对象(数据格式)转换大全|2. h5ad转Seuratobj。
当然,如果你更熟悉rds文件,也可以在shell中做如下转换:
!/usr/bin/Rscript ~/stereopy/docs/source/_static/annh5ad2rds.R --infile 'Demo_MouseBrain/seurat_out.h5ad' --outfile 'Demo_MouseBrain/seurat_out.rds'1.3.4 GEF输出
创建新的GEF用于输出:
# 数据读取data_path = './Demo_MouseBrain/SS200000135TL_D1.tissue.gef'data = st.io.read_gef(file_path=data_path, bin_size=50)# 选择对应基因取子集data.tl.filter_genes(gene_list=['H2al2a','Gm6135'], inplace=True)# 仅保存过滤后的结果st.io.write_mid_gef(data=data,output='./Demo_MouseBrain/my_filter.filtered.gef')# [2023-09-19 08:18:02][Stereo][3273491][MainThread][139900354834816][reader][1001][INFO]: read_gef begin ...# [2023-09-19 08:18:22][Stereo][3273491][MainThread][139900354834816][reader][1088][INFO]: the matrix has 35998 cells, and 24302 genes.# [2023-09-19 08:18:24][Stereo][3273491][MainThread][139900354834816][reader][1096][INFO]: read_gef end.# [2023-09-19 08:18:24][Stereo][3273491][MainThread][139900354834816][st_pipeline][37][INFO]: start to run filter_genes...# [2023-09-19 08:18:24][Stereo][3273491][MainThread][139900354834816][st_pipeline][40][INFO]: filter_genes end, consume time 0.5481s.# [2023-09-19 08:18:24][Stereo][3273491][MainThread][139900354834816][writer][287][INFO]: The output standard gef file only contains one expression matrix with mid count.Please make sure the expression matrix of StereoExpData object is mid count without normaliztion.
# 读取GEF文件data_path = './Demo_MouseBrain/SS200000135TL_D1.tissue.gef'data = st.io.read_gef(file_path=data_path, bin_size=50)# 预处理data.tl.cal_qc()data.tl.raw_checkpoint()data.tl.sctransform(res_key='sctransform', inplace=True)# 分群data.tl.pca(use_highly_genes=False, n_pcs=30, res_key='pca')data.tl.neighbors(pca_res_key='pca', n_pcs=30, res_key='neighbors')data.tl.umap(pca_res_key='pca', neighbors_res_key='neighbors', res_key='umap')data.tl.leiden(neighbors_res_key='neighbors', res_key='leiden')# 更新本地已存在的gefexist_path = './Demo_MouseBrain/my_filter.filtered.gef'st.io.update_gef(data=data,gef_file=exist_path,cluster_res_key='leiden',)
保存于已存在的GEF文件:
1.4 以AnnData格式操作
AnnData对象在Python亦是单细胞/空转分析的硬通货,Stereo中对象与其的相互转换与交互值得我们单独探究一番:
1.4.1 AnnData来源h5ad读取
import stereo as stdata = st.io.read_h5ad('Demo_MouseBrain/anndata_output.h5ad')# 此时读入的便是一个AnnData对象:data# AnnData object with n_obs × n_vars = 35998 × 24302# obs: 'orig.ident', 'x', 'y'# uns: 'bin_size', 'bin_type', 'raw_cellname', 'raw_counts', 'raw_genename', 'resolution', 'sn'# obsm: 'spatial'
# data._ann_data可以完全作为一个AnnData对象处理,而不是StereoExpData对象data._ann_data# AnnData object with n_obs × n_vars = 35998 × 24302# obs: 'orig.ident', 'x', 'y'# uns: 'bin_size', 'bin_type', 'raw_cellname', 'raw_counts', 'raw_genename', 'resolution', 'sn'# obsm: 'spatial'
1.4.2 各数据存储位置
# 表达矩阵:data._ann_data.X# <35998x24302 sparse matrix of type '<class 'numpy.float64'>'# with 41464124 stored elements in Compressed Sparse Row format>
# 空间坐标信息:data._ann_data.uns# OverloadedDict, wrapping:# {'bin_size': 50, 'bin_type': 'bins', 'raw_cellname': array(['24481313596250', '26199300516500', '26843545608050', ...,# '48533130461250', '38439957317800', '53687091208300'], dtype=object), 'raw_counts': <35998x24302 sparse matrix of type '<class 'numpy.uint32'>'# with 41464124 stored elements in Compressed Sparse Row format>, 'raw_genename': array(['0610005C13Rik', '0610006L08Rik', '0610009B22Rik', ..., 'mt-Nd4l',# 'mt-Nd5', 'mt-Nd6'], dtype=object), 'resolution': 500, 'sn': batch sn# 0 -1 SS200000135TL_D1}# With overloaded keys:# ['neighbors'].
# 细胞及相关注释信息:data._ann_data.obs

# 基因及相关注释信息:data._ann_data.var

1.4.3 数据处理
AnnData可直接参与大部分用于StereoExpData的计算
# 例如以下这些:data.tl.cal_qc()data.tl.raw_checkpoint()data.tl.normalize_total(target_sum=1e4)data.tl.log1p()data.tl.highly_variable_genes(min_mean=0.0125, max_mean=3, min_disp=0.5, res_key='highly_variable_genes', n_top_genes=None)data.tl.pca(use_highly_genes=True, hvg_res_key='highly_variable_genes', n_pcs=20, res_key='pca_test', svd_solver='arpack')data.tl.neighbors(pca_res_key='pca_test', n_pcs=30, res_key='neighbors_test', n_jobs=8)data.tl.umap(pca_res_key='pca_test', neighbors_res_key='neighbors_test', res_key='umap_test', init_pos='spectral')data.tl.leiden(neighbors_res_key='neighbors_test', res_key='leiden_test')
# 计算得到的结果也会自动保存于data._ann_data中data._ann_data# AnnData object with n_obs × n_vars = 35998 × 24302# obs: 'orig.ident', 'x', 'y', 'total_counts', 'n_genes_by_counts', 'pct_counts_mt', 'leiden_test'# var: 'n_cells', 'n_counts', 'mean_umi', 'means', 'dispersions', 'dispersions_norm', 'highly_variable'# uns: 'bin_size', 'bin_type', 'raw_cellname', 'raw_counts', 'raw_genename', 'resolution', 'sn', 'highly_variable_genes', 'pca_test', 'neighbors_test', 'umap_test', 'leiden_test', 'gene_exp_leiden_test'# obsm: 'spatial', 'X_pca_test', 'X_umap_test'# obsp: 'neighbors_test_connectivities', 'neighbors_test_distances'
# 例如这里多出的细胞信息:data._ann_data.obs

# 可视化函数一样可以利用:data.plt.umap(res_key='umap_test', cluster_key='leiden_test')

1.4.4 数据输出
用于stereopy分析的AnnData对象也可以存为h5ad,
data._ann_data.write_h5ad('./test_result/SS200000135TL_D1.stereo.h5ad')更多空间转录组分析技巧可见:空间转录组学习手册合辑
相关文章:

日常吐槽。
一、写在前面 stereopy日常出bug(github issue里得有一半的问题是我提的,当然也有可能是因为我菜),stereopy自己生成的anndata自己不能计算空间共现关系,还是靠squidpy才能计算。另外还要一些函数一开并行计算就报错,这里留一些s…...

2025最新版Node.js下载安装~保姆级教程
1. node中文官网地址:http://nodejs.cn/download/ 2.打开node官网下载压缩包: 根据操作系统不同选择不同版本(win7系统建议安装v12.x) 我这里选择最新版win 64位 3.安装node ①点击对话框中的“Next”,勾选同意后点…...
机器学习:学习记录(二)
1. 机器学习中的常用函数 logistic函数(sigmoid函数):非线性激活函数,将R区间映射到(0,1)区间 ReLU函数:非线性激活函数,简单可以写作max(0,x),在0处不可导,但是可以人为定义其导数…...

迁移学习 Transfer Learning
迁移学习(Transfer Learning)是什么? 迁移学习是一种机器学习方法,它的核心思想是利用已有模型的知识来帮助新的任务或数据集进行学习,从而减少训练数据的需求、加快训练速度,并提升模型性能。 …...

实现:多活的基础中间件
APIRouter : 路由分发服务 API Router 是一个 HTTP 反向代理和负载均衡器,部署在公有云中作为 HTTP API 流量的入口,它能识别 出流量的归属 shard ,并根据 shard 将流量转发到对应的 ezone 。 API Router 支持多种路由键&am…...

Mybatis源码01 - 总体框架设计
Mybatis总体框架设计 文章目录 Mybatis总体框架设计一:MyBatis架构概览1:接口层1.1:使用传统的MyBatis提供的API1.2:使用Mapper接口 2:数据处理层【核心】2.1:参数映射和动态SQL语句生成2.2:SQL…...

在大型语言模型(LLM)框架内Transformer架构与混合专家(MoE)策略的概念整合
文章目录 传统的神经网络框架存在的问题一. Transformer架构综述1.1 transformer的输入1.1.1 词向量1.1.2 位置编码(Positional Encoding)1.1.3 编码器与解码器结构1.1.4 多头自注意力机制 二.Transformer分步详解2.1 传统词向量存在的问题2.2 详解编解码…...

Selenium WebDriver自动化测试(扩展篇)--Jenkins持续集成
文章目录 一、引言二、Jenkins简介三、安装部署Jenkins安装部署 四、集成Git与Maven安装必要的插件配置Git配置Maven 五、创建Job创建自由风格的项目配置源码管理配置构建触发器配置构建环境配置构建步骤配置Post-build Actions 六、触发构建示例:GitHub Webhook触发…...

Wiki文档转换为Word技术
一、技术背景与目标 Wiki系统导出的文档通常以HTML格式存在,且内容分散在多个文件中,每个页面对应一个HTML文件。然而,Microsoft Word(Word)在处理HTML文件时,仅支持单个HTML文件的导入。因此,为了将Wiki导出的内容转换为Word可识别的格式,必须将分散的HTML文件整合为一…...

1.【线性代数】——方程组的几何解释
一 方程组的几何解释 概述举例举例一1. matrix2.row picture3.column picture 概述 三种表示方法 matrixrow picturecolumn picture 举例 举例一 { 2 x − y 0 − x 2 y 3 \begin{cases} 2x - y 0 \\ -x 2y 3 \end{cases} {2x−y0−x2y3 1. matrix [ 2 − 1 − 1 …...

力扣1448. 统计二叉树中好节点的数目
Problem: 1448. 统计二叉树中好节点的数目 文章目录 题目描述思路复杂度Code 题目描述 思路 对二叉树进行先序遍历,边遍历边对比并更新当前路径上的最大值pathMax,若当pathMax小于等于当前节点值,则好节点的数目加一 复杂度 时间复杂度: O (…...
——C#注释和命名法详解)
【C#零基础从入门到精通】(二)——C#注释和命名法详解
【C#零基础从入门到精通】(二)——C#注释和命名法详解 C# 中的注释 定义 在 C# 里,注释是一种特殊的代码文本,它不会被编译器执行,主要用于对代码进行解释、说明,帮助开发者更好地理解代码的功能、用途、实现思路以及注意事项等,提升代码的可读性和可维护性。 注释类型…...

SQLServer的创建,表创建,主键,约束,模糊查询
设置 注意: 设置完成之后 重新启动 创建数据库 注意: 这个目标路径必须要有该文件名的文件夹 -- 指向 master 数据库,告诉它我们要创建一个新的数据库操作了 use master go-- 创建数据库 create database StudentManageDB on primary (-- 以下四个组成部分缺一不可…...
架构设计指南)
DeepSeek深度思考:客户端(Android/iOS)架构设计指南
目标读者:中高级开发者、架构师 适用场景:大型复杂应用开发、跨团队协作、长期维护迭代 一、架构设计核心原则 1.模块化(Modularization) 横向拆分:按功能边界划分(如登录、支付、消息模块)纵向…...

亚远景-精通ASPICE:专业咨询助力汽车软件开发高效合规
在竞争日益激烈的汽车行业,软件开发已成为决定成败的关键因素。ASPICE(汽车软件过程改进和能力确定) 作为行业公认的软件开发框架,为汽车制造商和供应商提供了实现高效、合规开发的路线图。 然而,ASPICE 的实施并非易…...

OpenCV 相机标定流程指南
OpenCV 相机标定流程指南 前置准备标定流程结果输出与验证建议源代码 OpenCV 相机标定流程指南 https://docs.opencv.org/4.x/dc/dbb/tutorial_py_calibration.html https://learnopencv.com/camera-calibration-using-opencv/ 前置准备 制作标定板:生成高精度棋…...

项目场景拷打
补偿事务解决超卖 通过补偿事务避免超卖问题,可以通过以下几种方式实现: 1. 使用数据库事务与锁机制 事务管理:将库存扣减和订单生成操作放在同一个数据库事务中,确保操作的原子性。如果事务中任何一个步骤失败,则整…...

Vue2生命周期面试题
在 Vue 2 中,this.$el 和 this.$data 都是 Vue 实例的属性,代表不同的内容。 1. this.$el this.$el 是 Vue 实例的根 DOM 元素,它指向 Vue 实例所控制的根节点元素。在 Vue 中,el 是在 Vue 实例创建时,指定的根元素&…...

【每日一题 | 2025】2.3 ~ 2.9
个人主页:GUIQU. 归属专栏:每日一题 文章目录 1. 【2.3】P8784 [蓝桥杯 2022 省 B] 积木画2. 【2.4】P8656 [蓝桥杯 2017 国 B] 对局匹配3. 【2.5】[ABC365D] AtCoder Janken 34. 【2.6】P8703 [蓝桥杯 2019 国 B] 最优包含5. 【2.7】P8624 [蓝桥杯 2015…...

使用OpenGL自己定义一个button,响应鼠标消息:掠过、点击、拖动
button需要有一个外观 外观 大小跟随窗口改变,采用纯色背景、纯色文字 文字 大小跟随窗口改变 button需要获得鼠标消息 掠过 鼠标掠过时 button 出现阴影,鼠标掠过后 button 阴影消失 点击 点击后进入相应事件 拖动 改变图标所在位置 需要在g…...
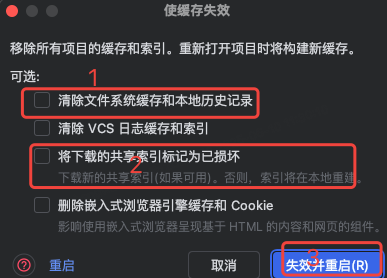
idea大量爆红问题解决
问题描述 在学习和工作中,idea是程序员不可缺少的一个工具,但是突然在有些时候就会出现大量爆红的问题,发现无法跳转,无论是关机重启或者是替换root都无法解决 就是如上所展示的问题,但是程序依然可以启动。 问题解决…...
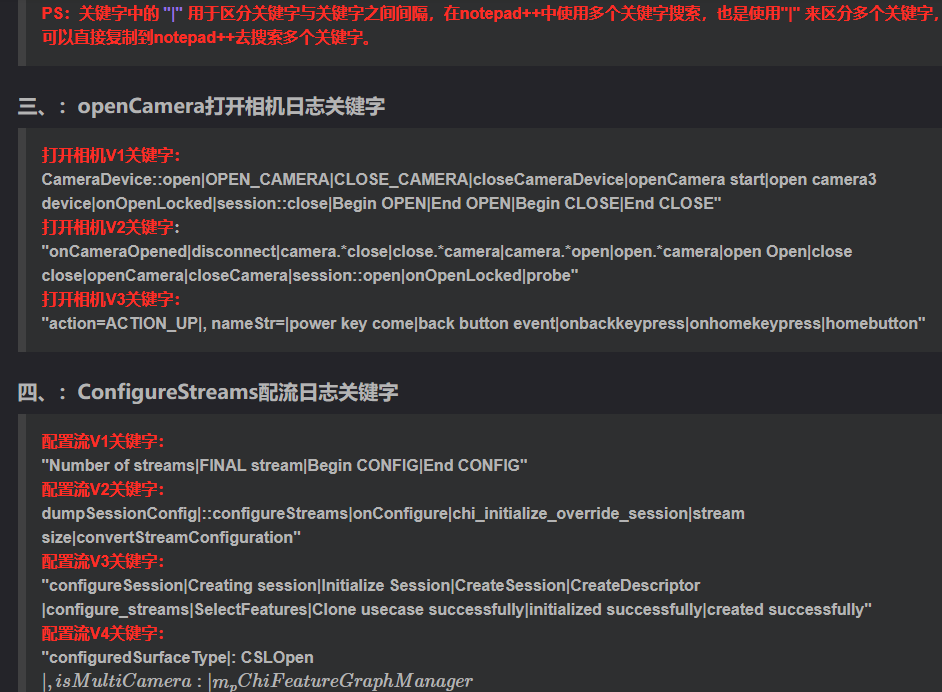
相机Camera日志实例分析之二:相机Camx【专业模式开启直方图拍照】单帧流程日志详解
【关注我,后续持续新增专题博文,谢谢!!!】 上一篇我们讲了: 这一篇我们开始讲: 目录 一、场景操作步骤 二、日志基础关键字分级如下 三、场景日志如下: 一、场景操作步骤 操作步…...

Swift 协议扩展精进之路:解决 CoreData 托管实体子类的类型不匹配问题(下)
概述 在 Swift 开发语言中,各位秃头小码农们可以充分利用语法本身所带来的便利去劈荆斩棘。我们还可以恣意利用泛型、协议关联类型和协议扩展来进一步简化和优化我们复杂的代码需求。 不过,在涉及到多个子类派生于基类进行多态模拟的场景下,…...

el-switch文字内置
el-switch文字内置 效果 vue <div style"color:#ffffff;font-size:14px;float:left;margin-bottom:5px;margin-right:5px;">自动加载</div> <el-switch v-model"value" active-color"#3E99FB" inactive-color"#DCDFE6"…...

MODBUS TCP转CANopen 技术赋能高效协同作业
在现代工业自动化领域,MODBUS TCP和CANopen两种通讯协议因其稳定性和高效性被广泛应用于各种设备和系统中。而随着科技的不断进步,这两种通讯协议也正在被逐步融合,形成了一种新型的通讯方式——开疆智能MODBUS TCP转CANopen网关KJ-TCPC-CANP…...

鱼香ros docker配置镜像报错:https://registry-1.docker.io/v2/
使用鱼香ros一件安装docker时的https://registry-1.docker.io/v2/问题 一键安装指令 wget http://fishros.com/install -O fishros && . fishros出现问题:docker pull 失败 网络不同,需要使用镜像源 按照如下步骤操作 sudo vi /etc/docker/dae…...
网络编程(UDP编程)
思维导图 UDP基础编程(单播) 1.流程图 服务器:短信的接收方 创建套接字 (socket)-----------------------------------------》有手机指定网络信息-----------------------------------------------》有号码绑定套接字 (bind)--------------…...
:观察者模式)
JS设计模式(4):观察者模式
JS设计模式(4):观察者模式 一、引入 在开发中,我们经常会遇到这样的场景:一个对象的状态变化需要自动通知其他对象,比如: 电商平台中,商品库存变化时需要通知所有订阅该商品的用户;新闻网站中࿰…...
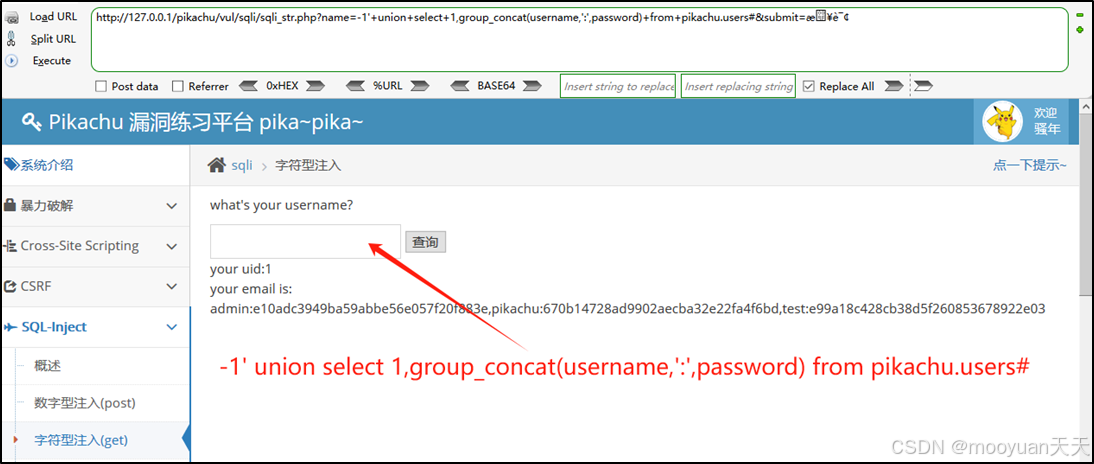
pikachu靶场通关笔记19 SQL注入02-字符型注入(GET)
目录 一、SQL注入 二、字符型SQL注入 三、字符型注入与数字型注入 四、源码分析 五、渗透实战 1、渗透准备 2、SQL注入探测 (1)输入单引号 (2)万能注入语句 3、获取回显列orderby 4、获取数据库名database 5、获取表名…...
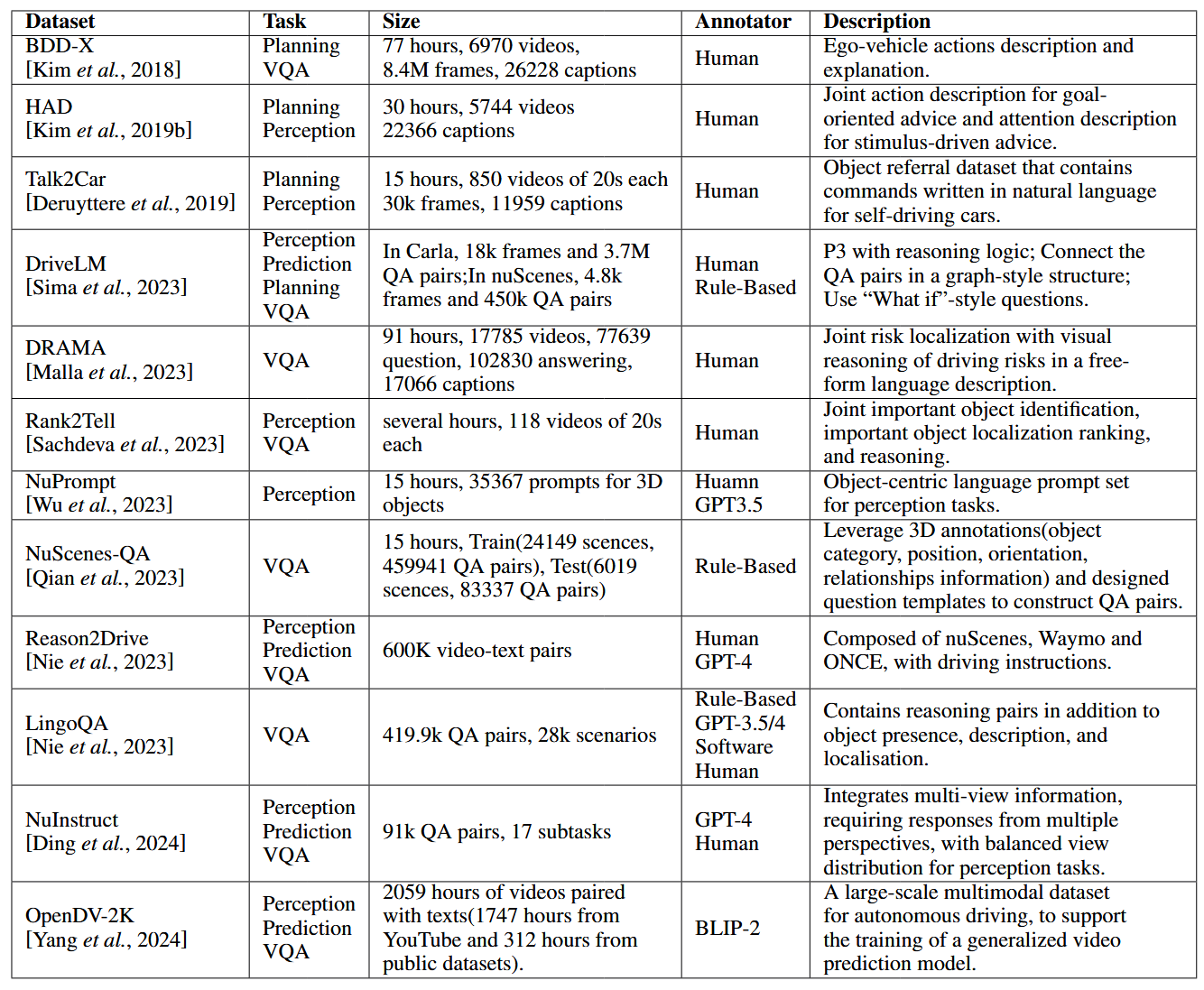
论文阅读:LLM4Drive: A Survey of Large Language Models for Autonomous Driving
地址:LLM4Drive: A Survey of Large Language Models for Autonomous Driving 摘要翻译 自动驾驶技术作为推动交通和城市出行变革的催化剂,正从基于规则的系统向数据驱动策略转变。传统的模块化系统受限于级联模块间的累积误差和缺乏灵活性的预设规则。…...
